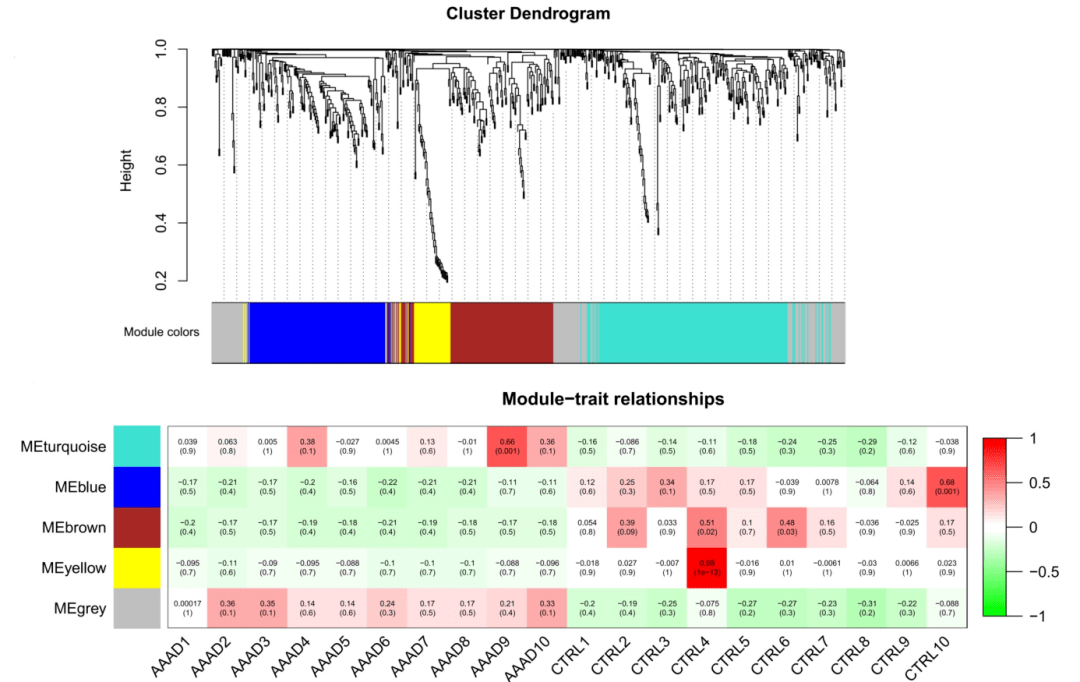
WGCNA分析,是加权基因共表达网络分析。在文献中,我们经常能够看到这样的WGCNA分析图。例如文献“Expression
profiles of circRNAs and the potential diagnostic value of serum
circMARK3 in human acute Stanford type A aortic dissection”中的Figure 4就是通过WGCNA分析得到的。
前两天,有位老师看到一个WGCNA脚本,希望我帮忙修改。原始脚本网址:https://github.com/erebboah/WGCNA/blob/master/run_WGCNA_ER_consensus.R
1 示例文件
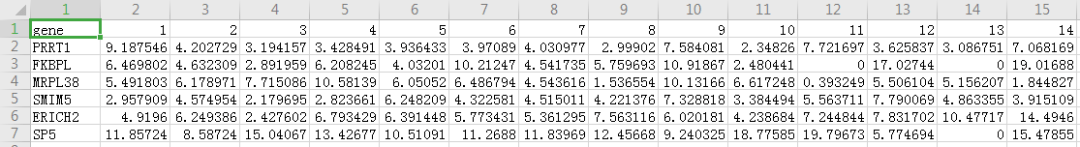
示例文件中共有2个文件,其中FPKM.csv文件为标准化后的表达量文件。
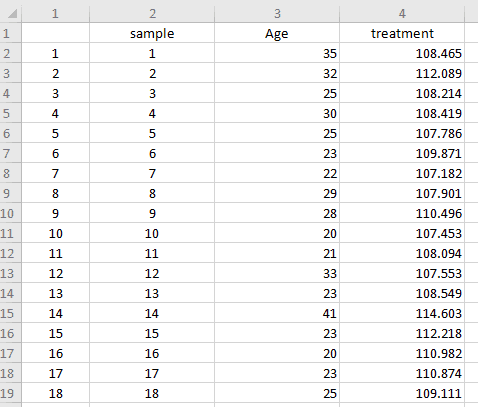
Clinic_data.xlsx文件为各个样品对应的临床数据文件。
2 修改脚本
由于原始脚本的项目中有不少输入文件缺失,也没有rda文件。我们只能通过WGCNA_tutorial_ER.pdf进行研究,并整理输入数据。
!!!**************************************************
library("openxlsx")
datExpr = read.csv("FPKM.csv",row.names = 1)
datExpr = t(datExpr)
multiExpr=list(list(data = datExpr))
exprSize = checkSets(multiExpr)
nSets = exprSize$nSets
nGenes = exprSize$nGenes
nSamples = exprSize$nSamples
targets = read.xlsx("Clinic_data.xlsx",rowNames = TRUE)
multiMeta = list(sample = list(data = targets))
setLabels = c("Samples")
gsg = goodSamplesGenesMS(multiExpr, verbose = 1)
gsg$allOK
save(multiExpr, multiMeta, nSets, nGenes, nSamples, setLabels, targets, datExpr, file = "Consensus_dataInput.rda")
!!!**************************************************
3 输出结果
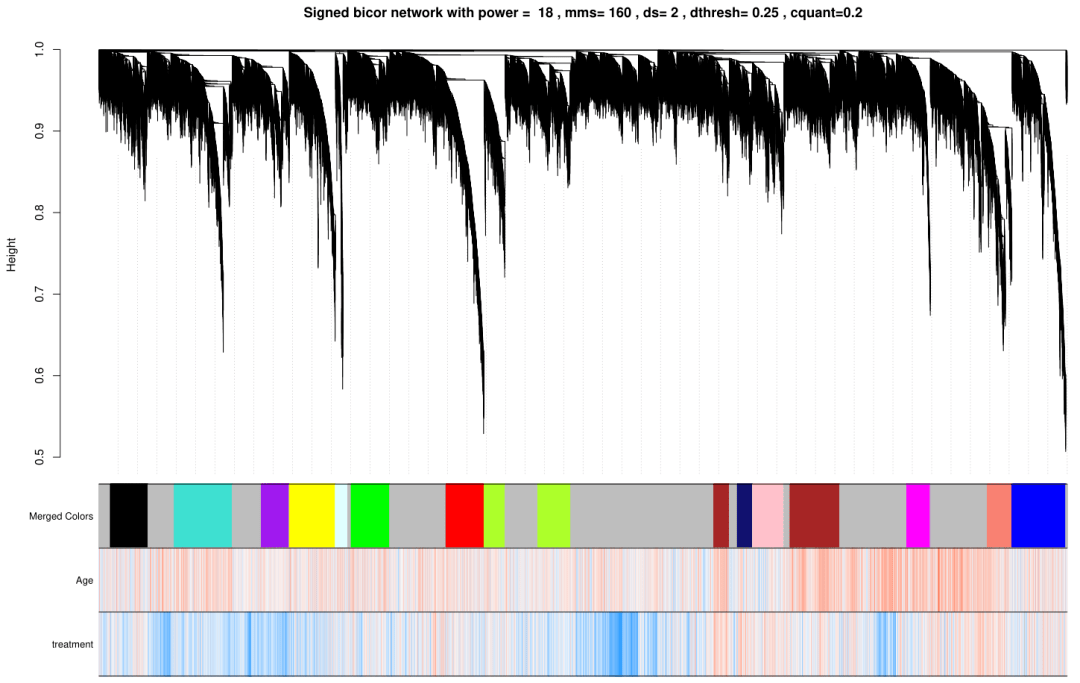
使用修改后的脚本进行WGCNA分析,选择合适的softpower,mms,ds和dthrsh等参数,输出WGCNA的分析结果。
4 更多的问题
老师还有其他的数据也需要用该脚本分析,希望使用相同的脚本进行分析,但是由于文件格式等问题经常报错。接下来就看看如何解决这些问题吧。
(1)运行“library("WGCNA")”时报错:there is no package called 'WGCNA'。
解决方法:使用R包前需要先安装R包,输入命令以下就行了。
!!!**************************************************
install.packages("WGCNA")
!!!**************************************************
(2)使用不同文件进行分析时,从文件中读取数据时出错。
解决方法:对于不同文件格式的文件需要修改部分参数。
!!!**************************************************
datExpr2 = read.csv("FPKM2.txt",row.names = 1,sep = "\t")
# read.xlsx函数只能用于读取Excel文件
targets2 = read.xlsx("Clinic_data2.xlsx",rowNames = TRUE)
!!!**************************************************
(3)运行pickSoftThreshold函数时超出线程,报错:OpenBLAS blas_thread_init: pthread_create failed for ...
解决方法:减少线程或者使用单线程。
!!!**************************************************
allowWGCNAThreads()
!!!**************************************************
(4)运行WGCNA脚本的时间过长。
解决方法:保存全部结果到rda文件中。
!!!**************************************************
save(list=ls(), file="data.rda")
!!!**************************************************
!
!
!
注:!!!*******之间为R脚本内容。
转自:纪伟讲测序