用ChIPseeker对ChIP-seq数据进行可视化,图表直观颜值高
2022-5-10 萌小白
ChIP-seq主要用来研究蛋白质和DNA的相互作用,ChIPseeker 可以用来对ChIP-seq数据进行注释与可视化,下面我们就来介绍一下如何用ChIPseeker对chip-seq数据进行可视化操作。
操作步骤
把所有sample_peaks文件放在工作路径下,格式为
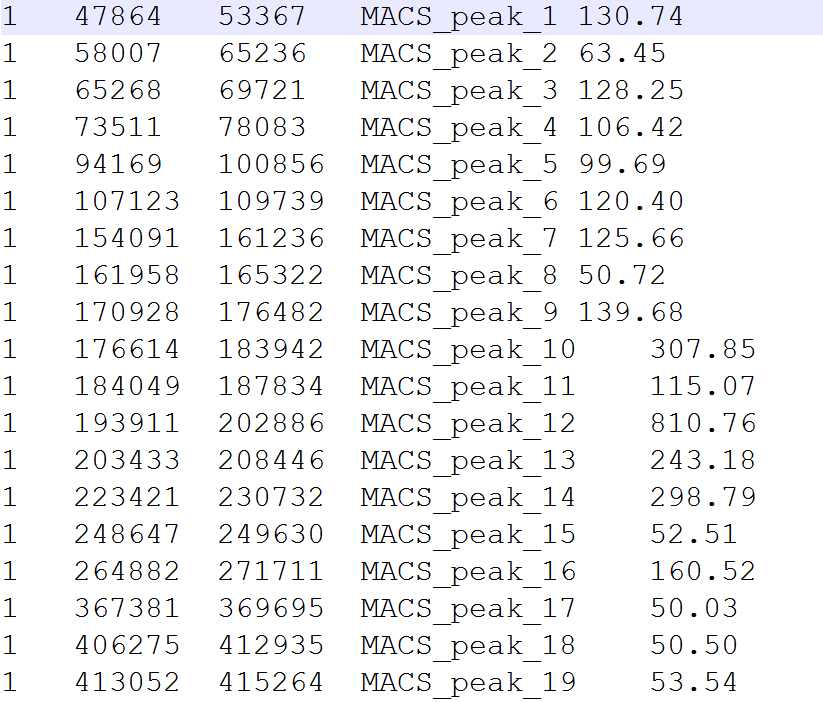
#安装程序
#source("http://bioconductor.org/biocLite.R") #biocLite("ChIPseeker") #biocLite("TxDb.Athaliana.BioMart.plantsmart28")
#开始分析
library("ChIPseeker") library(ggplot2)
library("TxDb.Athaliana.BioMart.plantsmart28") txdb <-
TxDb.Athaliana.BioMart.plantsmart28 get_files_data<-function () { dir
<- getwd() files <- list.files(dir) protein <- gsub(pattern =
"GSM\\d+_(\\w+_\\w+)_.*", replacement = "\\1", files) protein <-
sub("_Chip.+", "", protein) res <- paste(dir, files, sep = "/") res
<- as.list(res) names(res) <- protein return(res)}
files<-get_files_data()
#读入启动子信息
promoter <- getPromoters(TxDb=txdb,upstream=3000, downstream=3000)
#画覆盖图
covplot(files[[4]])

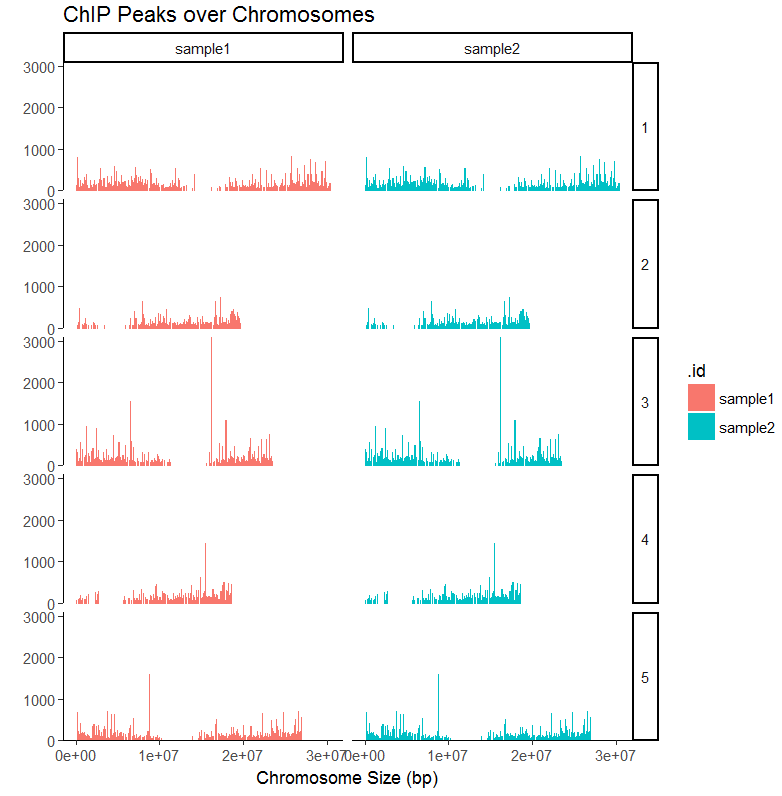
#画两个样品的覆盖图
peak1=GenomicRanges::GRangesList(sample1=readPeakFile(files[[1]]),sample2=readPeakFile(files[[2]]))
covplot(peak1, weightCol="V5") + facet_grid(chr ~ .id)
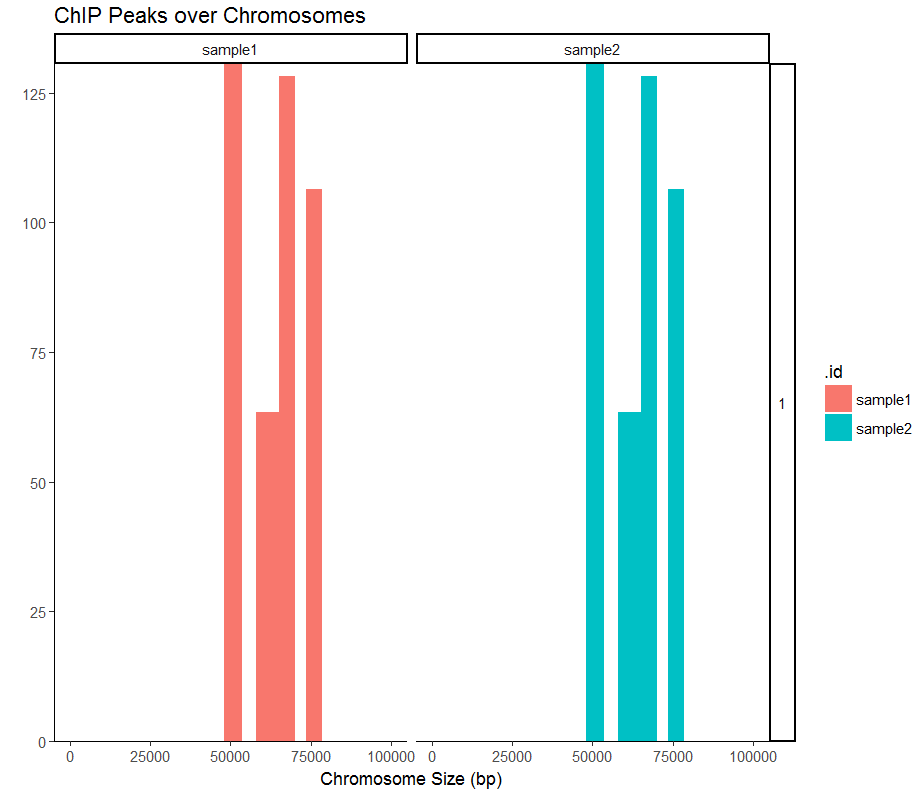
#放大一个区域
covplot(peak1, weightCol="V5", chrs=c("1", "2"), xlim=c(1, 100000)) + facet_grid(chr ~ .id)
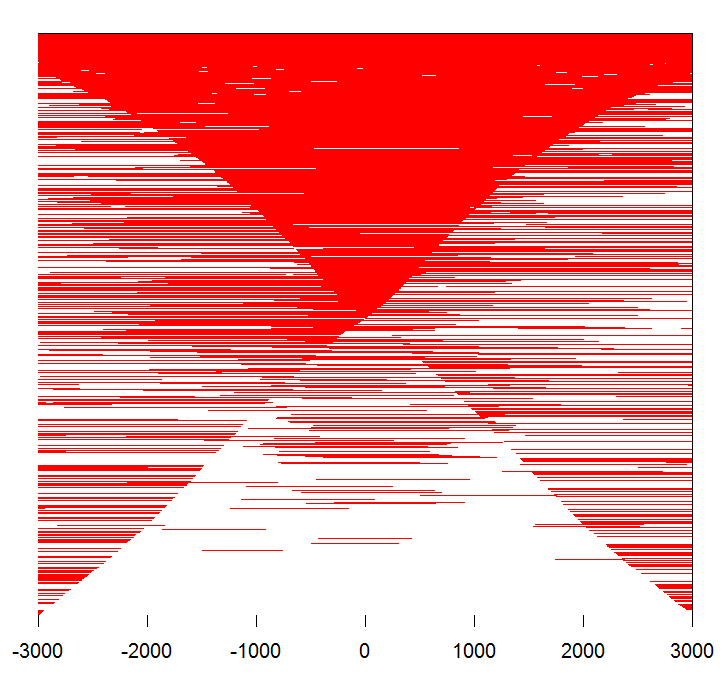
#画其中一个热图
peak <- readPeakFile(files[[4]]) tagMatrix <-
getTagMatrix(peak, windows=promoter) tagHeatmap(tagMatrix, xlim=c(-3000,
3000), color="red")
#画结合区域热图
peakHeatmap(files,weightCol="V5",TxDb=txdb,upstream=3000,downstream=3000,color=rainbow(length(files)))
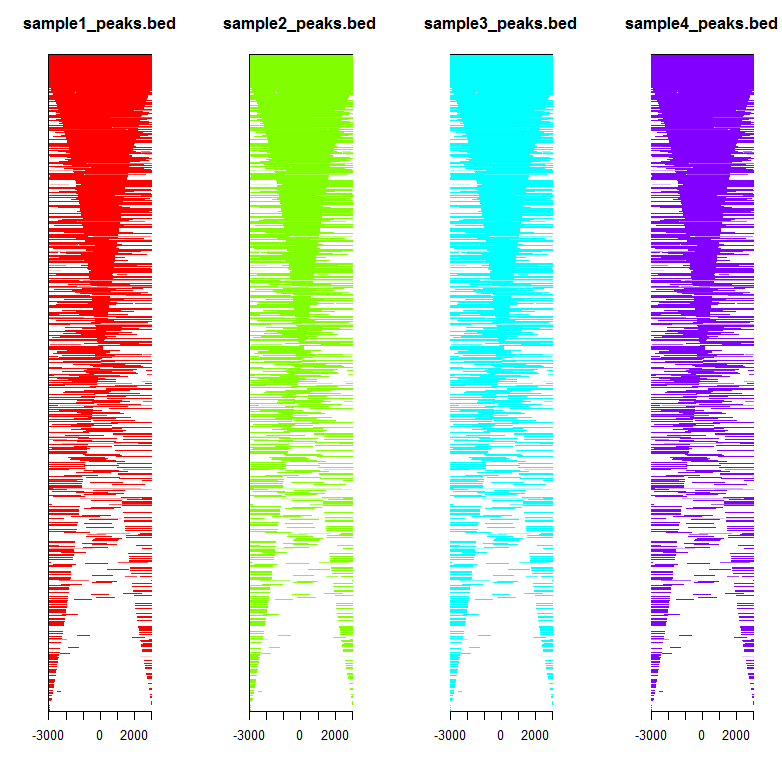
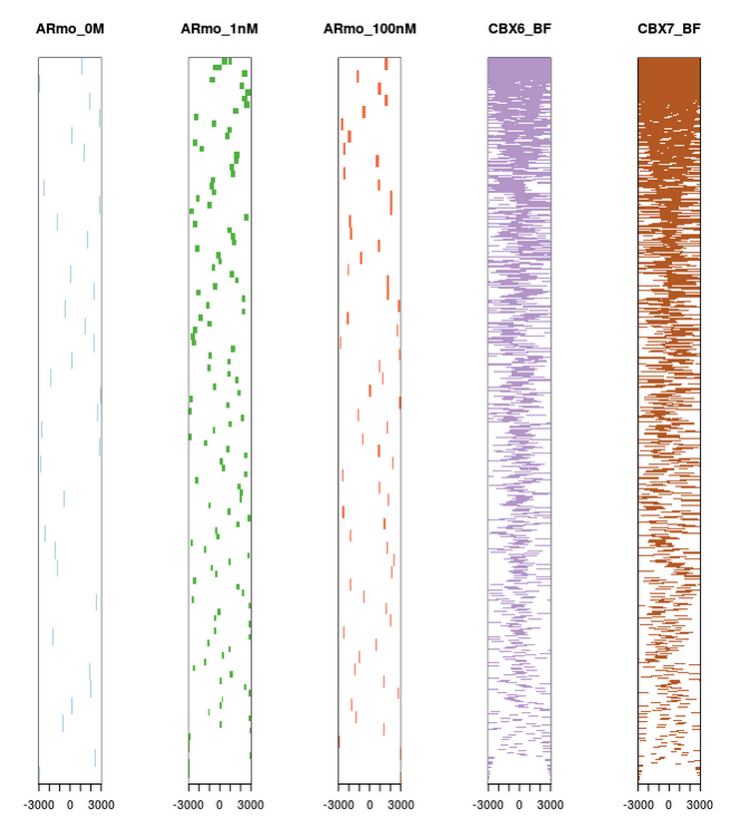
如图所示,前三个样品结合位点不在转录起始区域,后两个在。说明前三个样品不是调控转录的。
#结合强度图
plotAvgProf(tagMatrix, xlim=c(-3000, 3000),xlab="Genomic Region (5’->3’)",ylab = "Read Count Frequency")
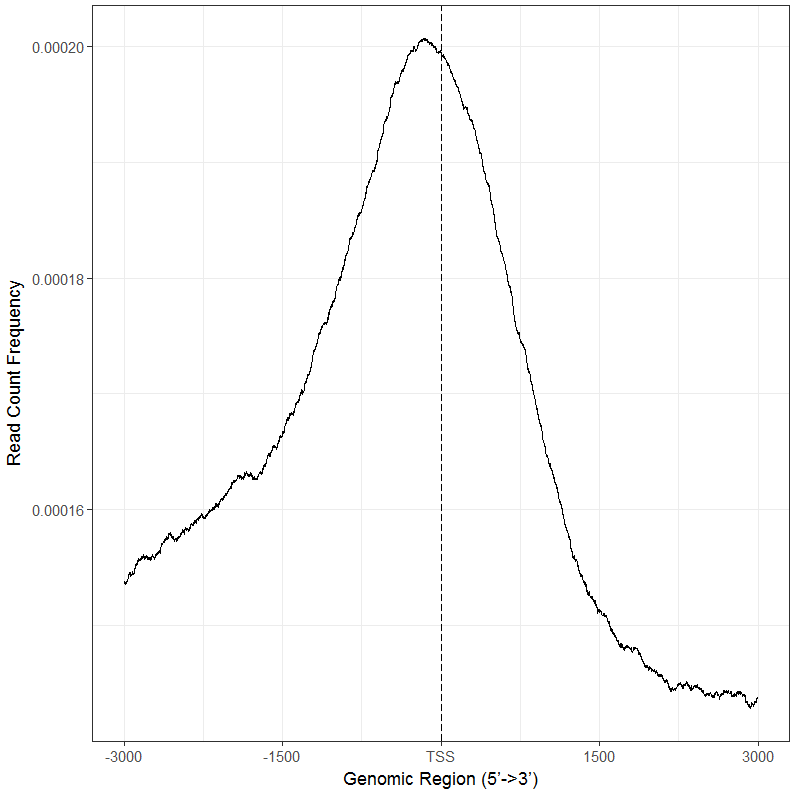
可以看到转录起始区域结合强度最高
#直接以文件做图,结果与上图一致
plotAvgProf2(files[[4]],
TxDb=txdb,upstream=3000, downstream=3000,xlab="Genomic Region
(5’->3’)",ylab = "Read Count Frequency")
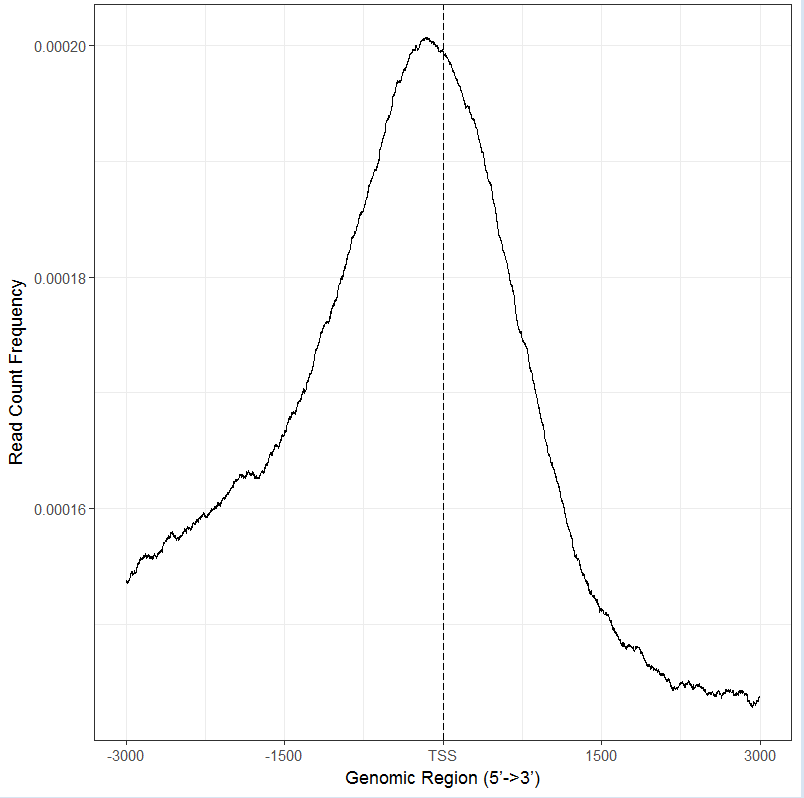
#加上bootstrap值
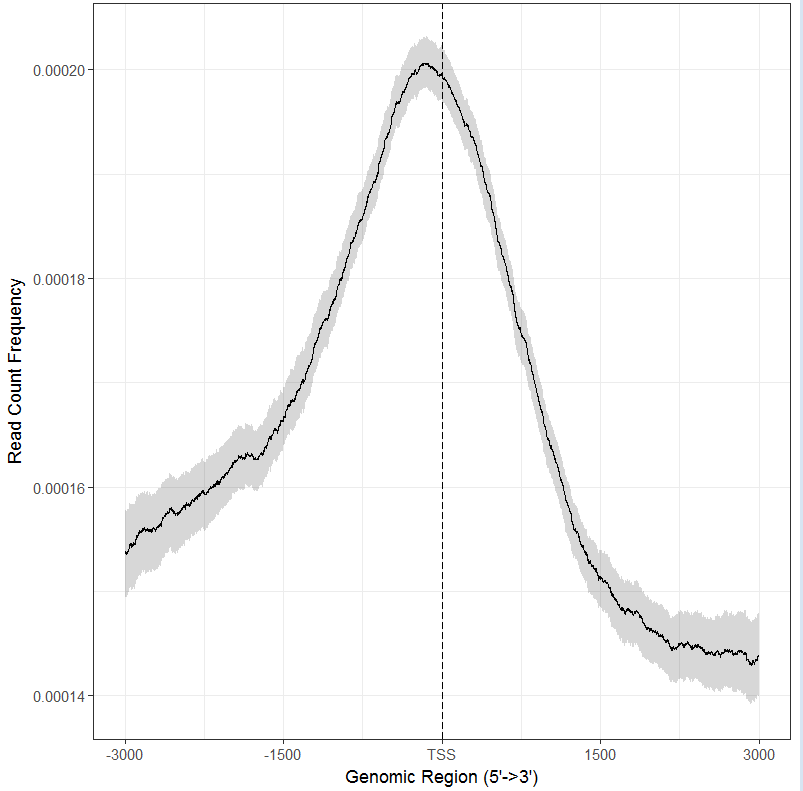
#所有数据一起做图
tagMatrixList <- lapply(files,getTagMatrix,windows=promoter) plotAvgProf(tagMatrixList, xlim=c(-3000, 3000))
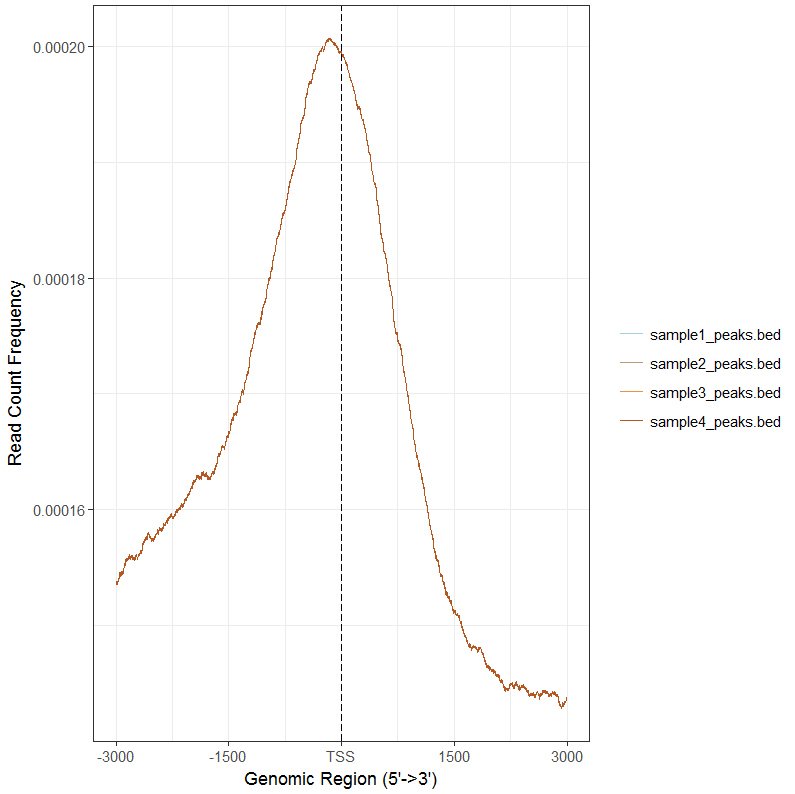
#分面,同时计算置信区间
plotAvgProf(tagMatrixList, xlim=c(-3000, 3000),conf=0.95,resample=500, facet="row")
#画饼状图
peakAnno <- annotatePeak(files[[4]], tssRegion=c(-3000, 3000),TxDb=txdb, annoDb="org.At.tair.db") plotAnnoPie(peakAnno)
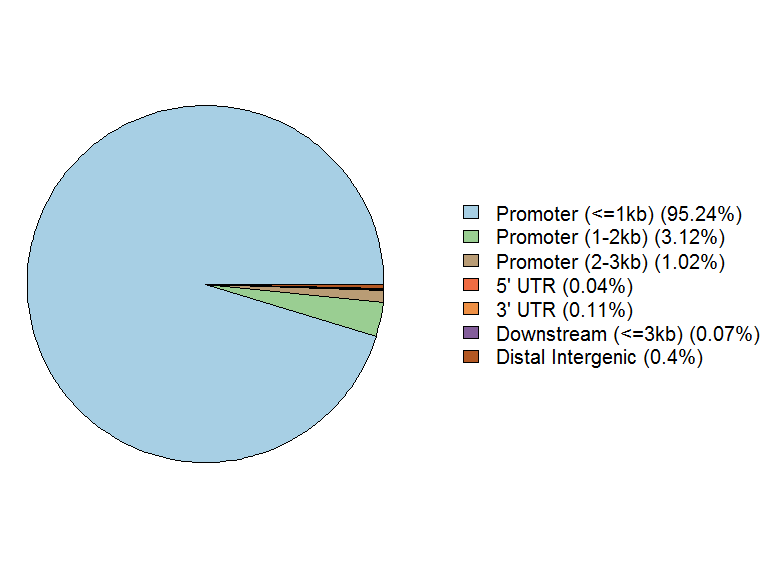
#画柱状图
plotAnnoBar(peakAnno)
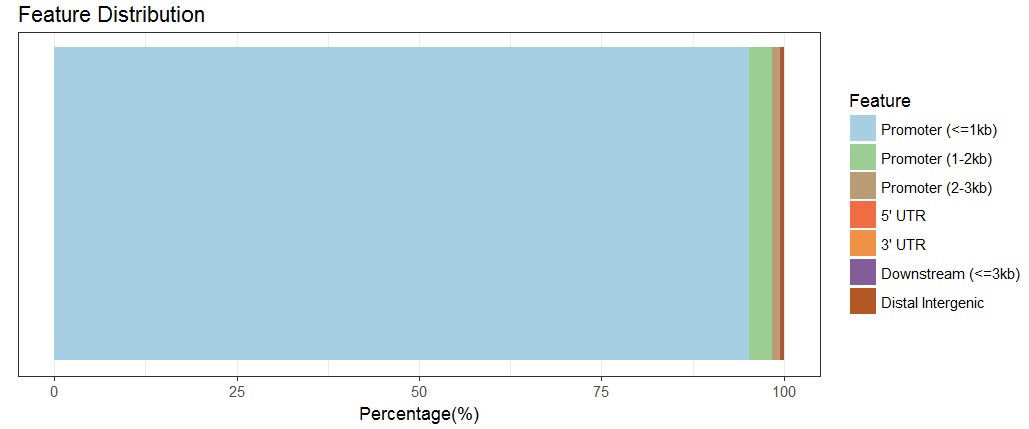
#画维恩饼图
vennpie(peakAnno)
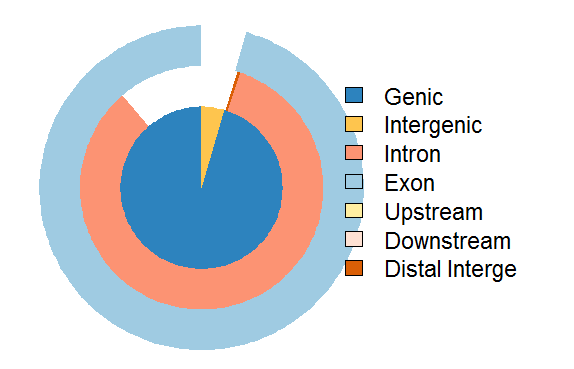
#画 upset图
upsetplot(peakAnno)
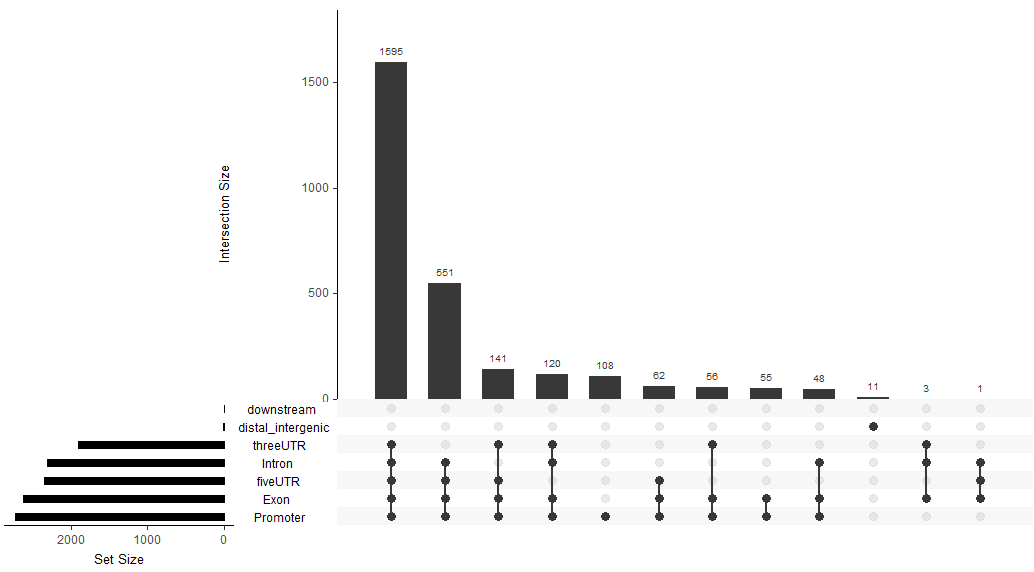
#结合起来
upsetplot(peakAnno, vennpie=TRUE)
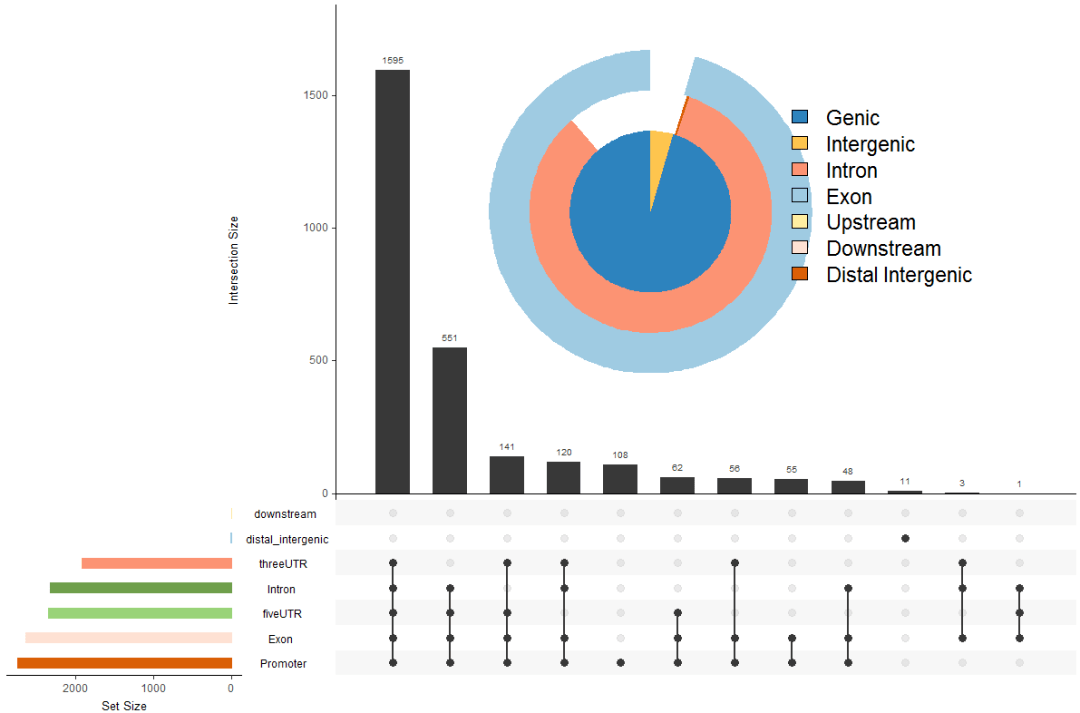
#离最近基因的距离
plotDistToTSS(peakAnno,title="Distribution of transcription factor-binding loci\nrelative to TSS")
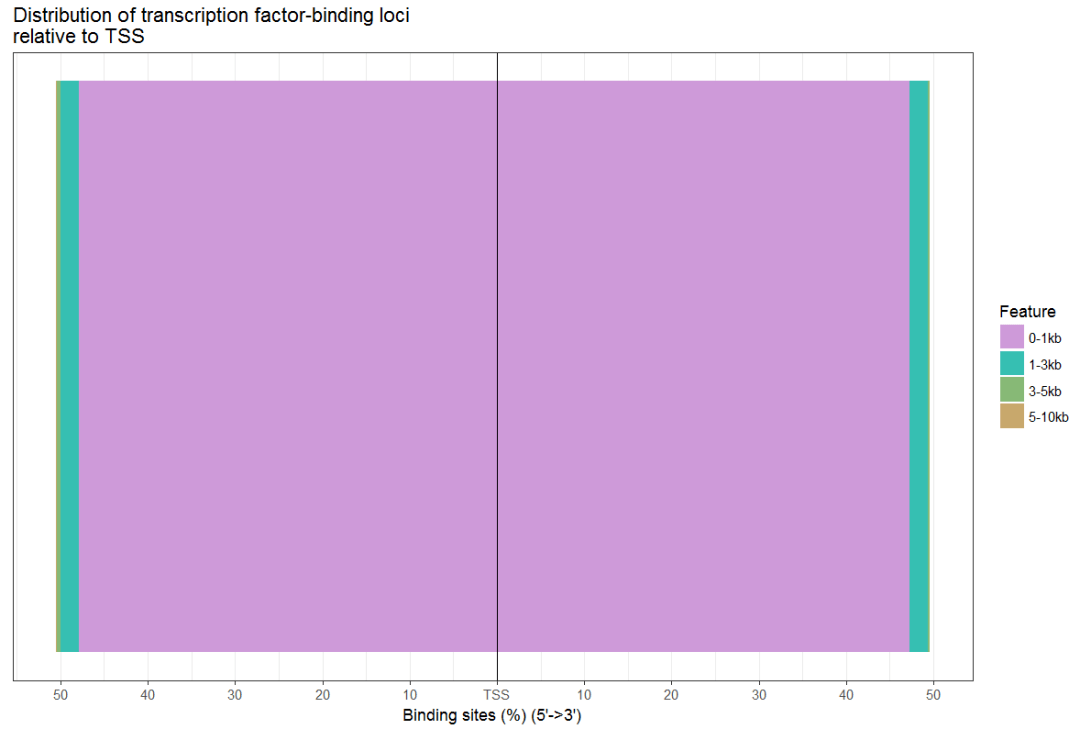
#同时画多个柱状图
peakAnnoList <- lapply(files, annotatePeak, TxDb=txdb,tssRegion=c(-3000, 3000), verbose=FALSE) plotAnnoBar(peakAnnoList)
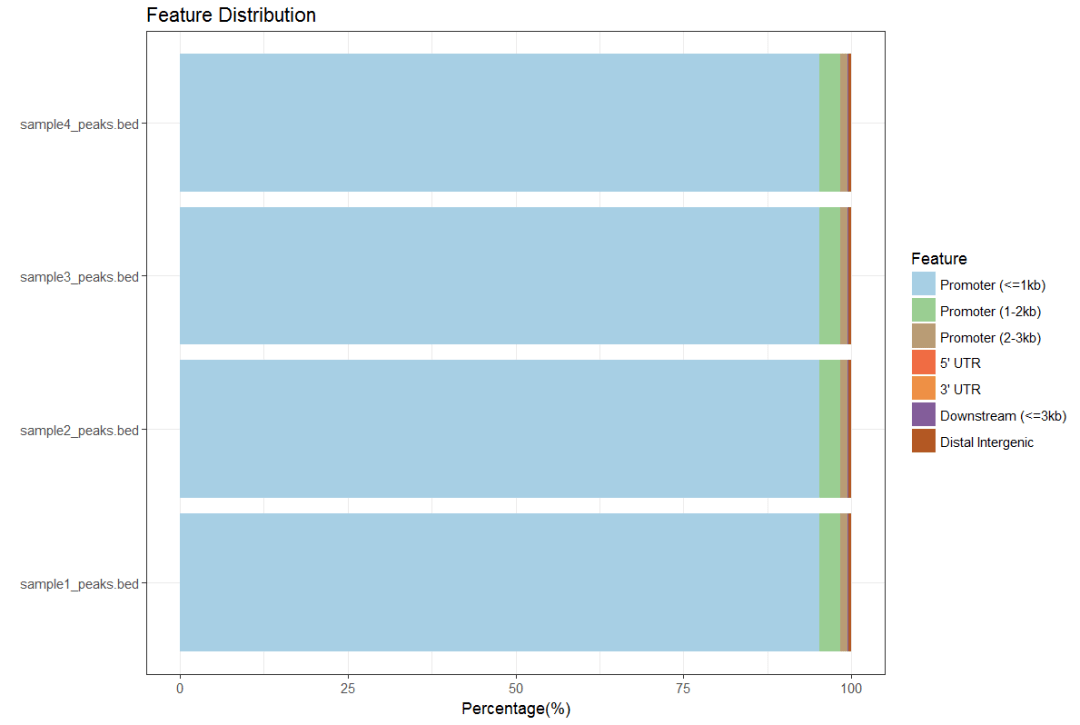
#同时画多个距离图
plotDistToTSS(peakAnnoList)
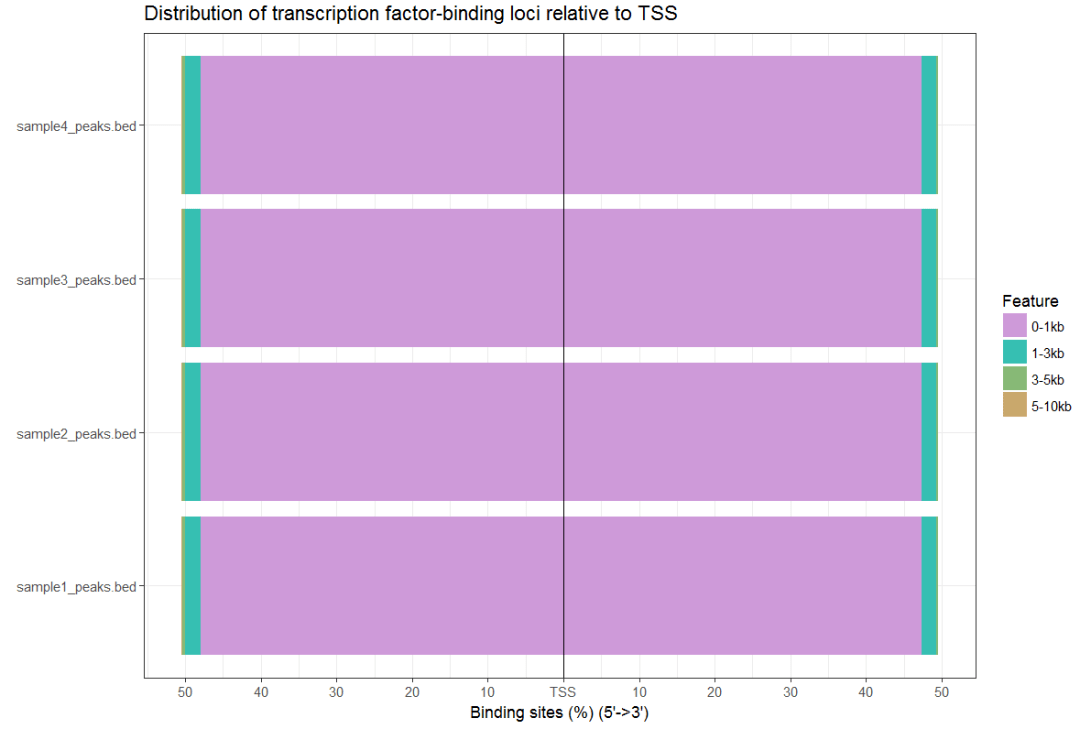
#画维恩图
genes <- lapply(peakAnnoList, function(i)as.data.frame(i)$geneId) vennplot(genes[2:4], by="Vennerable")
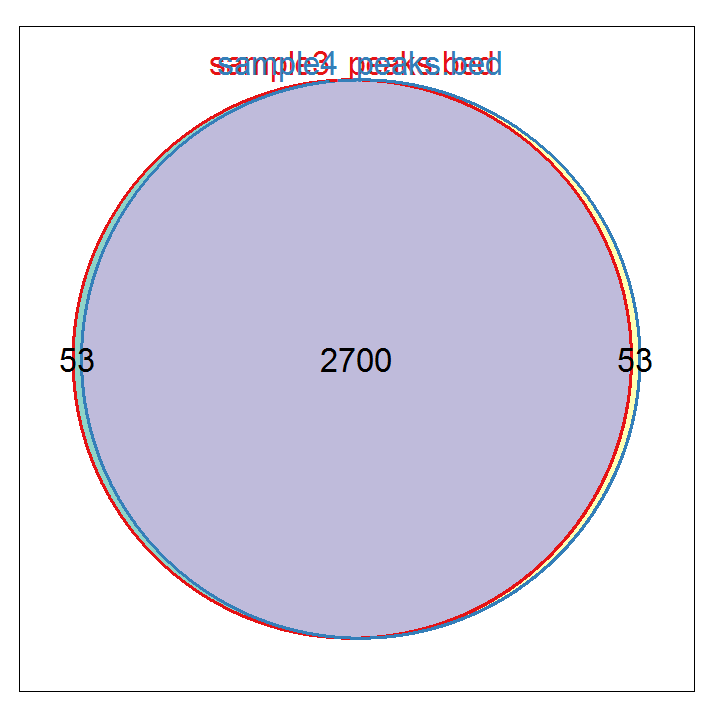
#输出注释信息
peakAnnotable<-as.data.frame(peakAnno) write.table(peakAnnotable,file="peakanno.result",sep="\t",quote=F)
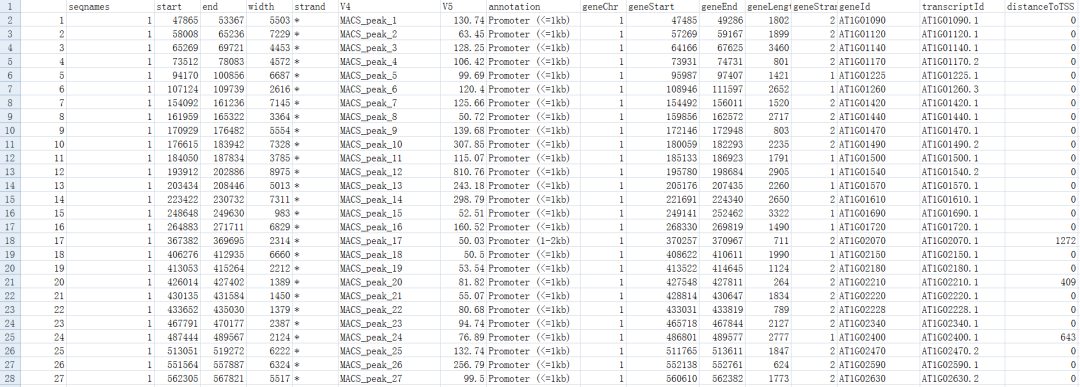
转自:科研日精进
发表评论: