大家好,今天和大家分享的是一月份发表在Front Aging
Neurosci(IF:3.633)杂志上的一篇文章,“Integrative Analysis of Hippocampus Gene
Expression Profiles Identifies Network Alterations in Aging and
Alzheimer’s
Disease”,文章中作者通过加权基因共表达网络分析以及加权蛋白互作网络分析,对GEO数据库中,海马体在年轻,年老以及阿尔兹海默症患者(AD)中的基因表达进行分析,为衰老和AD之间的复杂关系提供了新的见解。
Integrative Analysis of Hippocampus Gene Expression Profiles Identifies Network Alterations in Aging and Alzheimer’s Diseaser
海马体基因表达谱的综合分析确定衰老和阿尔茨海默氏病的网络变化
一、研究背景
阿尔兹海默症(AD)是一种神经退行性疾病,它会导致认知功能迅速下降最终导致痴呆,大多数AD病例为老年人。了解衰老和AD之间的关系,有助于获得与该疾病相关的标志物以及特定于细胞类型的识别方式。通过利用年轻人(20-50岁),老年人(70-99岁),AD患者的海马体基因表达谱进行分析,获得对衰老与AD关系更高层次的网络理解。
为何说本文看似做了很多的分析呢?我们简单来概括下:
1.WGCNA将数据分成了15个模块,常见的分析有“模块和临床变量相关性分析”。此外还用了5个数据集做进行验证模块的可重复性(中文翻译是模块保存分析,英文是module preservation analysis);
2.对不同模块间表达量在Young,Age和AD间做比较(柱状图,并无统计检验)
3.Young,Age和AD间进行差异分析后,对DEGs的数量用韦恩图可视化
4.不同模块间功能注释,注释后挑选一大堆的可能用于解释的通路
5.常见的PPI,鉴定出hub基因,并将这些hub基因的表达量变化(logFC)和Young,Age和AD联系起来
因此,基于上面的这些分析和可视化的方式,让本篇文章看起来格外“充实有料”!其实理清楚分析的思路,大概就是作者基于表达数据进行WGCNA,不仅使用5个GEO数据集验证了模块的可重复性,并且在各个模块中与临床变量,细胞,功能相联系,最后就是PPI来鉴定hub基因,然后将这些hub基因和Young,Age和AD相互联系。
二、研究思路

三、结果解析
1、加权基因共表达网络构建及模块—特征相关性分析
使用了18,754个基因来识别和描述与衰老和 AD 有关的模块,并 利用所有样本构建了一个独立于临床信息、年龄和性别的共表达网络。作者发现了15个共表达基因模块:
M2(黄色)、 M3(绿黄色)、 M4(洋红色)和 M5(粉红色)的 ME 表达值与衰老和 AD 呈正相关。
M4和 M5模块与老化,M2和 M3模块与 AD 有很强的相关性
M9(棕色)、 M10(绿松石色)和 M12(棕褐色)的 ME 表达值与衰老和 AD 呈负相关,但M9与M10与AD的关系相较于其与衰老的关系更强
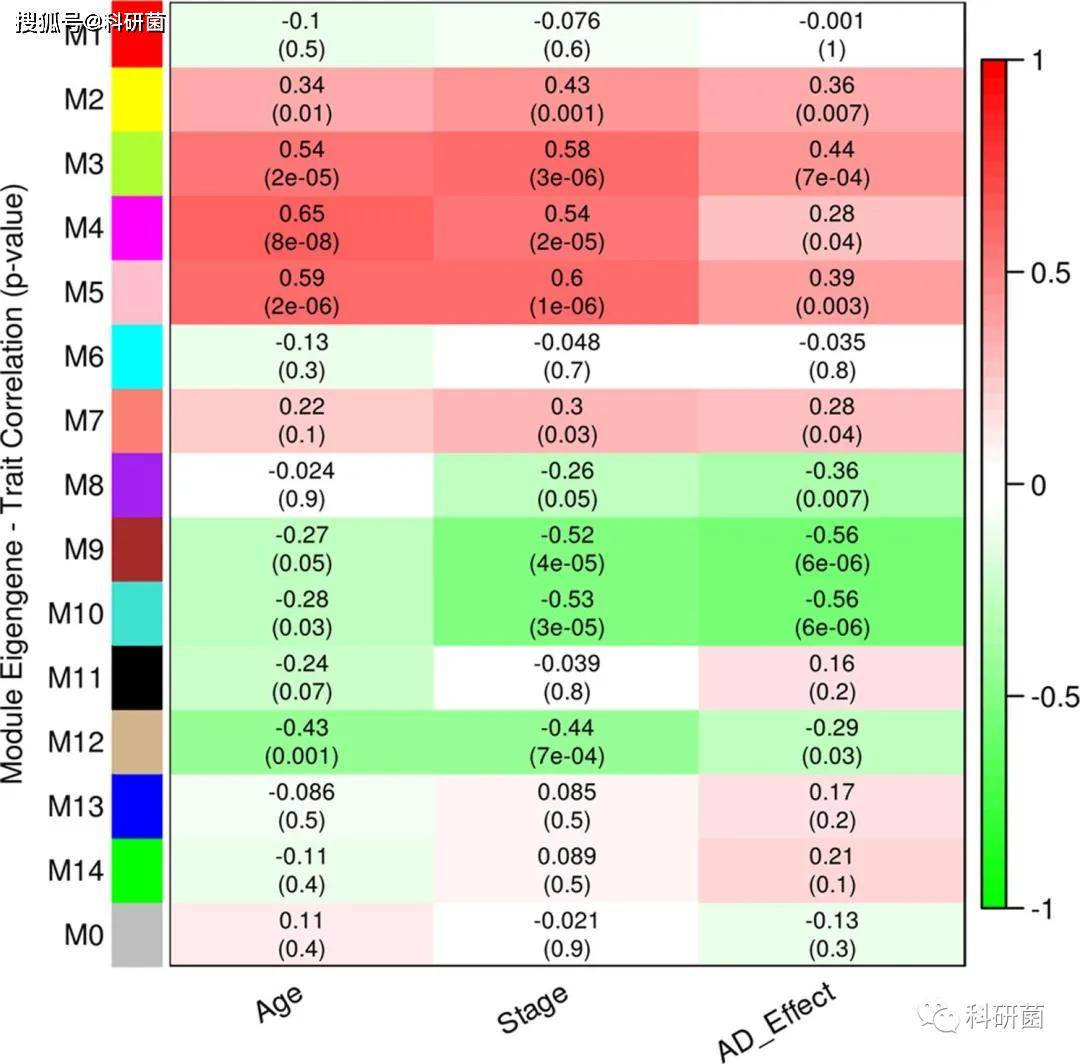
图2.模块特征基因表达值与各模块年龄,阶段以及AD的相关性
2、不同分组中的模特征基因(ME)表达值分析及差异基因分析
作者对不同分组样本之间的ME表达值进行了分析,结果显示,ME表达值存在组间差异。在AD与其他组别的比较中
模块 M2、 M3、 M4、 M5的基因表达上调
模块 M9、 M10和 M12的基因表达下调。
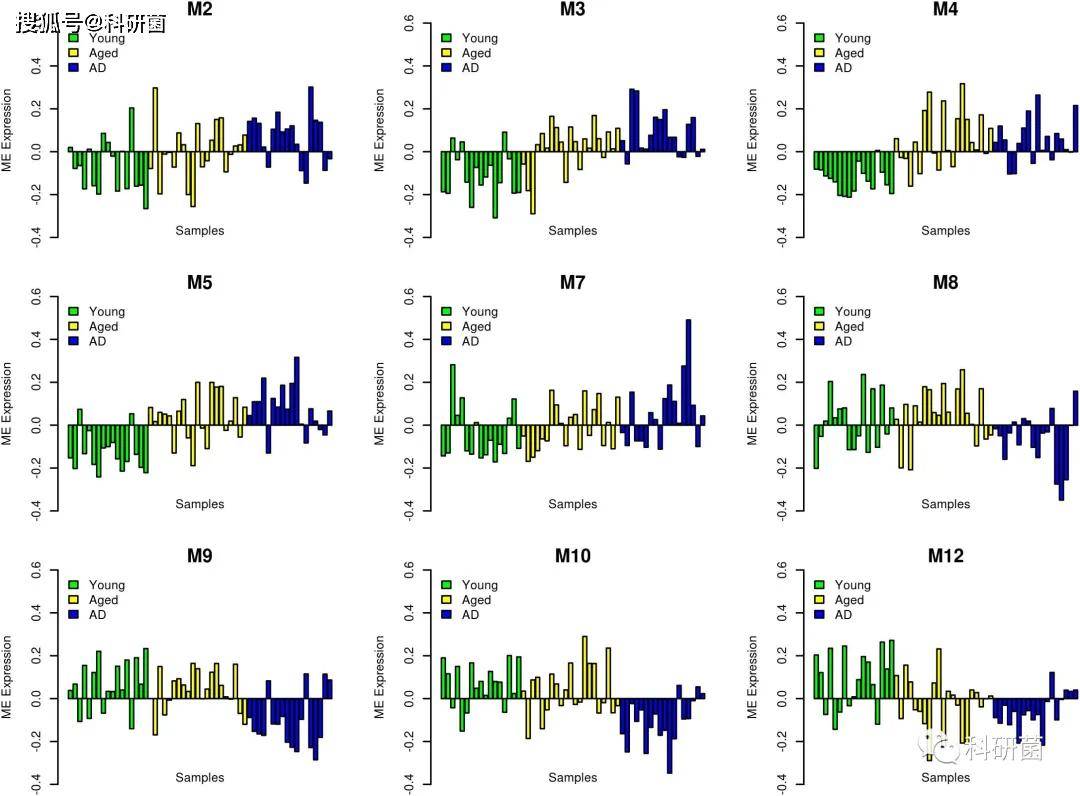
图3.ME表达值跨样本分析 样本分为:年轻(绿色)-老化(黄色)-AD (蓝色)。
随后,作者对不同分组中的差异表达基因(DEGs)进行了模块映射,绘制了年轻与老年、年轻与老年与老年与AD之间的上下调DEGs重叠图
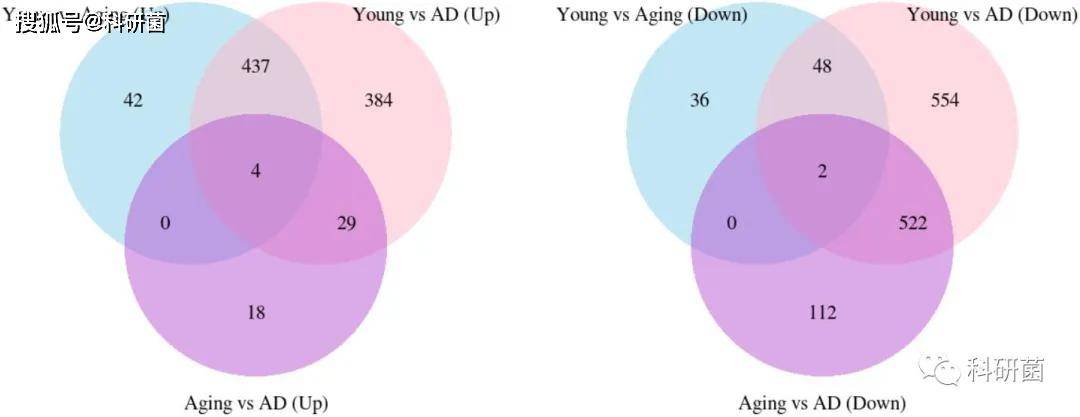
图4.DEGs上下调结果
此外,作者还对男女之间的ME表达差异进行了探究,研究发现
M5和 M7的 ME 表达值显示年轻人和老年人之间的差异取决于性别
M4(女性:R=0.77,p 值为1E-04; 男性:R=0.63,p 值为0.004)和 M5(女性:R=为0.72,p 值为5E-04; 男性:R=0.5,p 值为0.03)的性别效应程度略有不同
M7仅与女性组的衰老程度相关(R=0.46,p 值为0.05)
M8模块也显示了年轻组女性和男性之间存在表达差异
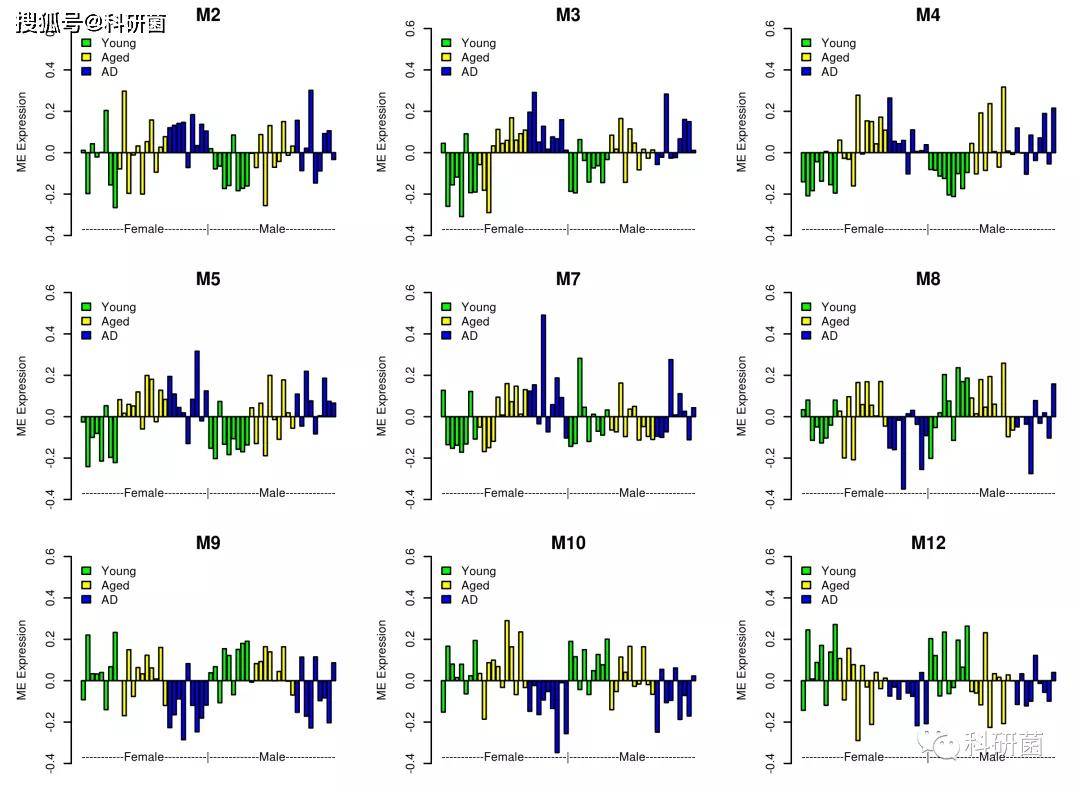
附图3.ME表达值跨样本分析(附加性别分组)
3、细胞型特异基因与模块的重叠
通过针对细胞型特异基因的分析,作者发现:
模块 M3和 M11(黑色)与星形胶质细胞有关模块 M8、 M9和 M12与神经元有关,模块 M5主要与内皮细胞有关,模块 M4与小胶质细胞有关,模块 M1(红色)与少突胶质细胞有关。
另外一方面,单元模块M10和M2与细胞型关联的意义较小
少突胶质细胞模块特异性与衰老和 AD 均无显著相关性
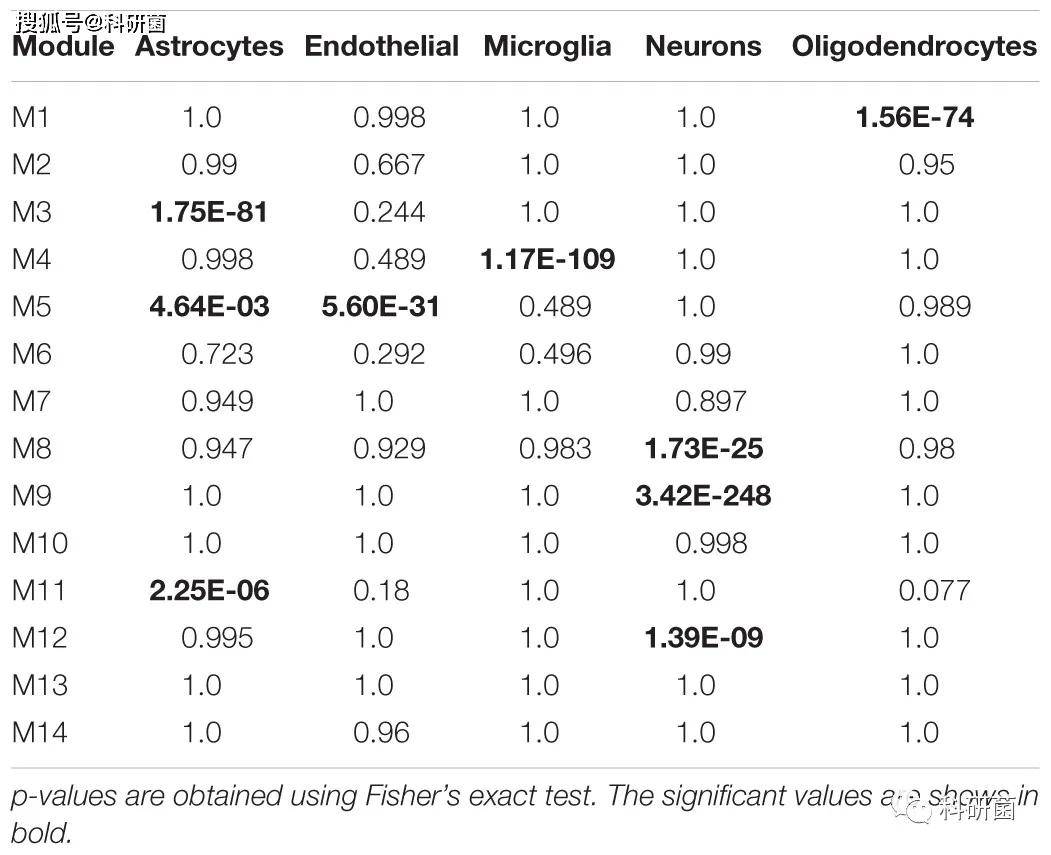
表1.细胞类型特异基因和模块之间的重叠
4、在多个GEO数据集中进行(module preservation analysis)模块保存分析
作者在这里使用利用 GSE1297、 GSE36980、 GSE84422、 GSE29378(包括 CA1和
CA3)和神经元丰富样本(GSE28146、
GSE5281)进行模块保存分析(用于验证WGCNA分析结果的迁移性,类似机器学习中的验证集),并使用Zsummary进行模块保存评分。
大多数与衰老和 AD 相关的模块显示中度至高度保存
与其他模块相比,M1、 M8、 M9、 M10和 M12模块具有较高的保存性,神经元(M8,M9和 M12) ,小胶质细胞(M4) ,内皮细胞(M5)和星形胶质细胞(M3)的模块特异性保存在多个数据集中
由于 AD 中神经元和神经胶质细胞同时受到影响,作者认为 AD 中神经元-神经胶质相互作用可能受到影响。
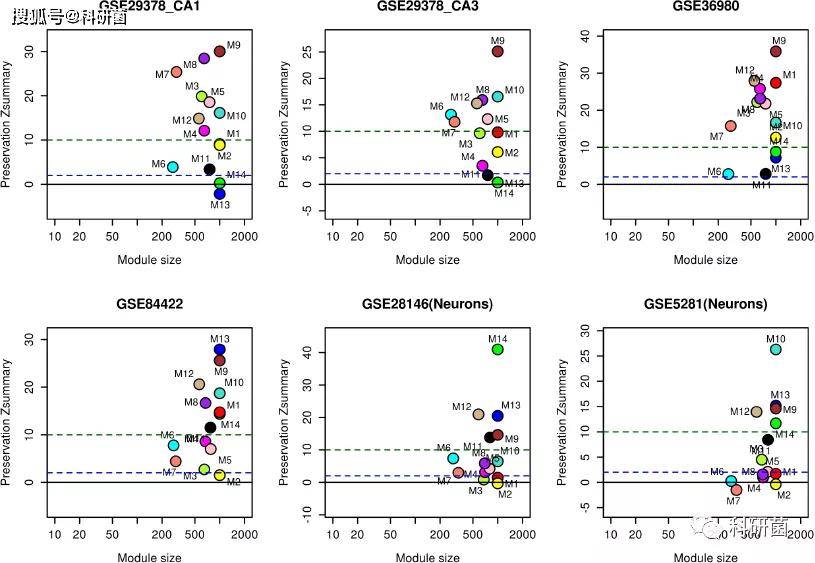
图5.利用海马数据集进行模块保存分析(Zsummary评分)
5、利用DAVID数据库进行GO基因富集分析
作者利用DAVID对生物过程和 KEGG 通路进行了功能富集分析。
小胶质细胞模块M4与生物学过程炎症反应、KEGG 途径吞噬体、Toll样受体信号传导和细胞因子-细胞因子受体相互作用有关。细胞组成与 MHC II 类蛋白复合物有关
核心基因包括:TYROBP、 TREM2、 ITGB2、 MYO1F、 C1QA、 C1QB、 C1QC 和 TGFB1
内皮细胞模块M5也与炎症反应和TNF 信号通路、补体和凝血级联以及 HIF-1信号通路相关,细胞组成与 MHC Ⅰ类蛋白复合物和细胞外间质蛋白复合物(ECM)有关
核心基因包括:TNFRSF1A、 MSN、 CLIC1和 IFITM2
星形胶质细胞模块M3与生物过程中的细胞粘附、KEGG通路中的脂肪酸降解和 HIPPO 信号通路
核心基因包括:EZR、 CDC42EP4、 ARHGEF26、 ARHGEF6
M2模块与生物过程中RNA间接,KEGG通路中核糖体,剪接体以及RNA转运有关。
核心基因包括:TFEB、 PAN2和 ARHGAP17
神经元相关模块M9与生物过程中化学突触传递、神经递质分泌和神经系统发育,KEGG通路中突触小泡、化学突触(谷氨酸能、胆碱能、
GABA能、血清素能和多巴胺能)和长时程增强作用,细胞组成神经元投射、树突(树突形态发生)、轴突(轴突 / 轴突导向)和突触后密度有关
核心基因包括:SYN1、 STMN2、 SYT5、 SNAP91、 PAK3、 UCHL1和 UBE2K
神经元相关模块M12中基因GADP1、 YWHAZ、 SYNJ1和 MAPK9随着年龄的增长而显著减少,而G3BP2和 ATP6AP2则随着 AD 的增加而显著减少
M10作为一个与AD相关的下调模块,与线粒体、核糖体和蛋白质折叠相关,KEGG通路包括氧化磷酸化、蛋白酶体、剪接体、氨酰基转移核糖核酸生物合成和内质网蛋白质加工,这其中包括蛋白质靶向、蛋白质相关降解和泛素连接酶复合物,表明AD患者的线粒体和ER功能受到影响。
核心基因包括:NDUFAB1、 VDAC3、 ATP5G3、 COPS4、 RTCA 和 POP4
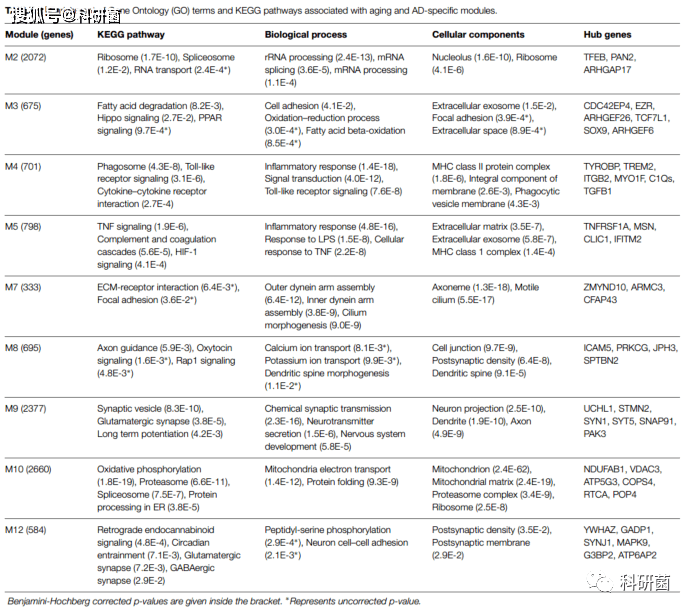
表2.GO富集和KEGG通路与衰老及AD关联模块分析
在少突胶质细胞M1中,作者发现大多数基因随着年龄的增长而下调,但在 AD 中下调的程度减少,有些病人这个模块被上调
在这个模块中髓鞘相关蛋白(MBP、 MOB 和 MOG)与髓鞘形成和少突胶质细胞前体分化的负性调节因子(LINGO1)一起在衰老过程中下调,指示髓鞘损伤和修复的动态稳态,可以掩盖其在老化和 AD 发病机制中的后果
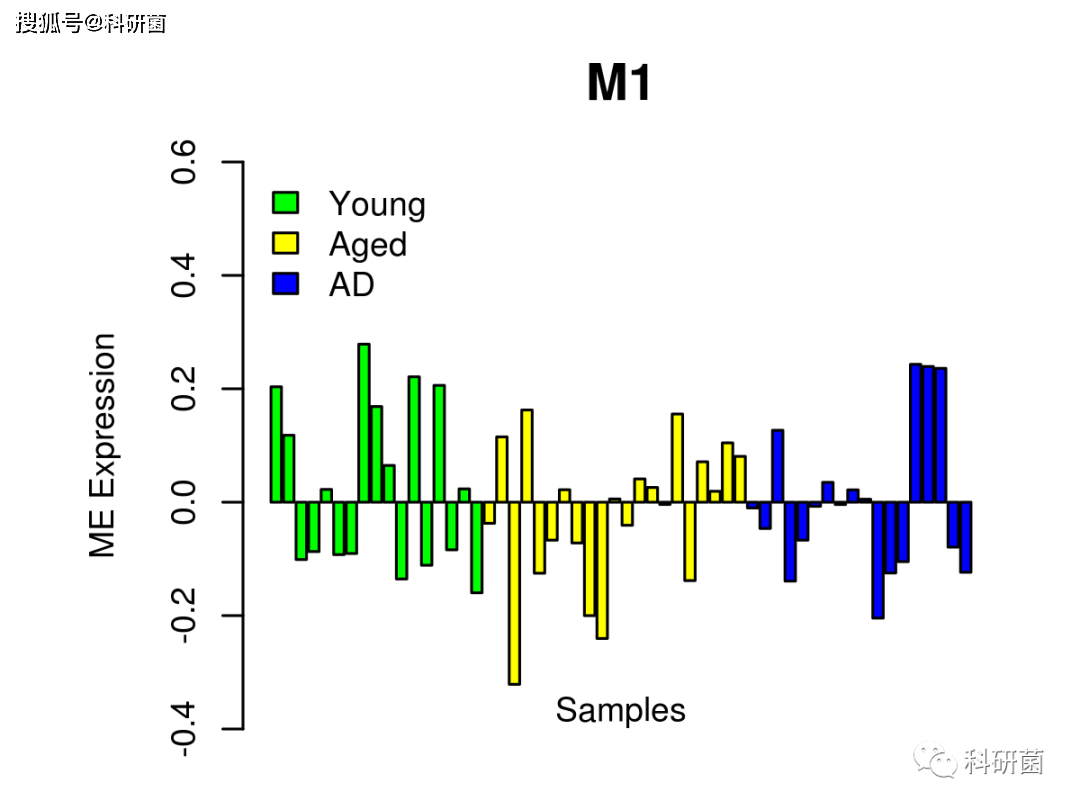
附图4.不同分组的少突胶质细胞M1模块基因表达
6、基因表达定位到人蛋白质相互作用网络的图论研究
作者将不同分组的基因表达与PPI网络结合,得到加权的PPI网络。综合PPI网络用来确定交互作用以及关系的测量。只有关系边界介于2000和 adj p 值0.05之间的相互作用才被认为是改变的相互作用。并绘制了老化和AD的上下调基因子网络。
在AD中,节点和相互作用的数量增加了。AD 中的大多数改变增加了老化(子网络)中存在节点的度,从而导致子网络中hub基因数量的增加。(如果对度等拓扑学名词不大明白可以看看这篇推送)
基于节点度,作者识别了老化和AD的PPI子网络中的枢纽基因及其相互作用。
CD44、 VEGFA、 HIF1A、 VIM、 FOS、 CEBPB、 CDKN1A、 SHC1、 tgf 1和 SYK 作为上调衰老的子网络枢纽基因
在衰老过程中观察到,VEGFA 与 HIF1A 的相互作用增强。结果指示HIF1A、 PFKFB3和 LDHA
之间的关键相互作用,它们调节有氧糖酵解的代谢开关。作者指出,与年轻人相比,随着年龄的增长,己糖激酶2(HK2)和丙酮酸脱氢酶激酶1(PDK1)基因的上调也进一步证实了这一点。
DNA损伤应答基因 CDKN1A 与其相互作用基因 GADD45B 在衰老过程中上调。
随着年龄的增长,星形胶质细胞标志物 GFAP、 S100A8、 ALDH1L1和 CHI3L1的表达也持续增加(图6A)
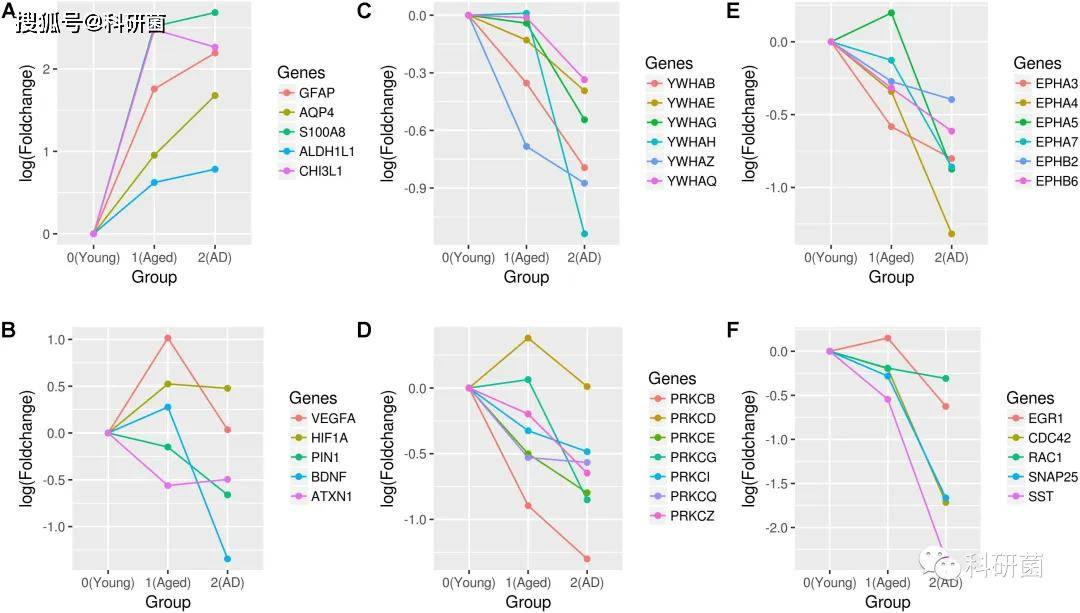
图6.与衰老和 AD 相关基因的表达谱。(a)星形胶质细胞标记物、(b)神经保护基因(VEGFA、 HIF1A、 PIN1、 BDNF 和
ATXN1)、(c)14-3-3蛋白、(d) PRKC 亚型、(e) ephrin 受体、(f)突触和肌动蛋白细胞骨架基因(EGR1、
CDC42、 RAC1、 SNAP25和 SST)的折叠变化。Foldchange的变化基于年轻组的表达。
在衰老网络中,免疫炎症相关的的基因也存在上调(CEBPB、 FOS、 STAT3、 tgf 1和 SYK)
在AD的子网络中,VEGFA不再是一个中心基因,与老化子网络相比,其相互作用的数量显著减少。可以观察到观察到血管内皮生长因子脂肪酸(VEGFA)在老化过程中表达增加,而在 AD 过程中表达减少(图6B) 。这提示 AD 患者海马区存在血管功能障碍的可能性。
与衰老相比,AD 中14-3-3家族的基因表达下调(图6C)
在 AD 中,作者还观察到丝氨酸 / 苏氨酸激酶 PKC 家族成员与编码激酶的基因肉瘤酪氨酸激酶(SRC)和 ephrin 受体一起下调(图6DE),有研究表明,这些蛋白激酶活性随着 AD 阶段的减少。
小结
这篇文章看上去很复杂,但如果了解了WGCNA以及PPI的相关分析方法后就会发现文章本质上并没有什么做起来的难点。作者通过一个数据库构建了基因共表达网络后,对不同分组中的ME表达进行了分析,随后将细胞型特异性和ME相关联,在多个GEO数据集中验证模块保存。随后作者使用DAVID数据库对基因表达进行富集分析,并根据既往文献结果进行论述。除此之外,作者还通过基因表达定位的PPI互作网络进行深入探究,使用R中的igraph包进行不同分组之间基因的关联进行评分度量,指示了部分AD与衰老之间的基因差别及对于发病机制的可能贡献,并同样依据既往的文献进行了进一步的讨论。