ROSE 软件挖掘「超级增强子」实战与代码分享
2025-9-3 萌小白
一、ROSE 软件是什么?
ROSE(Rank Ordering of Super-Enhancers)由 MIT Richard A. Young 实验室开发,是目前引用率最高的超级增强子预测工具。
rose软件下载地址:https://bitbucket.org/young_computation/rose/
软件分析原理:
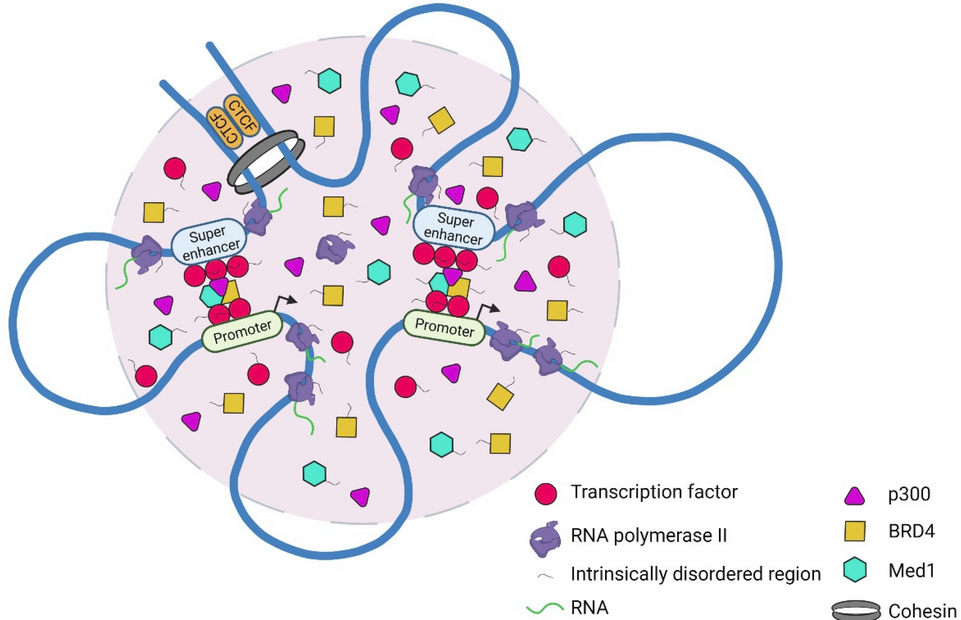
先用 H3K27ac(或 MED1/BRD4 等)ChIP-seq peaks 定义“增强子”区间;
将彼此 ≤12.5 kb 的增强子“缝合”(stitched)成连续区域;
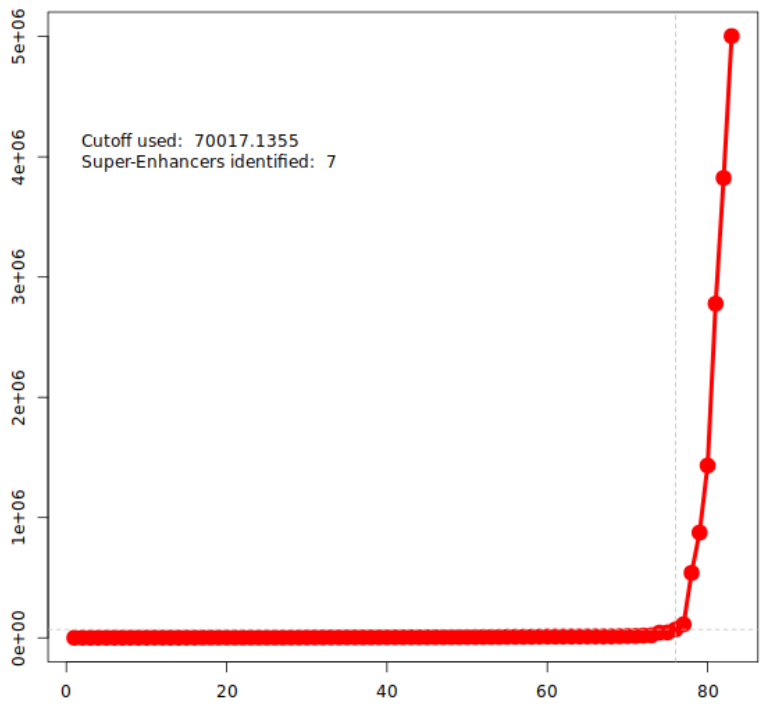
根据信号强度排序,拐点(inflection point)以上即为超级增强子,以下为普通增强子(typical enhancer)。
二、安装步骤与环境依赖
安装系统:Linux(CentOS/Ubuntu)
环境依赖:
Python 2.7(注:不可使用 Python3环境,会报错!)
R ≥ 3.6(用于绘图)
SAMtools 0.1.18(新旧版亦可,需支持sort -o)
一键下载:
三、输入文件准备
文件 | 说明 | 工具/注意事项 |
|---|---|---|
bam | ChIP 样本(H3K27ac) | bam文件需要有.bai索引文件 |
control bam(可选) | Input 或 IgG 对照 | bam文件需要有.bai索引文件 |
gff/bed | 增强子初筛结果 | 通常用 MACS2 鉴定的 broad peaks |
注意:软件默认支持人(hg19 / hg38)和鼠(mm9 / mm10)的参考基因组。
四、一条命令跑完 SE 鉴定
常用参数:
-g:基因组版本(内置 hg19/hg38/mm9/mm10)
-s:缝合距离,默认 12.5 kb
-t:排除 TSS ±2–3 kb,避免启动子干扰
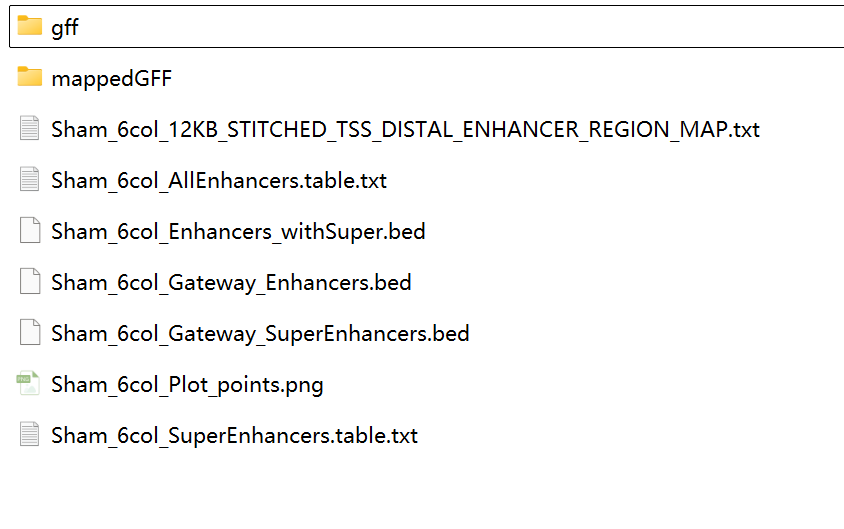
主要输出:
*_SuperEnhancers.table.txt—— 超级增强子列表
*_AllEnhancers.table.txt—— 所有增强子(含 TE/SE 标签)
*_Enhancers_withSuper.bed—— 可直接导入 UCSC/IGV 浏览器
*_Plot_points.png—— rank-plot 拐点图
五、SE 靶基因注释
使用ROSE_geneMapper.py对上一步生成的*_SuperEnhancers.table.txt进行基因注释,代码如下:
得到:
TO_GENE.txt —— 每个 SE 邻近基因(50 kb 窗口)
GENE_TO_ENHANCER.txt —— 基因 ←→ SE 反向索引
后续即可对 SE 关联基因做 GO/KEGG、GSEA、转录因子 motif 富集等分析。
六、非模式物种/自定义基因组
非模式物种/自定义基因组可参考以下步骤:
准备refGene.txt(UCSC genePred 格式)。
改名为mySpecies_refseq.ucsc放入annotation/。
修改ROSE_main.py内GENOME_DICT,添加新基因组键值。
其余步骤同上。
七、常见问题
染色体命名 —— bam、gff 必须统一带或不带chr(需要注意参考基因组信息!)。
Python2 环境 —— 若系统默认 Python3,可通过 conda 创建Python2环境:
conda create -n rose_py2 python=2.7
八、小结
ROSE 以“缝合-排序-拐点”三步法,把 ChIP-seq 峰直接转换成生物学意义明确的超级增强子列表。参考使用上述代码可快速完成从原始 bam 到SE鉴定及注释的全过程。
评论:
发表评论: