引
很多老师在写文章的时候,精力通常会放在文章中的技术或者实验细节,而疏忽原始数据上传的工作。文章准备接收时,审稿人需要老师提供文章中使用数据的登录号,一般时间要求较紧,对于没接触过数据上传的老师来说,可能会成为文章接收最后的绊脚石,为此我们撰写了一份高通量测序数据上传指南,可以让老师快速掌握数据上传的技能,助力老师文章发表。
目前高通量测序原始数据通常上传到NCBI的SRA(The Sequence Read
Archive)数据库,部分老师会纠结是否需要把测序数据上传至GEO数据库,这里无需多虑,因为测序数据上传到GEO之后,最终也是存放在SRA;而且SRA相对于GEO来说,上传过程更快速、便捷。
本指南以上传SRA数据库为例,整体篇幅以截图为主,并辅以文字描述,方便老师对照网页操作。
在数据上传过程中可能会要求填写实验细节,有些可能不明确或者不适用,如果没有特殊需要,部分细节可以不用过于纠结。数据成功上传后会生成一个Accession
Number,最终加入文章中,审稿人或者其他人能够通过该Accession Number查询、下载到对应数据。
话不多说,直接上干货~
01
登录/注册
1.1 进入NCBI数据上传主页
主页链接
https://submit.ncbi.nlm.nih.gov/subs/,如图:

Sequence Read Archive:填写样本信息、上传样本数据
BioProject:填写项目信息
BioSample :填写样本属性
1.2 Login in/Register (有账号可以跳过1.2和1.3)
点击右上角Login,如果在NCBI注册过,可以输入账号密码登陆,如果未注册可以点击Register for NCBI account注册:


1.3 验证邮箱
注册完毕后会向所填写的邮箱中发送验证邮件,点击邮件中的链接即可激活账号。再次回到第二步之后的界面,此时会自动登录。
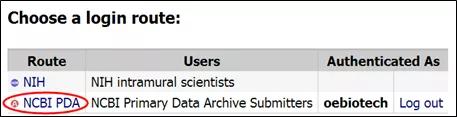
注:提交过程中如果出现以下的登陆方式选择,一律选择NCBI PDA登陆
02
创建研究项目-BioProject
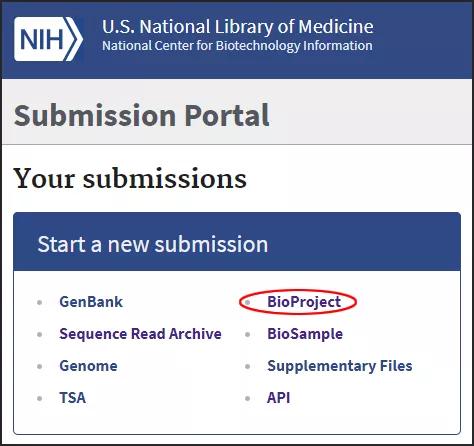
回到上传主页
https://submit.ncbi.nlm.nih.gov/subs/,点击BioProject:
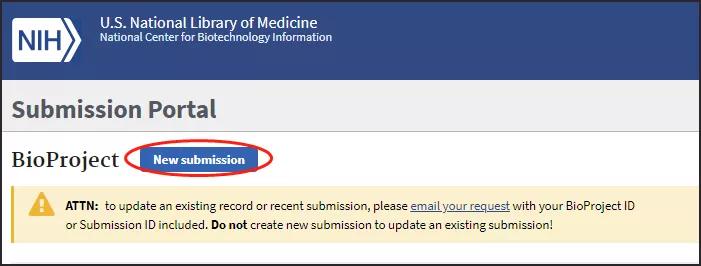
页面跳转后点击New submission:
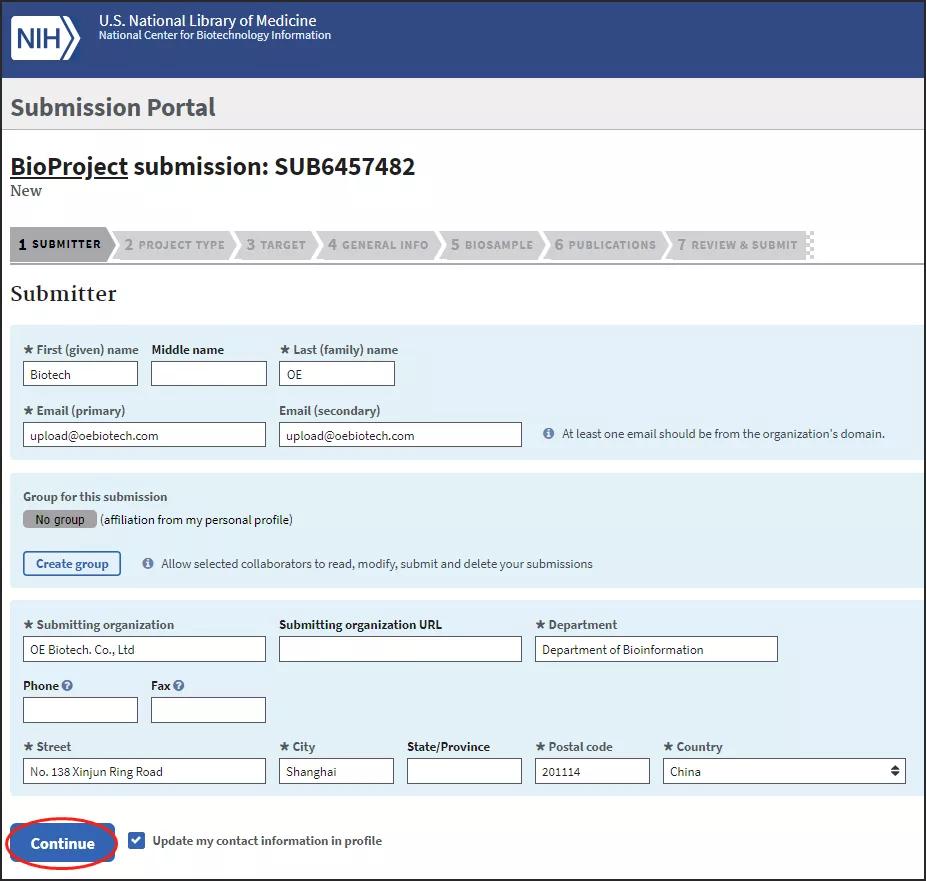
2.1 SUBMITTER
进入创建BioProject页面Submitter选项卡,填写必要个人信息:
2.2 PROJECT TYPE
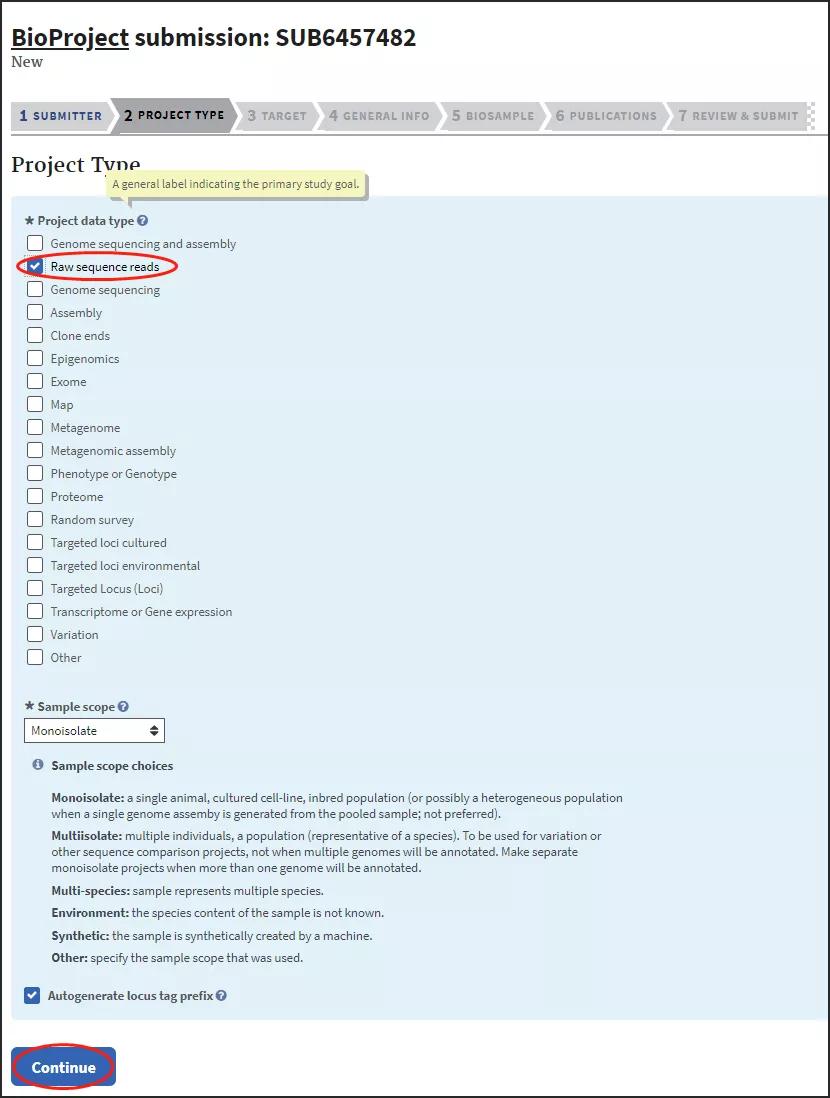
在Project Type选项卡中依次选择相应的类别,此处以普通转录组测序为例,勾选原始测序数据:
2.3 TARGET
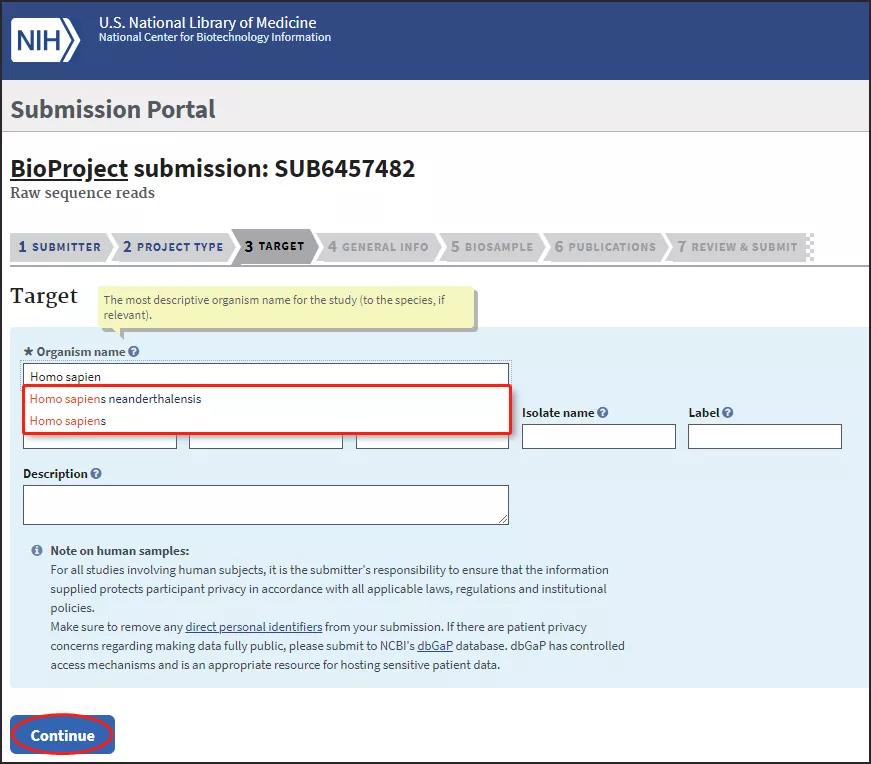
进入Target选项卡,填写物种名称 (此处填写的物种拉丁文名称需在NCBI中有收录,输入关键词后选择弹窗的物种信息,否则会提示找不到该物种名),继续下一步:
2.4 GENERAL INFO
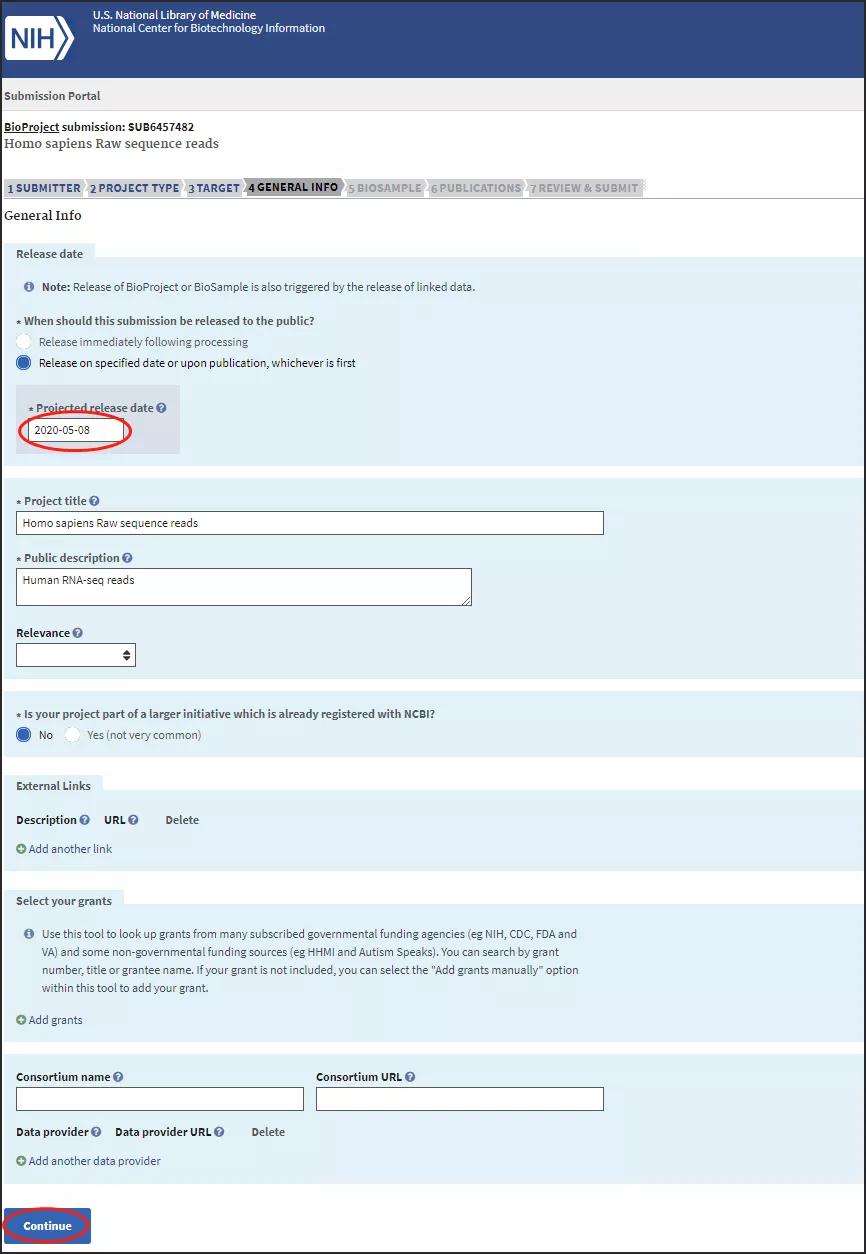
进入General Info选项卡;第一个选项是问此数据是否马上发布,还是指定特定日期发布(释放日期可以给NCBI写邮件更改)。其他填写带星号的必填内容,继续下一步:
2.5 BIOSAMPLE
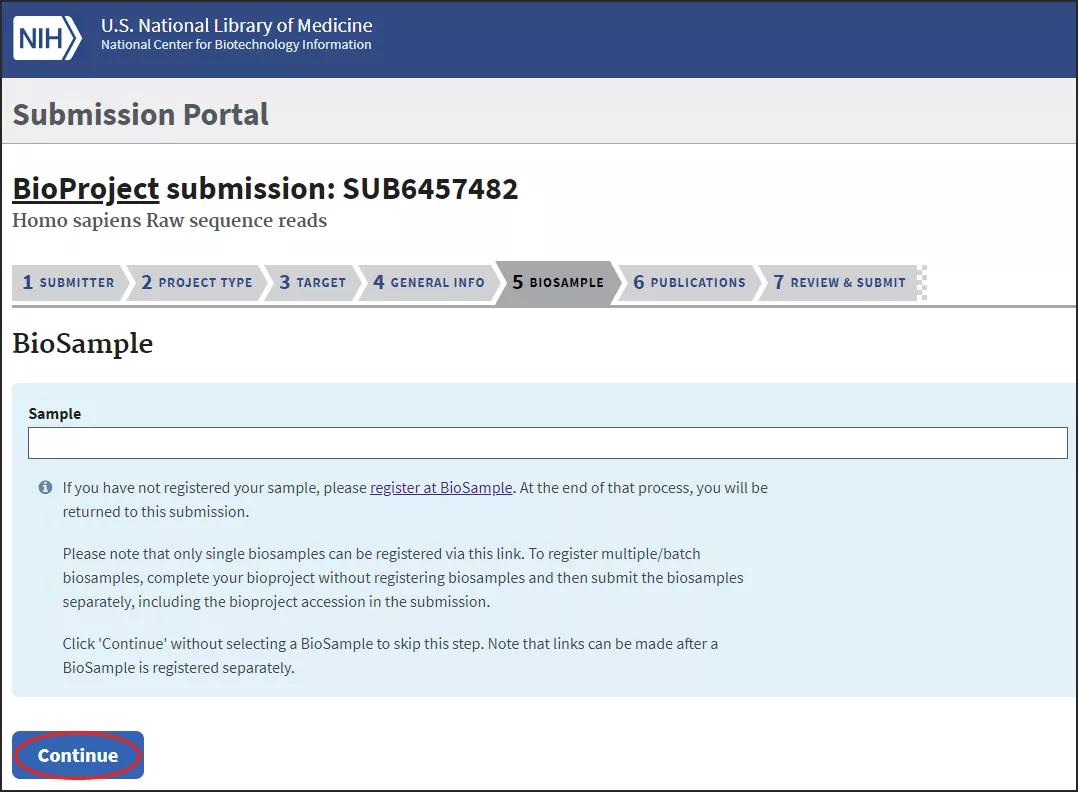
进入BioSample选项卡,需要指定项目中包含的BioSample,可以先略过,也可以先去创建BioSample。此处先略过,点击继续:
2.6 PUBLICATIONS
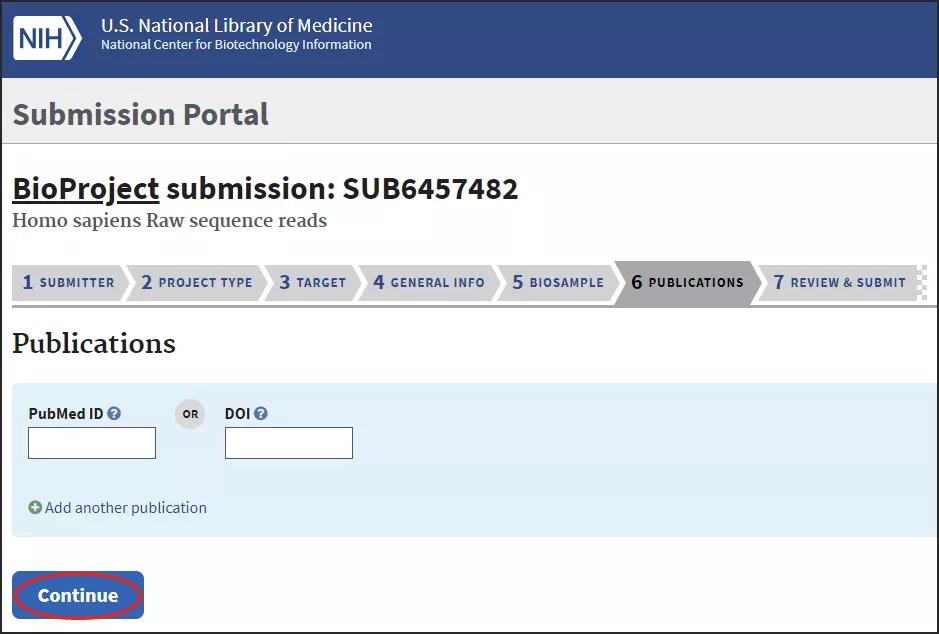
进入Publications选项卡,如有已发表文章,可以指定PubMedID或DOI,没有请略过:
2.7 OVERVIEW
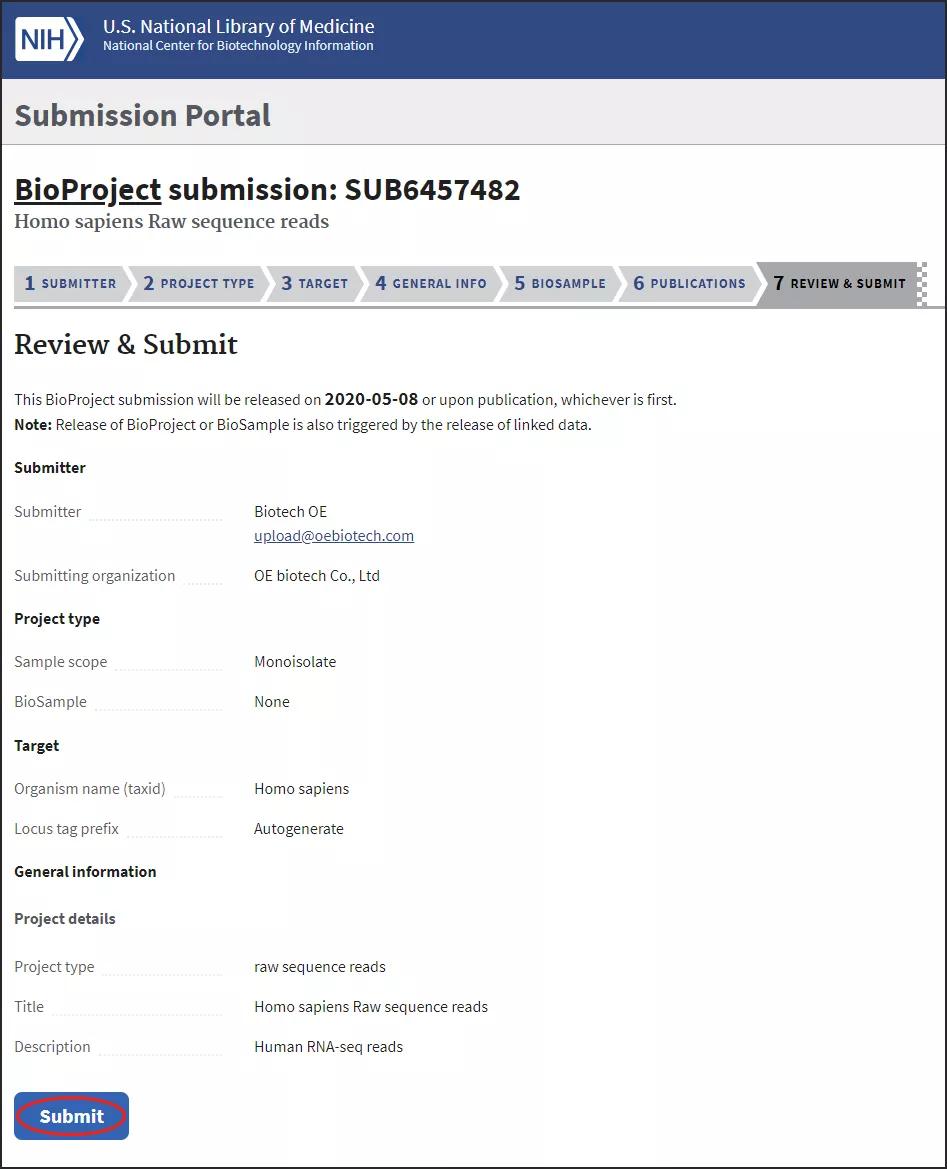
最后Overview中可以浏览概况,确认无误后点击Submit提交:
2.8 获得PRJ ID
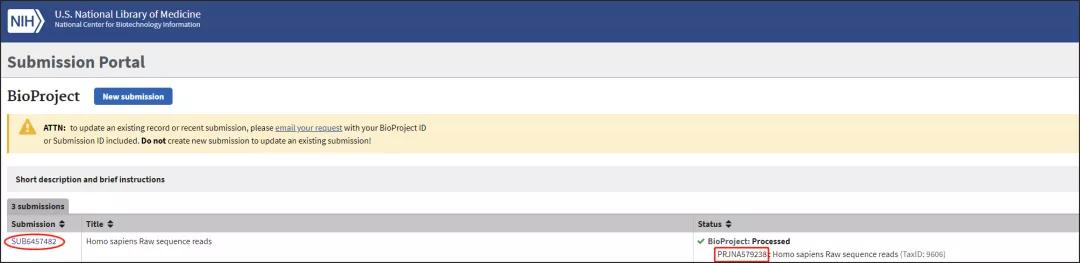
提交后会回到BioProject界面,列表中会显示刚才的项目已经提交正在审核(Awaiting processing)。一般几分钟就能审核完毕,刷新就可以看到Processed,并可以看到BioProject编号 (PRJ开头):
03
创建生物样品-BioSample
回到上传主页https://submit.ncbi.nlm.nih.gov/subs/,点击BioSample :

页面跳转后点击New submission:

3.1 SUBMITTER
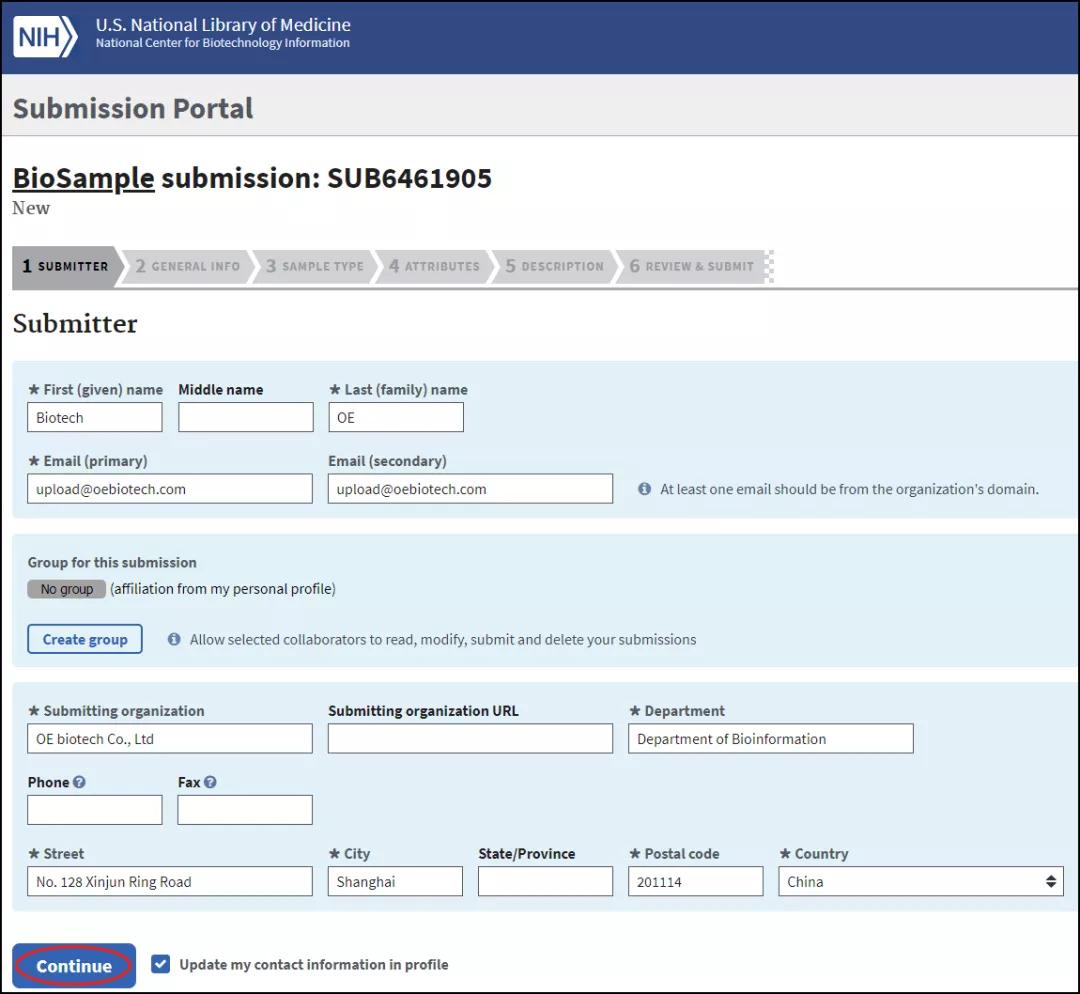
进入创建BioSample页面Submitter选项卡,填写/完善必要的个人信息:
3.2 GENERAL INFO
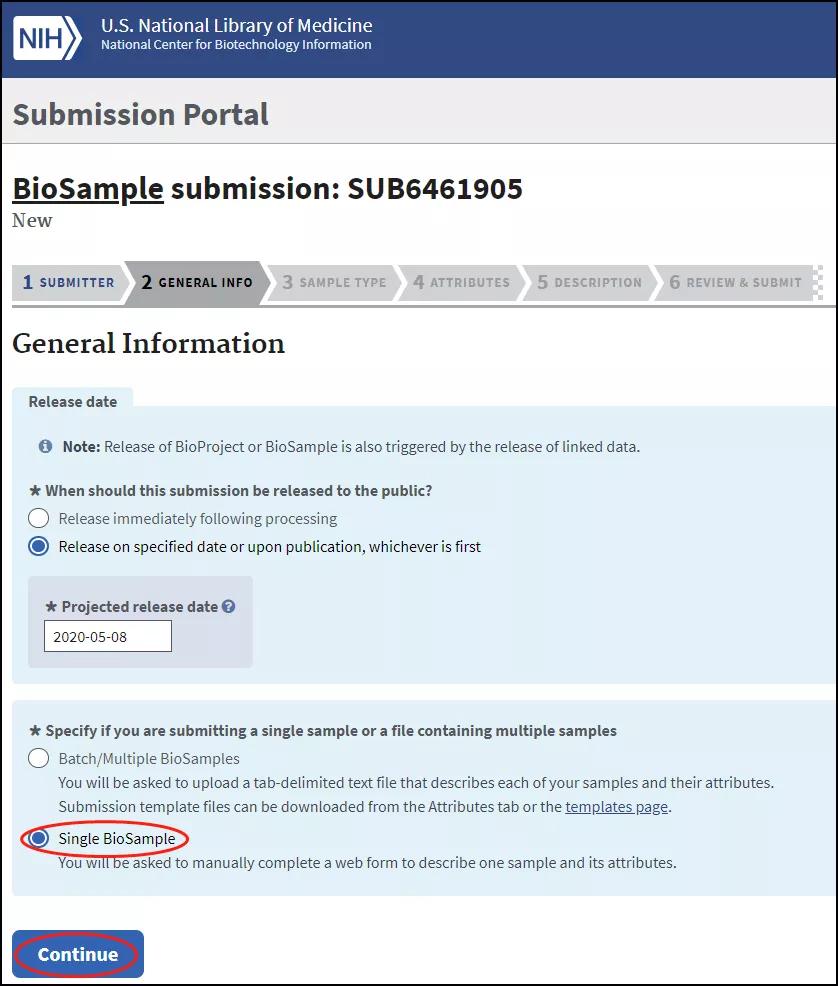
在General info选项卡中,第一个是选择是否立即发布(和上面的Bioproject的类似),第二个选择是问是否有多组样品,如果选多组样品的话,会要求上传表格文件,用于描述各样品的属性,比较繁琐;可选择单样品,不影响后续上传:
3.3 SAMPLE TYPE
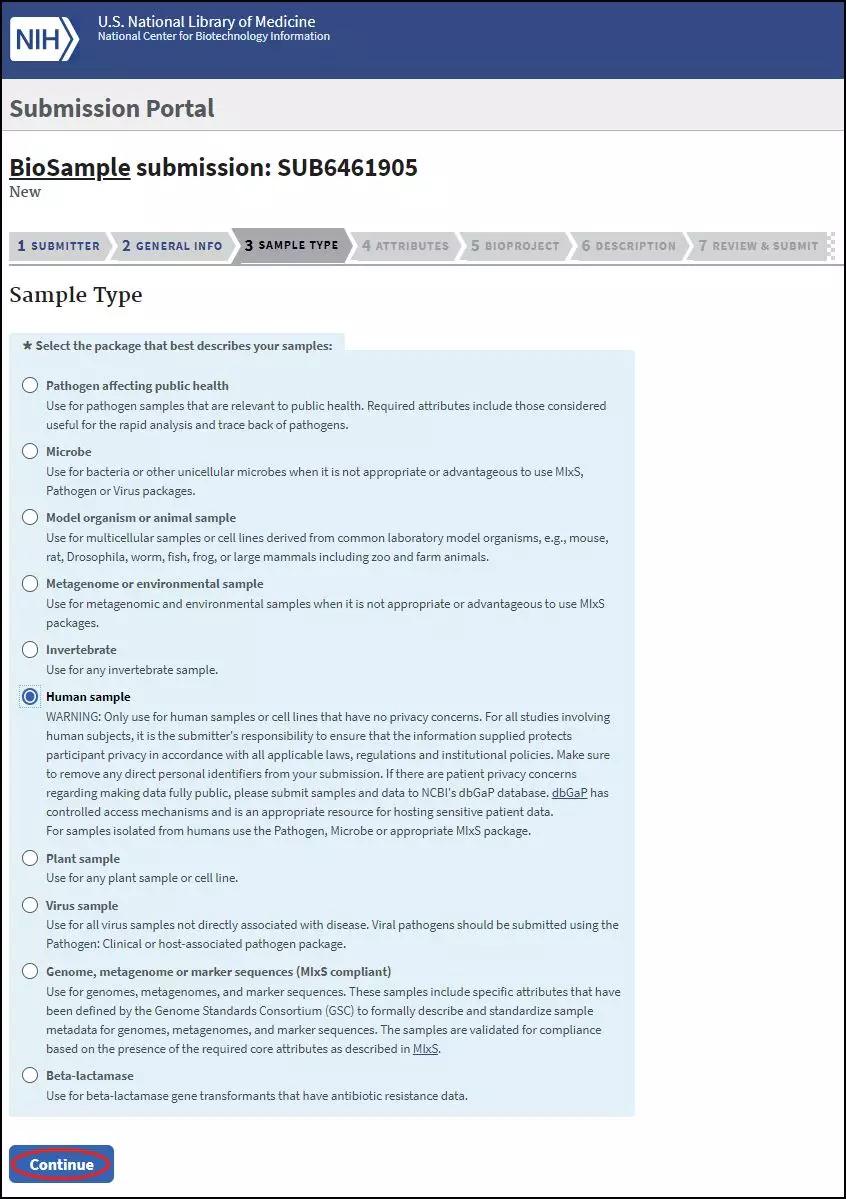
在Sample Type选项卡中根据样本实际情况选择:
3.4 ATTRIBUTES
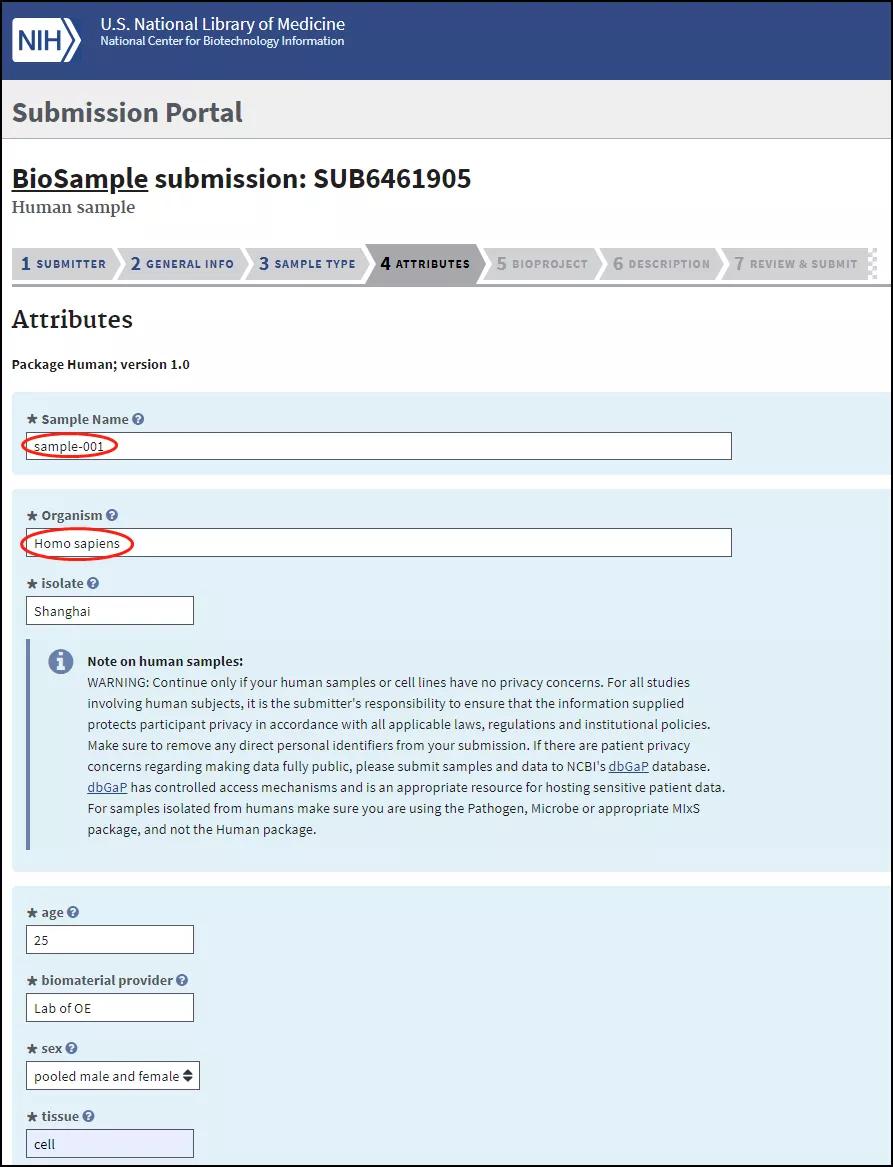
Attributes选项卡,按情况填写星号内容(此处Sample Name可以自行填写,物种信息参考上面Bioproject的操作,填写完整的拉丁文名称-需要下拉列表有显示对应的物种名):
3.5 BIOPROJECT
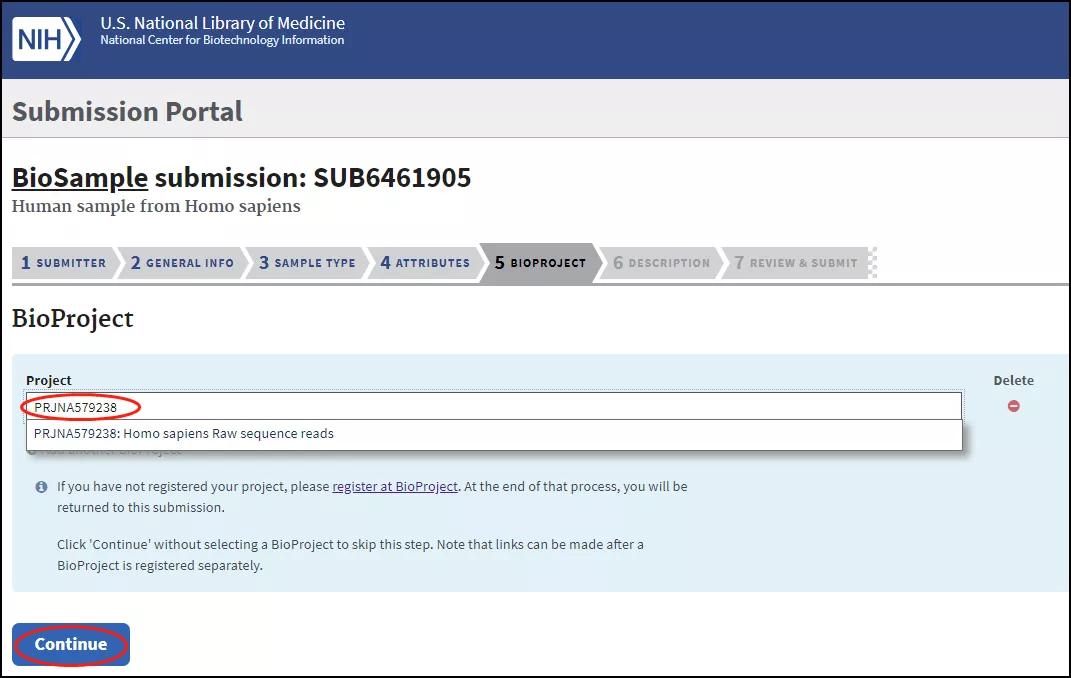
BioProject选项卡,输入刚才创建的BioProject编号:
3.6 DESCRIPTION
Description选项卡,信息如果不需要更改按照默认的即可:
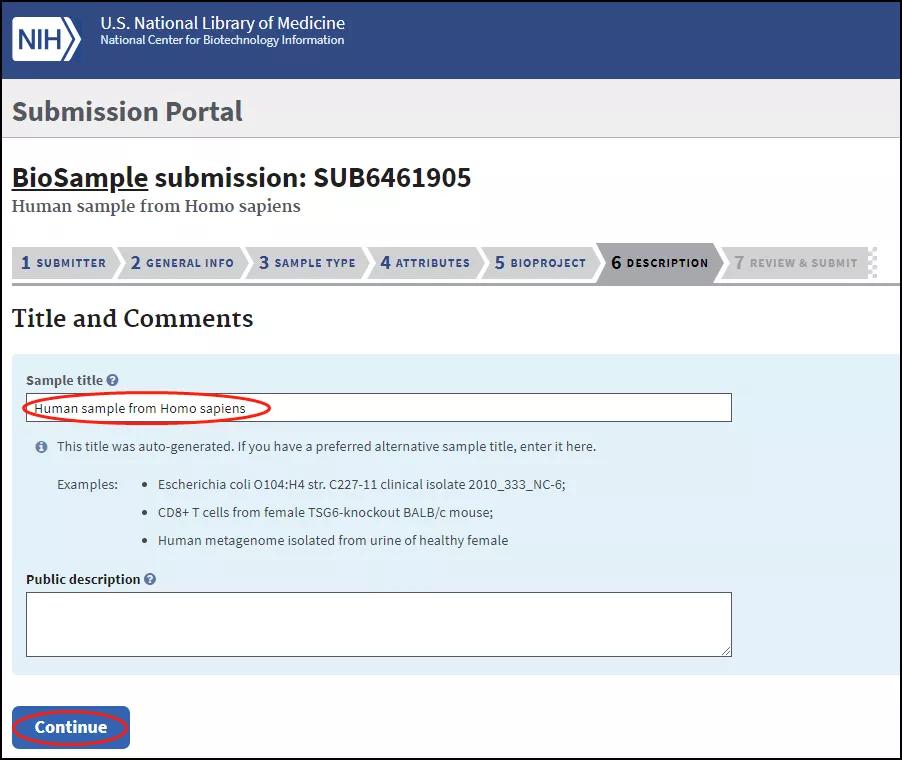
3.7 OVERVIEW
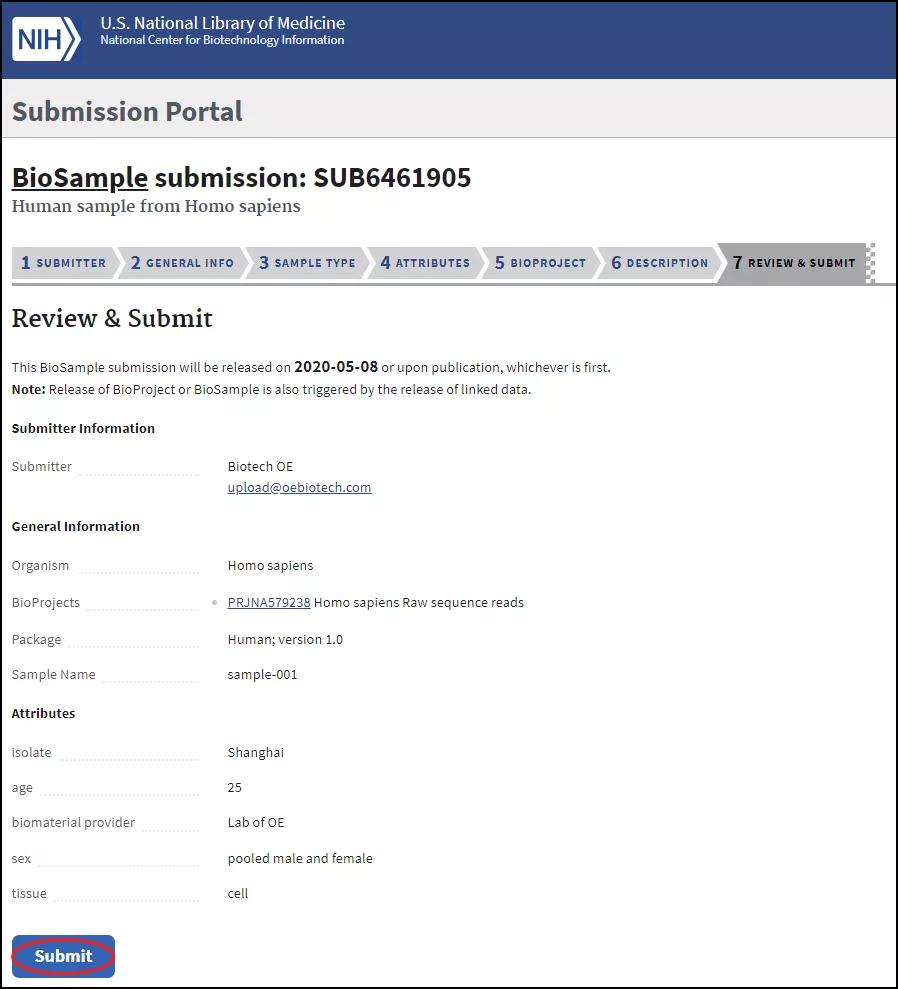
最后Overview中浏览概况,确认无误后点击Submit提交:
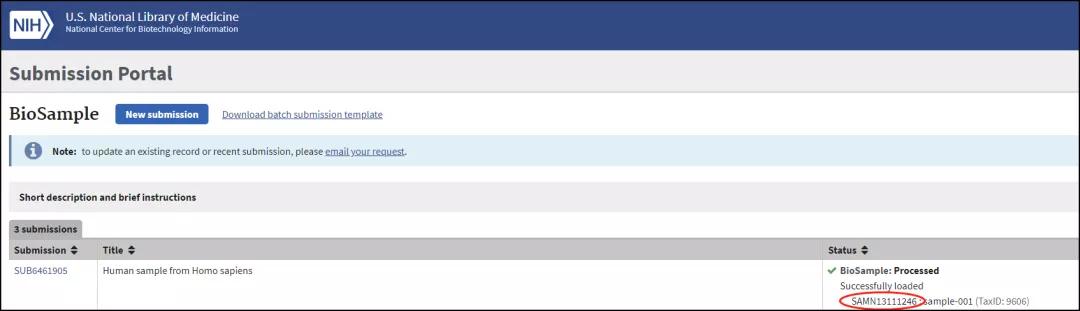
3.8 获得SAM ID
提交后会回到BioSample界面,列表中会显示刚才的项目已经提交正在审核(Awaiting processing)。一般几分钟就能审核完毕,刷新就可以看到Processed,并可以看到BioSample编号 (SAM开头):
04
创建SRA
回到上传主页
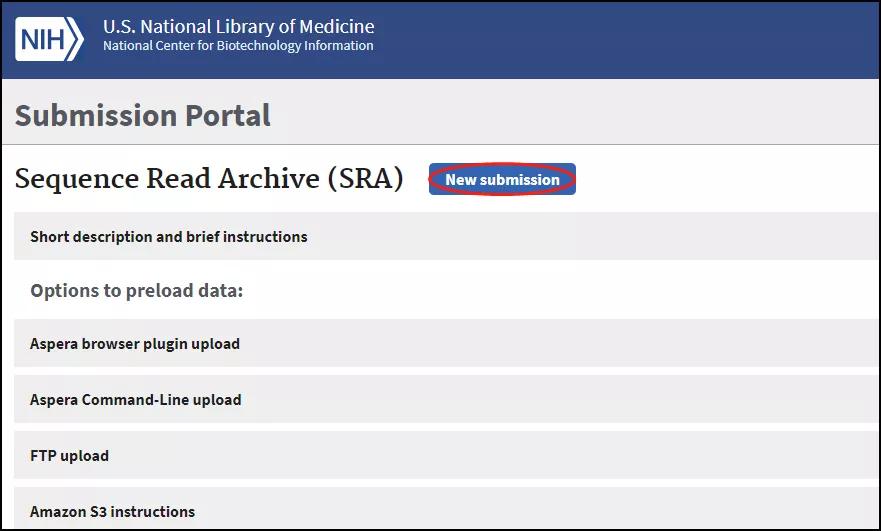
https://submit.ncbi.nlm.nih.gov/subs/,点击Sequence Read Archive:

页面跳转后点击New submission:
4.1 SUBMITTER
进入创建SRA页面Submitter选项卡,填写/完善必要个人信息:
4.2 GENERAL
在General选项卡里,选择前面创建的BioProject,是否已经创建好了BioSample选择Yes,发布日期根据实际情况选择:
4.3 METADATA
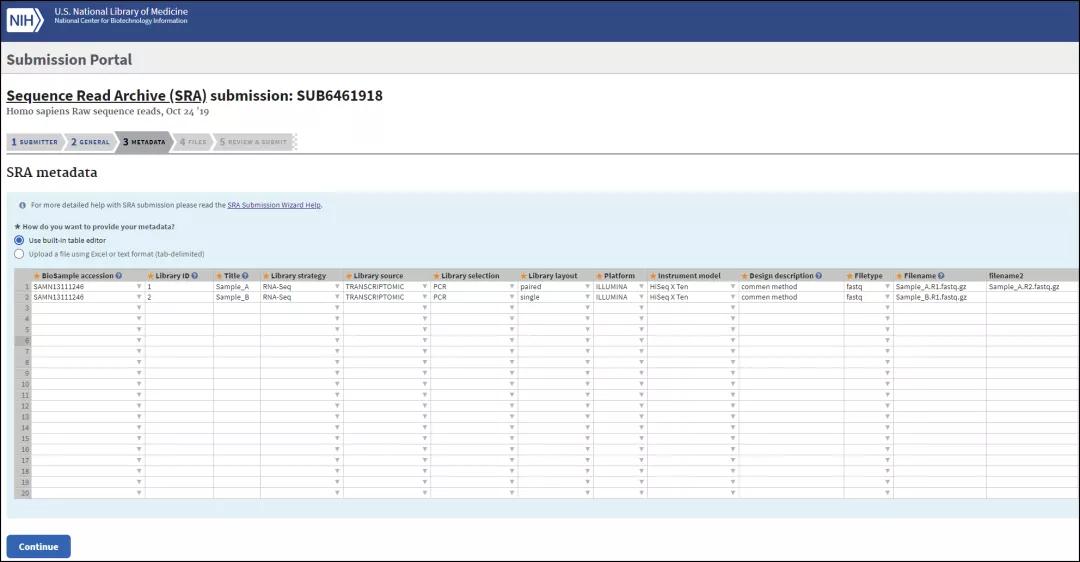
在Metadata选项卡,填写提交数据的相关实验信息,这一步可以直接在网页上填写,示例如下:


也可以下载模板表格填好之后再上传:
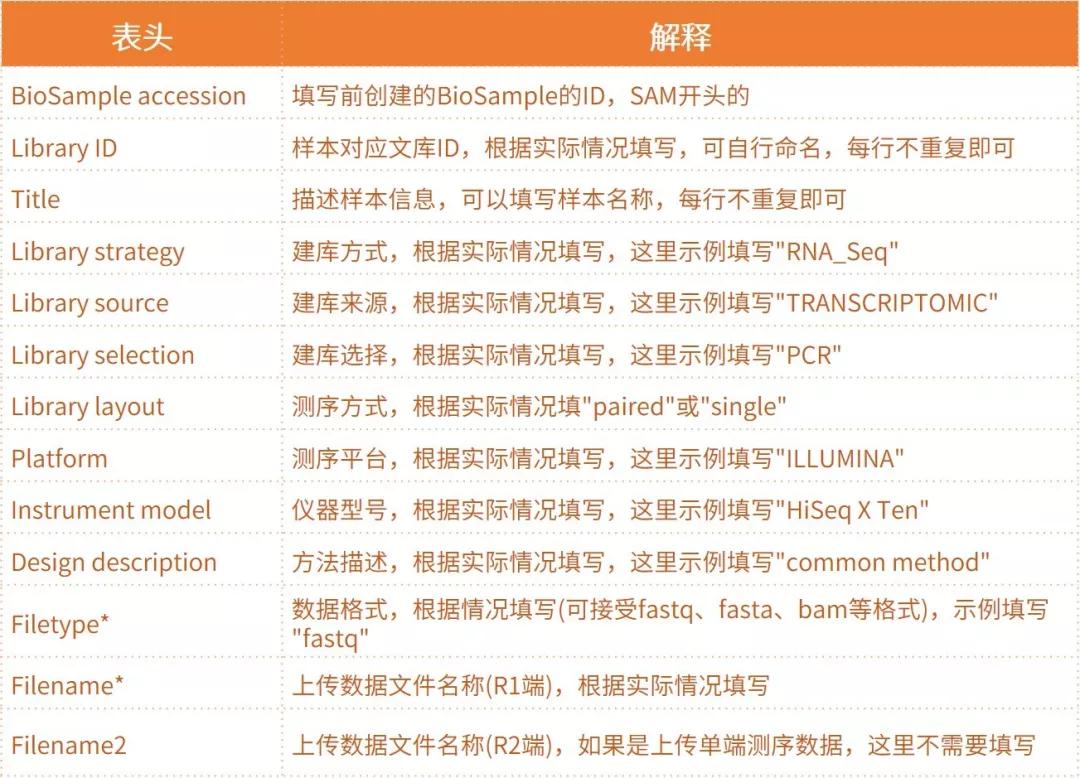
*Filetype如果只有fa文件也可以选择fastq格式(NCBI会默认上传的fa文件碱基质量值为30)
*Filename填写的文件名称必须和上传的序列文件名称一致(后台通过表格信息关联上传的数据,如果名称不一致会提示文件缺失),如“Filename”、“Flename2”分别填写了Sample_A.R1.fastq.gz、Sample_A.R2.fastq.gz,那么该样本的R1、R2端数据的文件名也必须是Sample_A.R1.fastq.gz、Sample_A.R2.fastq.gz。
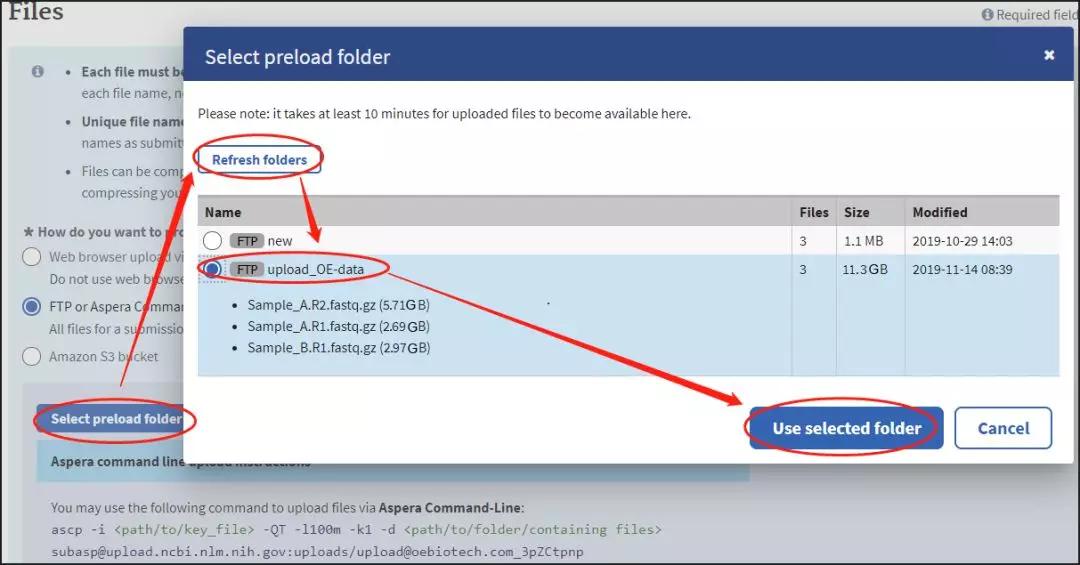
4.4 FILES
在Files选项卡里,对于上传数据的方式,我们通常选择FTP or Aspera Command Line file preload:即使用FTP工具(Filezilla)或命令行传输(ascp):
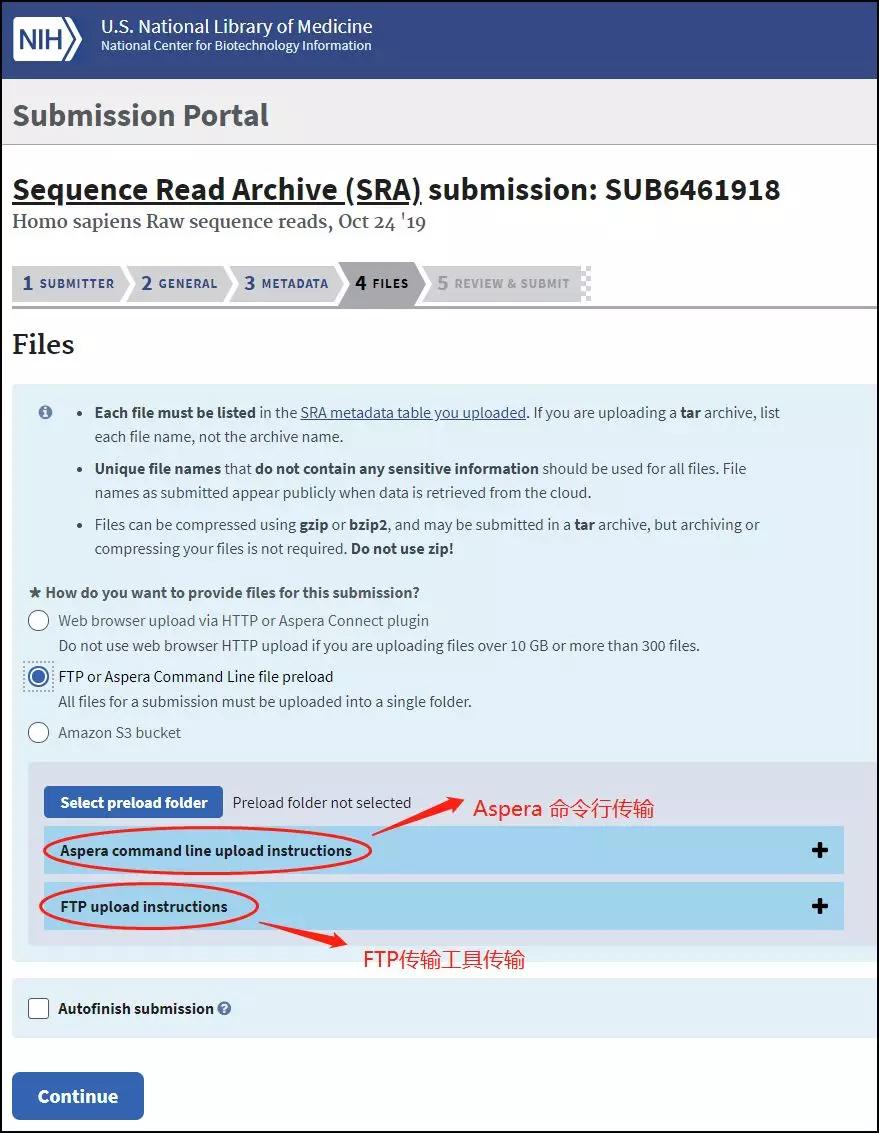
4.4.1 FTP传输-Filezilla
下载Filezilla(https://www.filezilla.cn/download/) 安装好之后并做如下设置:
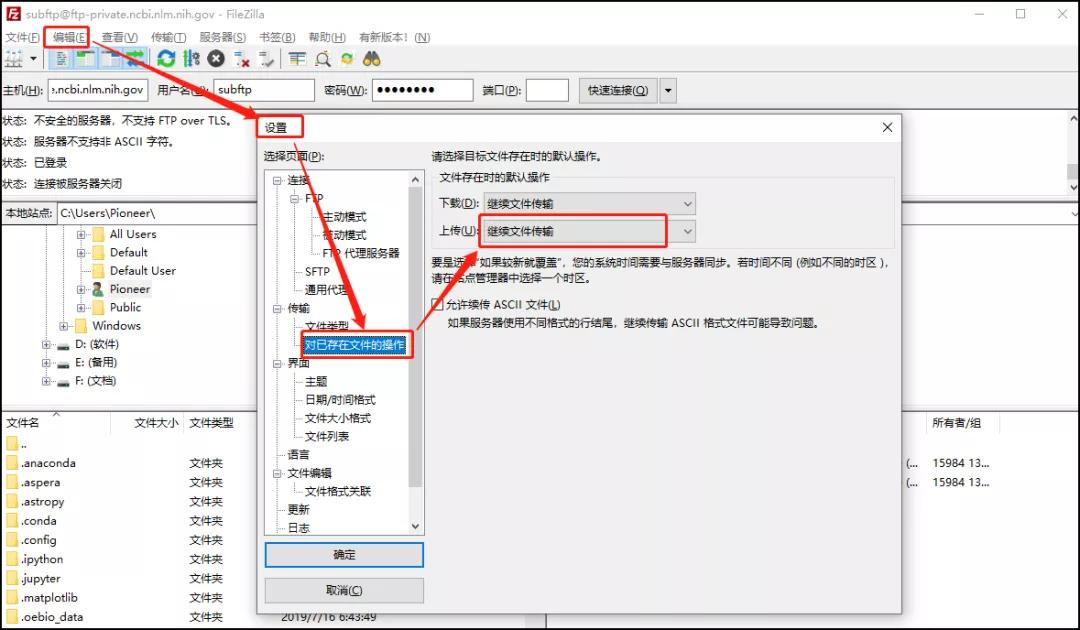
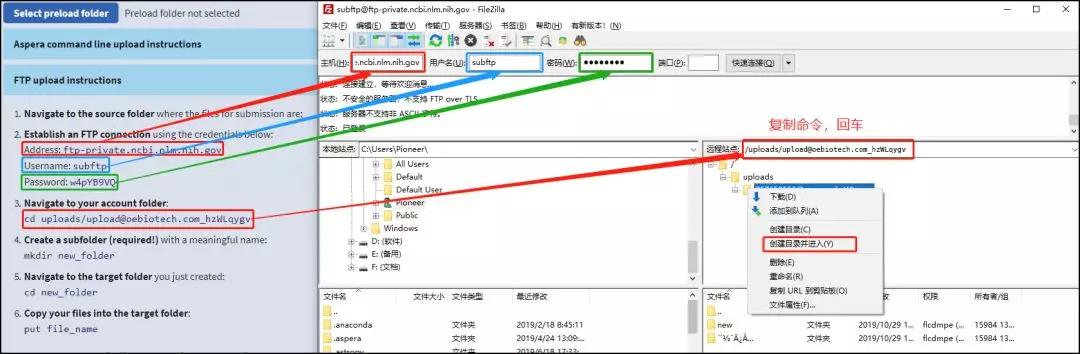
根据网页提供的FTP地址、账号、密码用Filezilla连接上服务器。如果一直读取目录失败,可以直接将3. Navigate to
your account
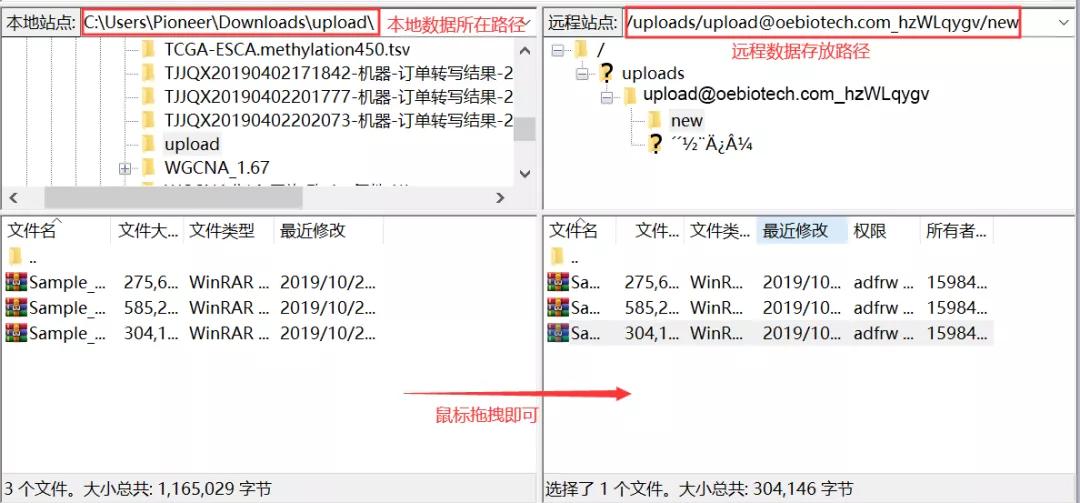
folder下面的目录(如“uploads/[email protected]_hzWLqygv”)复制进Filezilla的远程站点里(注意路径要以斜杠“/”开头),然后回车键就能进入目录了,进入目录后再创建一个子目录并进入(如果上传的数据不在新创建的子目录中上传的文件不会被检测到!):
将数据文件直接拖进右侧区域(新创建的子目录)等待上传完毕:
4.4.2 命令行传输-Aspera 上传/ascp
往SRA上传输数据的时候,有时会遇到Filezilla传输慢的情况,这时候就需要Aspera软件帮忙了。
软件官网下载链接:
https://downloads.asperasoft.com/en/downloads/62
网盘下载链接:
Windows:
https://pan.baidu.com/s/1OKgPKs2nEUH1ubuxAhXH8A 提取码: yw24
Mac OS:
https://pan.baidu.com/s/18oQ36MdNuMTm6OGUVJQJuw 提取码: gy55
下载好解压缩即可,解压缩的路径一会要用到,例如D:\Aspera\cli\bin
Aspera环境变量设置(以windows为例):右键我的电脑-属性-高级设置-环境变量设置-PATH里添加软件的路径(D:\Aspera\cli\bin)
windows 键+R 弹出运行框,输入cmd 回车进行进入cmd界面,输入ascp –h可正常显示 :
在Aspera command line upload instructions ,可以查看到命令行:
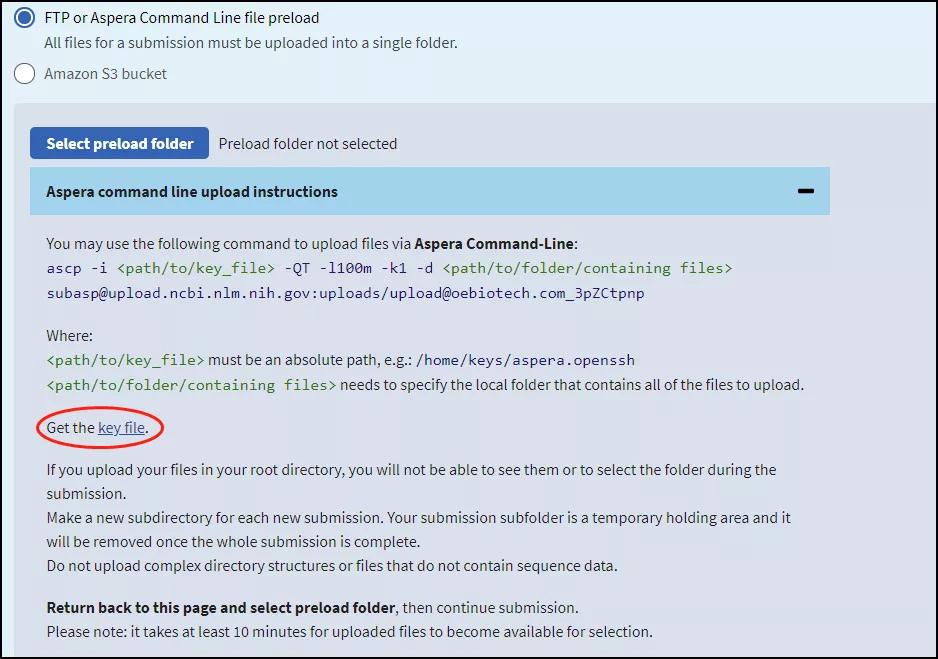
注意命令行中提及的key_file(上图红圈部分)点击下载key文件aspera.openssh并存放在指定位置,如 "D:\Aspera";数据上传完整命令如下:
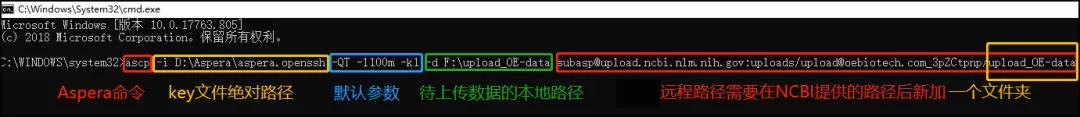
ascp -i D:\Aspera\aspera.openssh -QT -l100m -k1 -d F:\upload_OE-data
[email protected]:uploads/[email protected]_3pZCtpnp/upload_OE-data
-i 之后填写前面下载的aspera.openssh文件的绝对路径("D:\Aspera\aspera.openssh")
-d 之后填写需要上传的数据文件的路径,这个路径下除了待上传的原始数据最好不要存放其他文件("F:\upload_OE-data")
"空格"之后接NCBI的远程路径
("[email protected]:uploads/[email protected]_3pZCtpnp/upload_OE-data")
远程路径后面需要添加一个子目录("upload_OE-data"),否则上传的数据检测不到!
命令输入后回车显示传输,等待提示传输完毕,传输中断可重复之前的命令。
数据上传成功之后(这一过程消耗的时间根据文件数量、大小及网速决定)做如下操作:
确认没问题,点击继续:
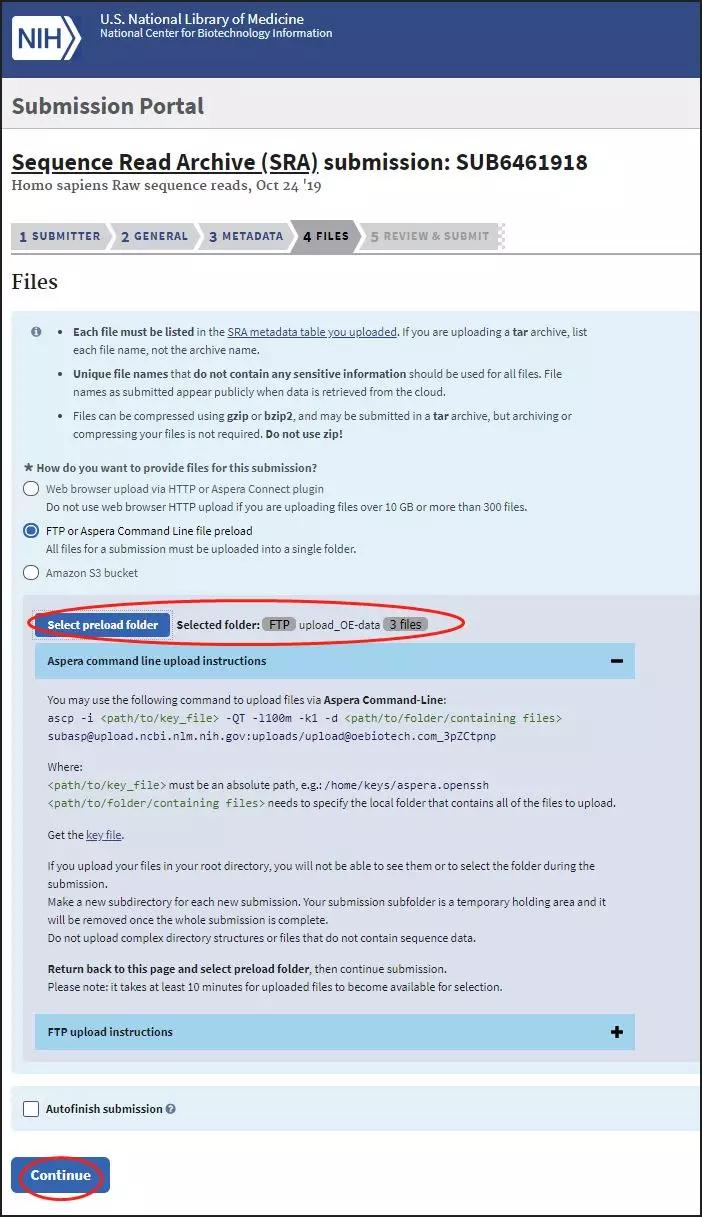
4.5 OVERVIEW
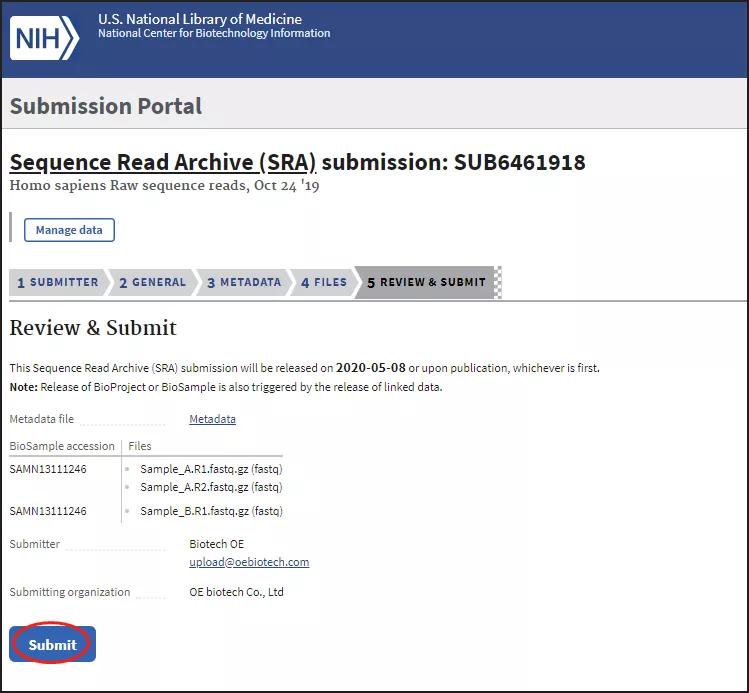
最后Overview中浏览概况,确认无误后点击Submit提交:
05
完成上传
可以在
https://submit.ncbi.nlm.nih.gov/subs/sra/查看审核状态:
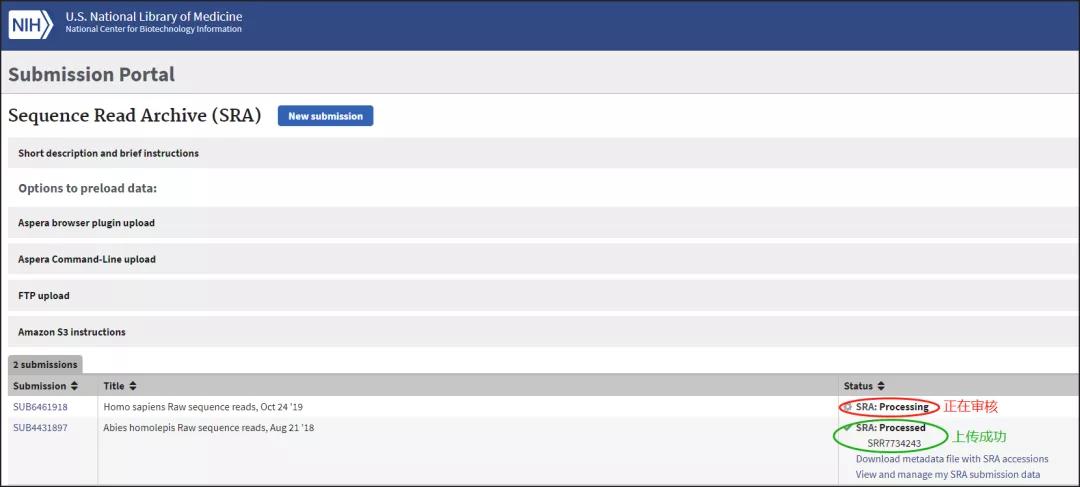
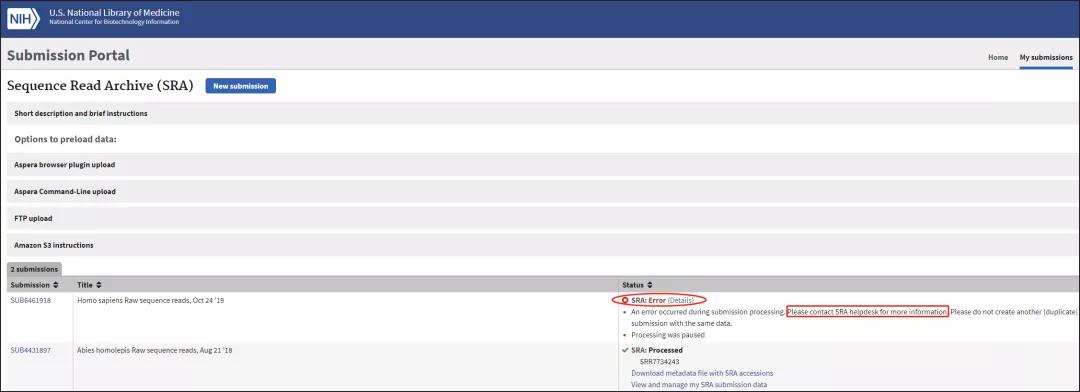
目前测序数据上传完成。NCBI会在后台审核数据,最后给出Accession Number,一般一天左右。
审核完毕的数据可能不会马上被NCBI收录,一般需要2-4天才能被搜索到(如果设定了发布时间,则在发布时间之后才能搜到)。可以在SRA的Submission中(https://www.ncbi.nlm.nih.gov/Traces/sra_sub/?login=pda)查询到目前的审核进度。
注意最终使用的Accession Number一般为Run的编号,SRR开头。可以点击Submission Id进入查看Accession Number:
Tips:上传过程中遇到任何问题(如下图的报错信息)可以直接邮件联系NCBI请求帮助(邮箱:[email protected])
至此,整个数据上传就已经全部完成了;Accession Number放进文章,完成文章接收的最后一步。
SRA数据上传的界面会时有更新,我们也会定期跟踪,及时更新,确保本指南的实用性。
本文系欧易生物原创