人类基因组计划自启动以来,目前已经出了很多版本的人类基因组,例如hg18, hg19, GRCh37,
GRCh38。为了将不同版本的基因组位置信息一一对应,UCSC
推出了一个基于基因组序列的转换工具LiftOver。它能够快速准确的将不同版本的基因组坐标进行相互转换,已被广泛应用于各种基因组研究之中。UCSC
分别提供了网页版和Linux命令行两个版本的LiftOver工具。网页版工具的输入文件大小限制为500M,当遇到比较大的文件时,需要使用Linux命令行版本进行转换。本文将通过实际例子介绍如何通过LiftOver两个版本的工具进行基因组坐标的转换。

1
网页版LiftOver使用介绍
进入UCSC主页(http://genome.ucsc.edu/),点击菜单栏上的Tools -> LiftOver进入LiftOver页面

其中主要的参数说明如下:
Original Genome: 原始的物种基因组,有很多物种(human, mouse, dog等),这里选择Human;
Original Assembly: 原始物种的基因组版本,根据需求选择,这里选择CRCh37/hg19;
New Genome: 新的要转换的物种基因组,根据需求选择,这里选择Human;
New Assembly: 新的要转换物种的基因组版本,根据需求选择,这里选择GRCh38/hg38;
输入文件:bed文件格式,可以复制粘贴输入,也可以上传文件进行输入,这里输入测试的hg19的bed坐标文件
待所有参数和数据准备完成,点击Submit/Submit File提交,进行转换。示例结果如下:
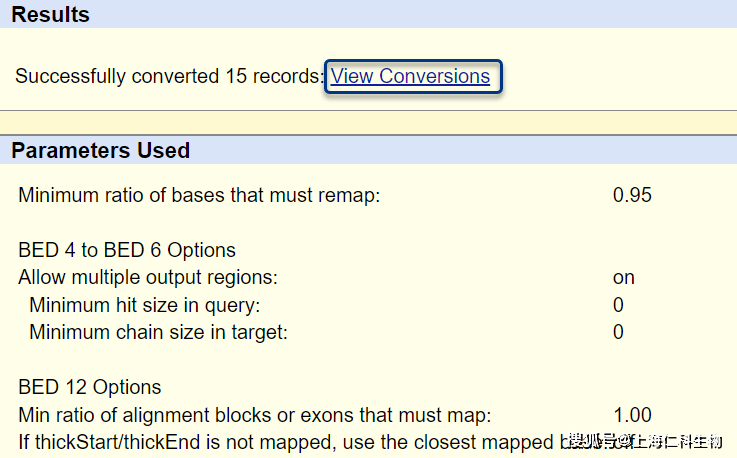
点击View Conversions下载结果,就得到了转换后的hg38基因组的坐标文件。

2
命令行版本LiftOver使用介绍
进入http://hgdownload.soe.ucsc.edu/admin/exe/下载页面,根据自己的系统选择对应版本的工具文件进行下载,这里以linux.x86_64的版本为例。
同时,根据下面的链接,按照不同基因组版本下载转换需要的map.chain注释文件https://hgdownload.soe.ucsc.edu/goldenPath/hg19/liftOver/hg19ToHg38.over.chain.gz
https://hgdownload.soe.ucsc.edu/goldenPath/hg38/liftOver/hg38ToHg19.over.chain.gz
在Linux服务器下面,参考下面的命令行进行操作。
#命令行
liftOver test_file.bed hg19ToHg38.over.chain test_file_hg38.bed test_file_unmapped.bed
#test_file.bed:输入的bed文件
#test_file_hg38.bed:转换成功的bed坐标文件
#test_file_unmapped.bed:转换失败的bed坐标文件
至此,我们已经掌握了如何利用网页版和命令行LiftOver工具进行不同版本基因组坐标的转换,在实际应用中,用户可以根据自己的习惯进行选择网页版和命令行版本进行操作。下期,我们将介绍如何利用UCSC Table Browser提取dbSNP rs号在基因组上的坐标信息。