从小白的角度,一刻钟复现生信套路
各位小伙伴大家好,这里是美丽专栏。前面两期,我们复现了两篇非肿瘤生信文献,今天,我们继续开展非肿瘤文章的复现。话不多说,开始今天的复现之旅吧。
往期传送门
TCGA+GEO单基因生信SCI,15分钟手把手带你复现(内附详细操作教程)
最简配的2区生信SCI长啥样?360度无死角让你彻底搞懂,复现!思路原理都在这里了!建议收藏!(附详细操作过程) 别不信!近4分的非肿瘤套路,我用一刻钟零代码就可以复现!瞅一眼就会了(附详细操作教程)
慈母级生信分析教程!按这个方法搞,一晚上肝一篇生信文章!(附超详细复现教程)
文章复现是生信小白成长为大神的最佳路径。在本篇文章中,美丽将会手把手教您6图9表逐个步骤的文章复现。
今天为大家带来一篇2020年3月份发表于Autoimmunity(影响因子:2.125)的非肿瘤生信文章套路复现。
话不多说,我们开始吧!
题目
Deciphering crucial genes in coeliac disease by bioinformatics analysis
.

材料与方法一:患者数据收集情况


材料与方法二:图表结果及复现
01
使用工具
CTD数据库(http://ctdbase.org/)
Genecards数据库(https://www.genecards.org/)
DISEASE数据库(https://diseases.jensenlab.org/Search)
仙桃工具(https://www.xiantao.love)
String(https://www.string-db.org/)
WebGestalt网站(http://www.webgestalt.org/)
GeneMANIA网站(http://genemania.org/)
02
复现任务
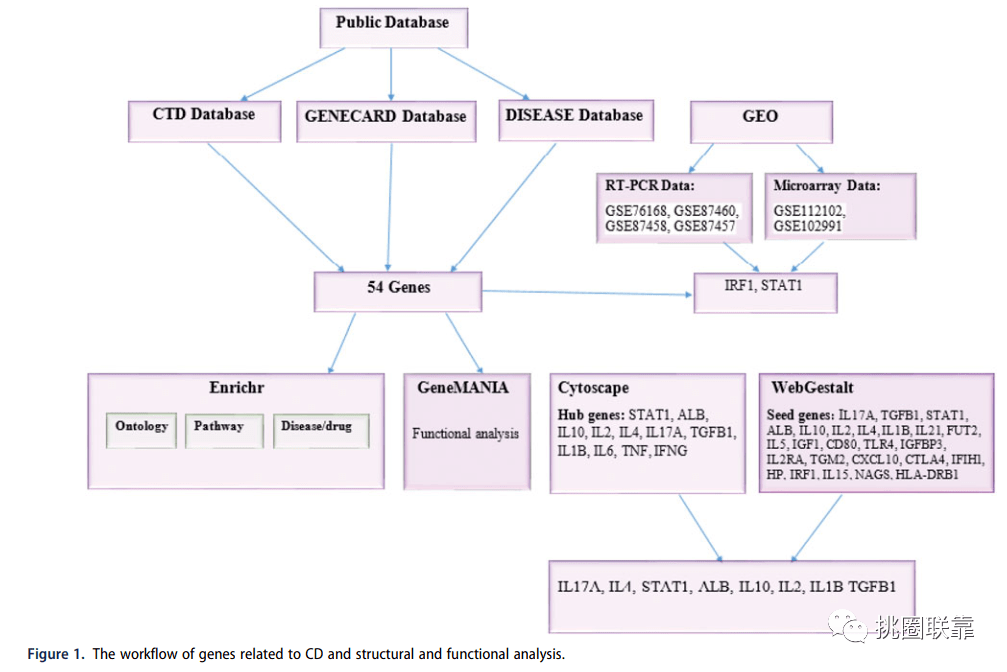
图1 技术路线图
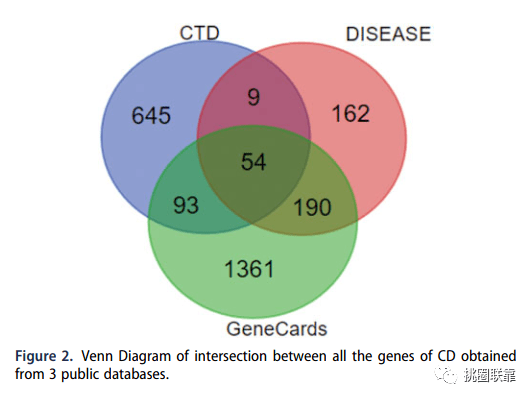
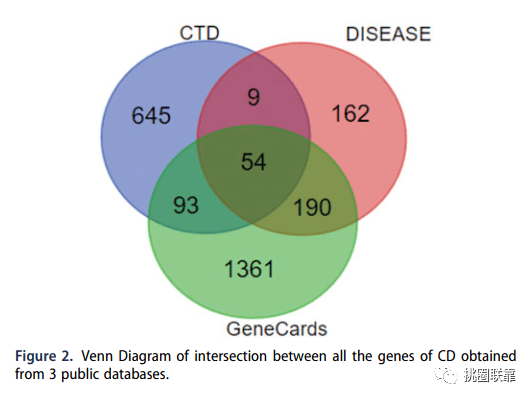
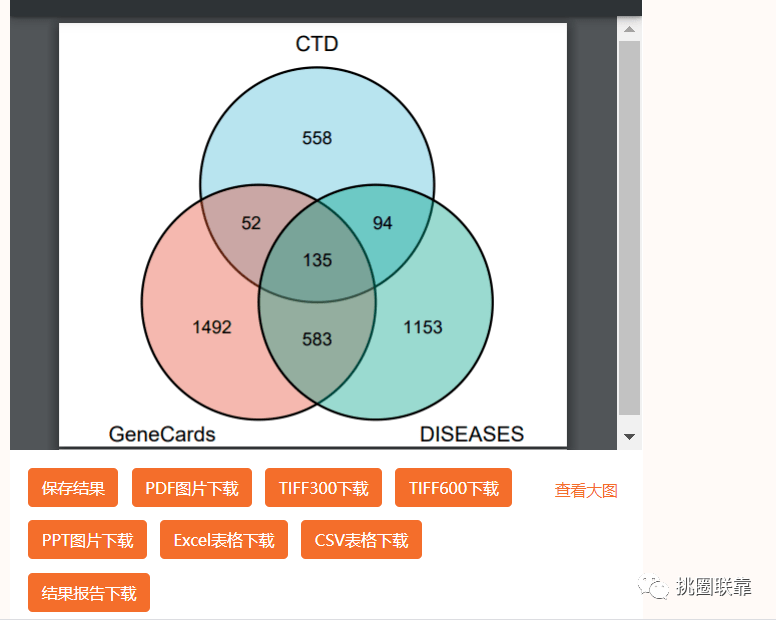
图2 三组数据库的疾病韦恩图
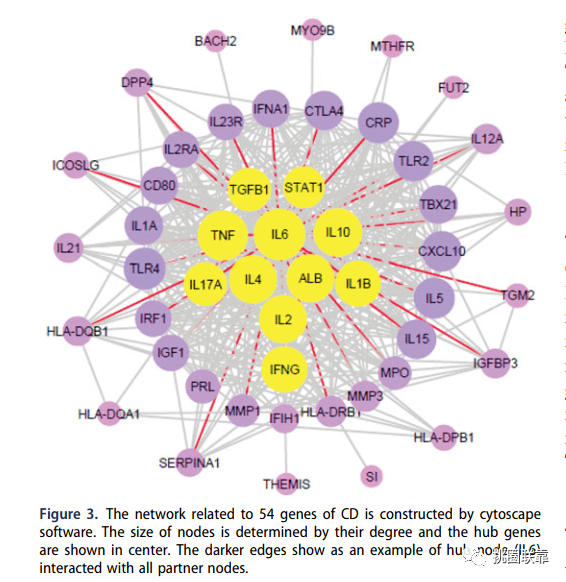
图3 差异基因的蛋白互作网络
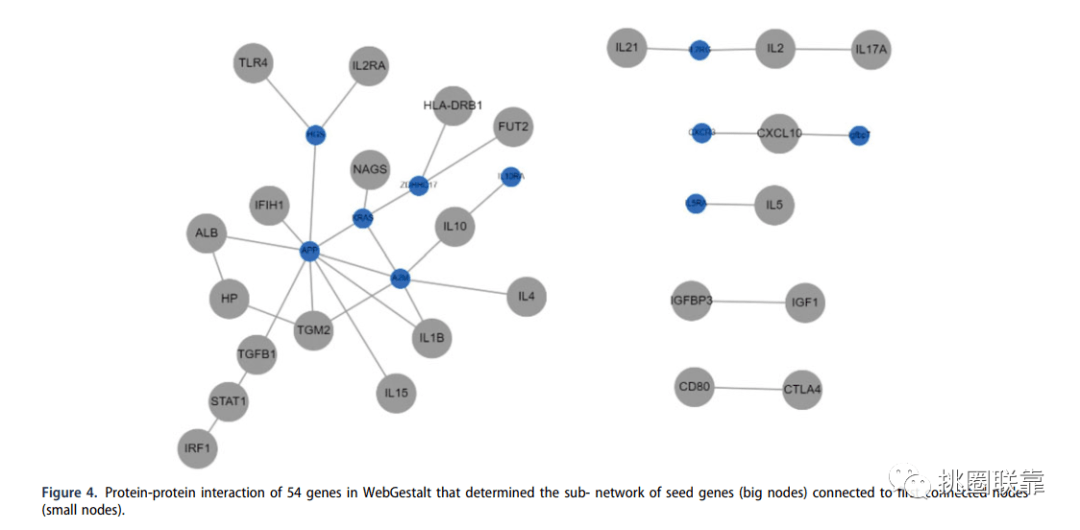
图4 差异基因的PPI分析②
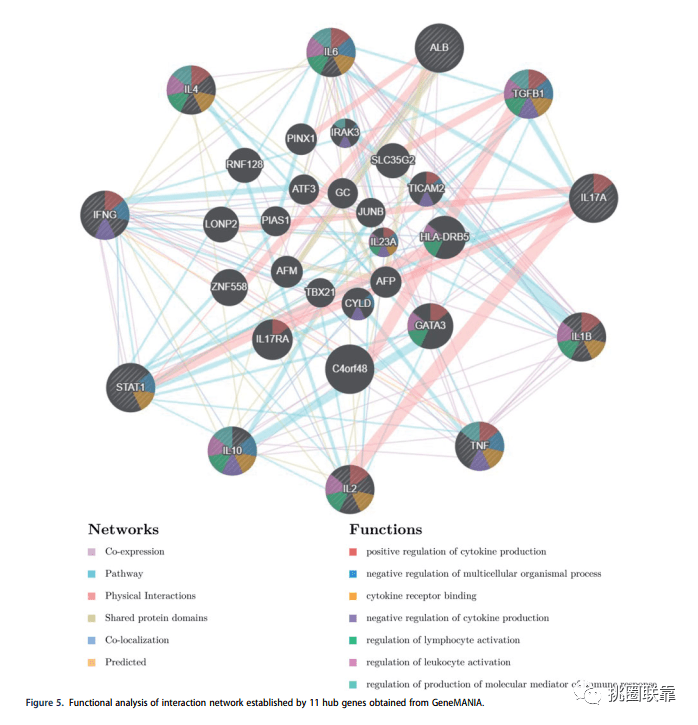
图5 hub基因的富集分析
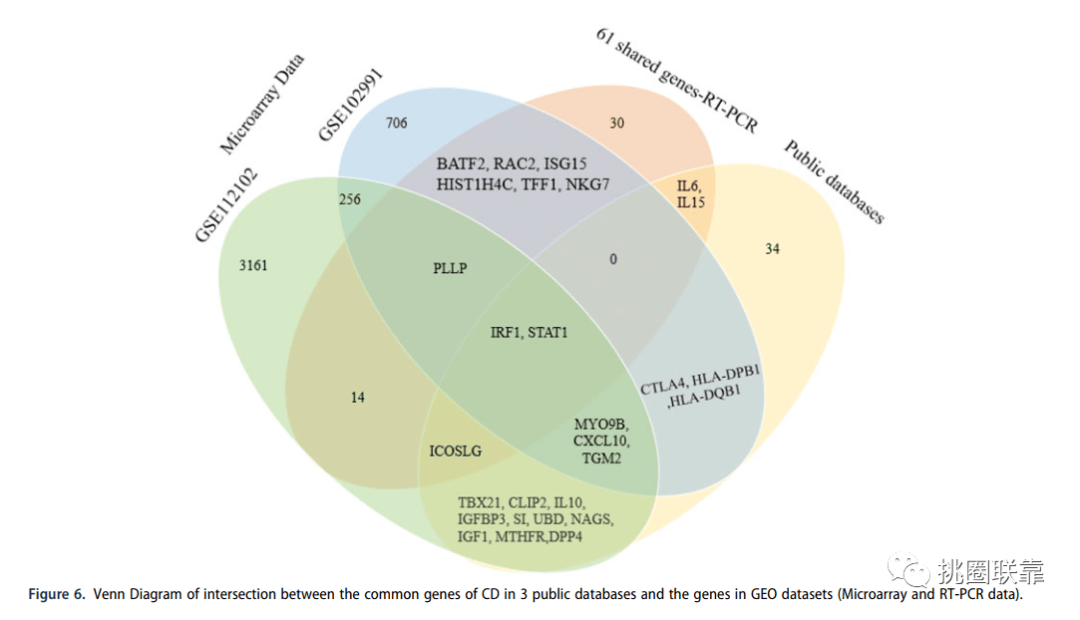
图6 多套数据集的共同表达基因韦恩图
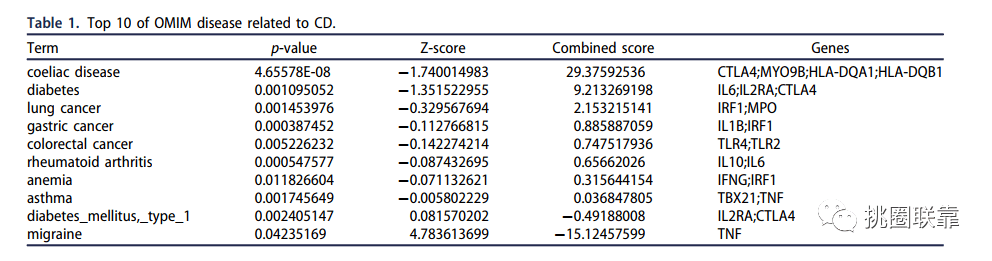
表1 在OMID中,CD相关的前十种疾病
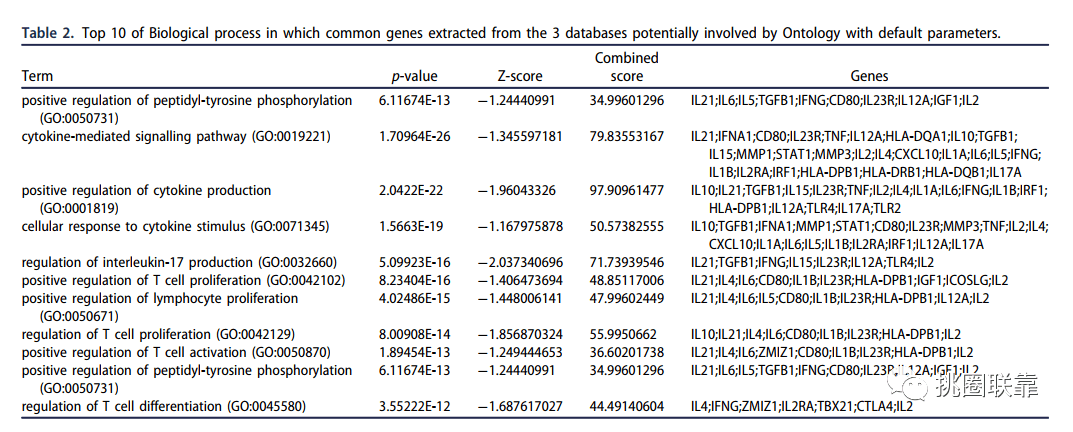
表2三种数据集的GO-BP通路结果
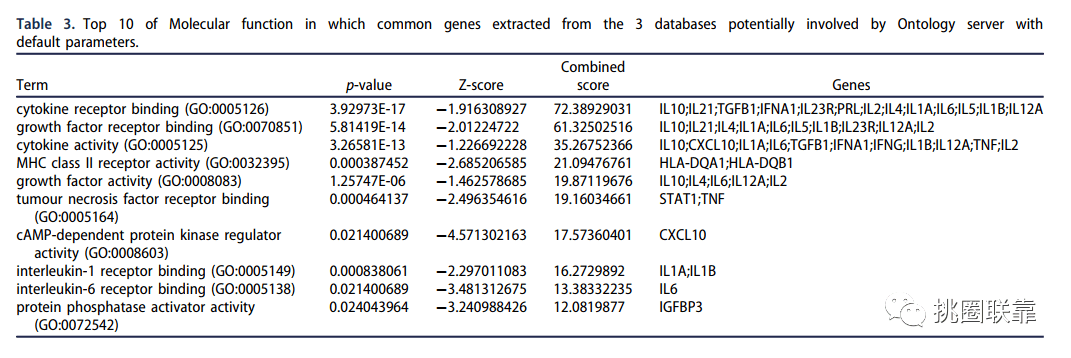
表3三种数据集的GO-MF通路结果
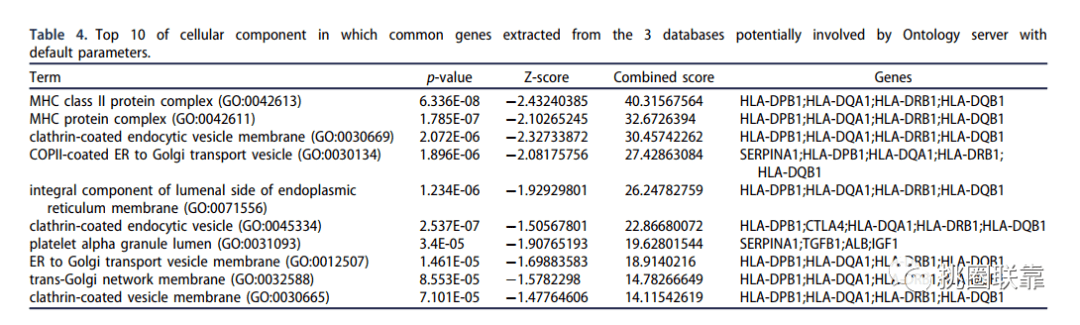
表4三种数据集的GO-CC通路结果
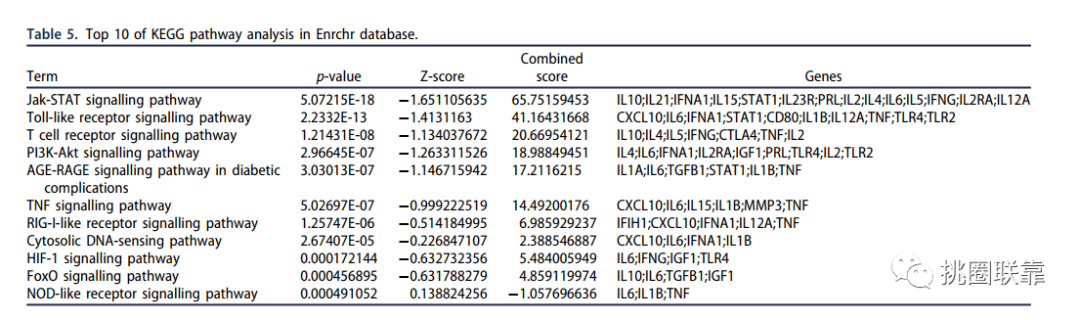
表5三种数据集的KEGG富集分析结果
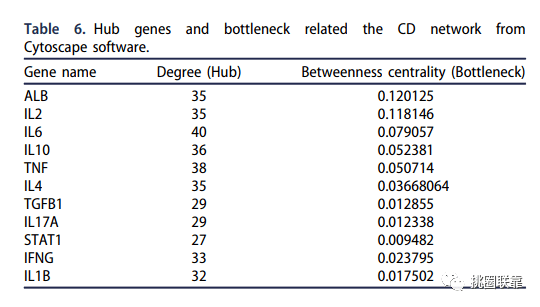
表6 hub基因信息
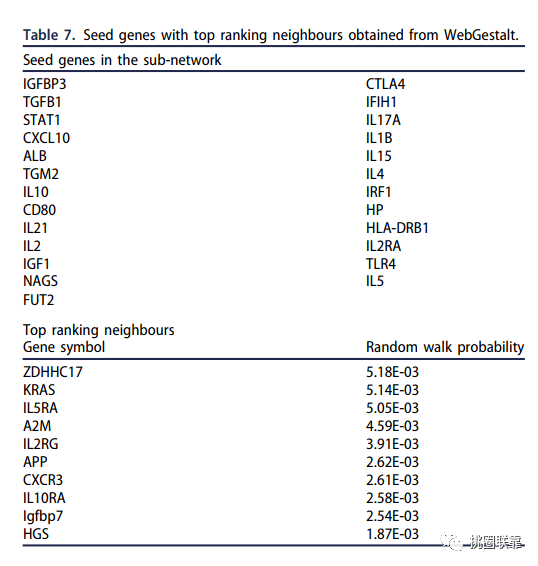
图7 WebGesalt获取的种子基因
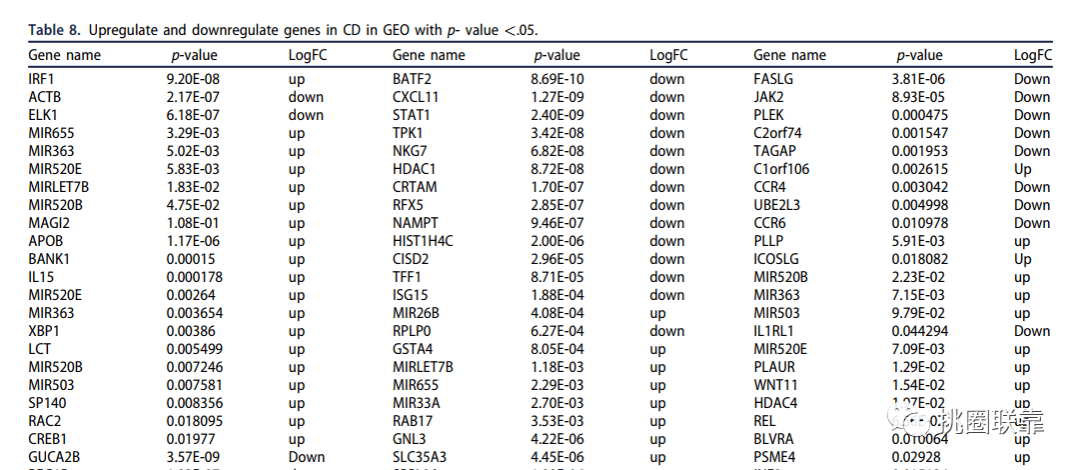
图8 GEO数据挖掘的上调、下调基因
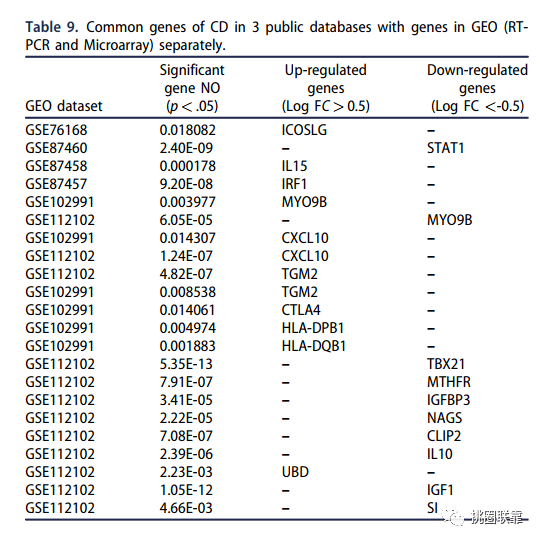
图9 GEO数据集的共同差异基因展示
03
复现步骤
复现任务1 图2 三组数据库的疾病韦恩图
数据准备
打开CTD数据库(http://ctdbase.org/ )
输入Celiac Disease
点击search
选择疾病
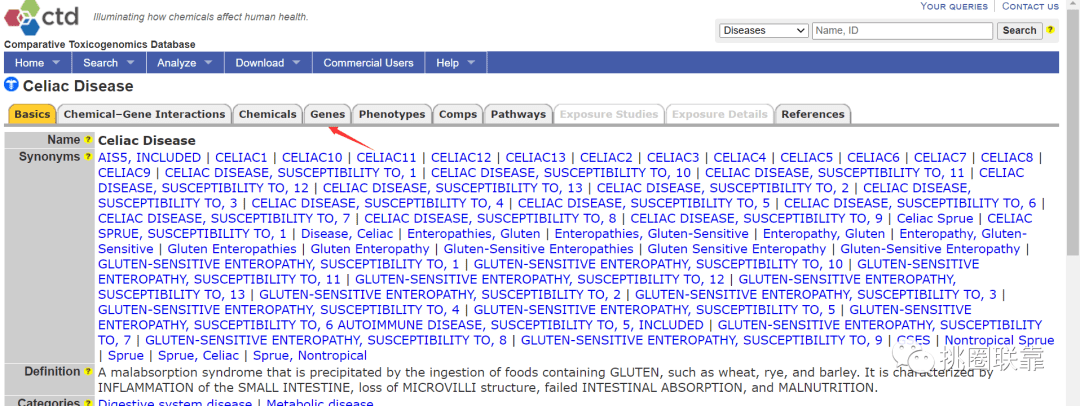
点击Genes
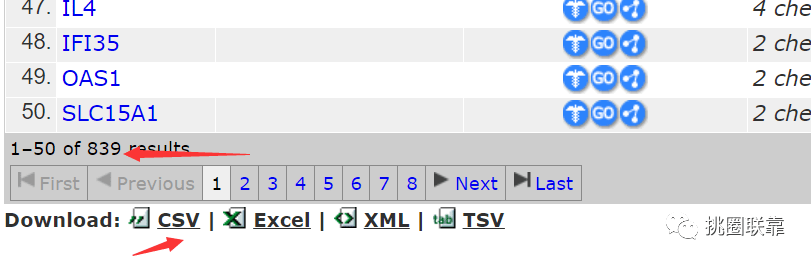
最下方有下载,可以选择不同的格式。即可获取CTD数据库中与CD疾病有关的基因信息。
打开Genecards数据库(https://www.genecards.org/)
输入Celiac Disease

选择全部导出
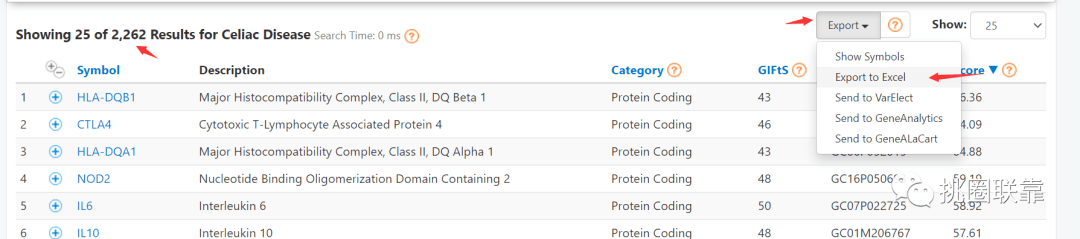
即可在Genecards数据库获取与疾病相关的基因集。
打开DISEASE数据库(https://diseases.jensenlab.org/Search)
输入Celiac Disease
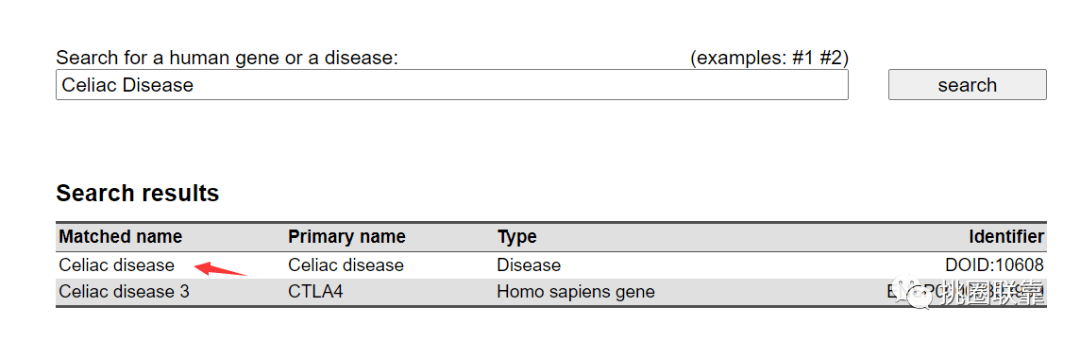
选择第一个疾病
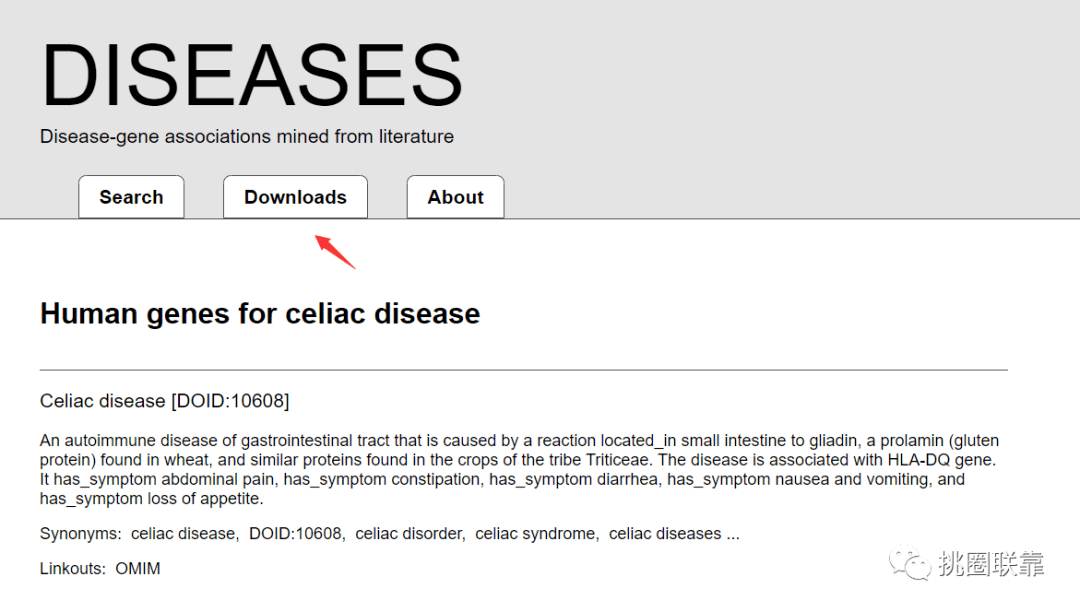
点击download下载
Tips:同时在图片下方,有链接,可以与OMIM数据库联合使用,绘制 表1。
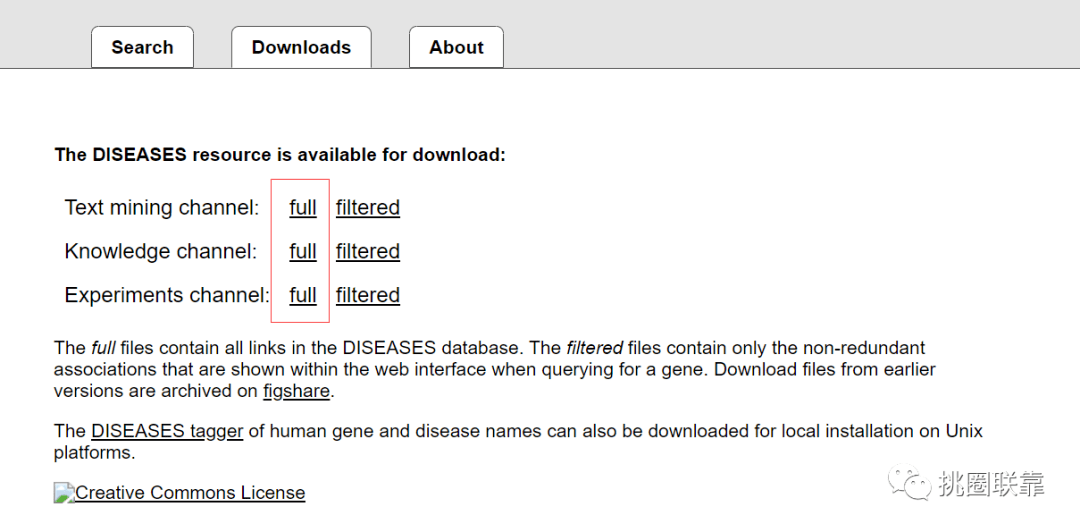
将三种格式的full文件全部下载。即可获取DISEASES数据库中与疾病相关的基因信息。
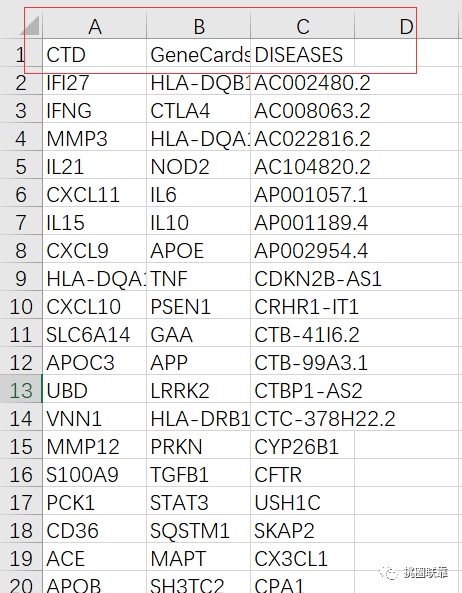
接下来,将上述三个数据库获得的基因进行归纳,整理成如上表的格式。
作图
打开仙桃工具(https://www.xiantao.love)
下载结果报告
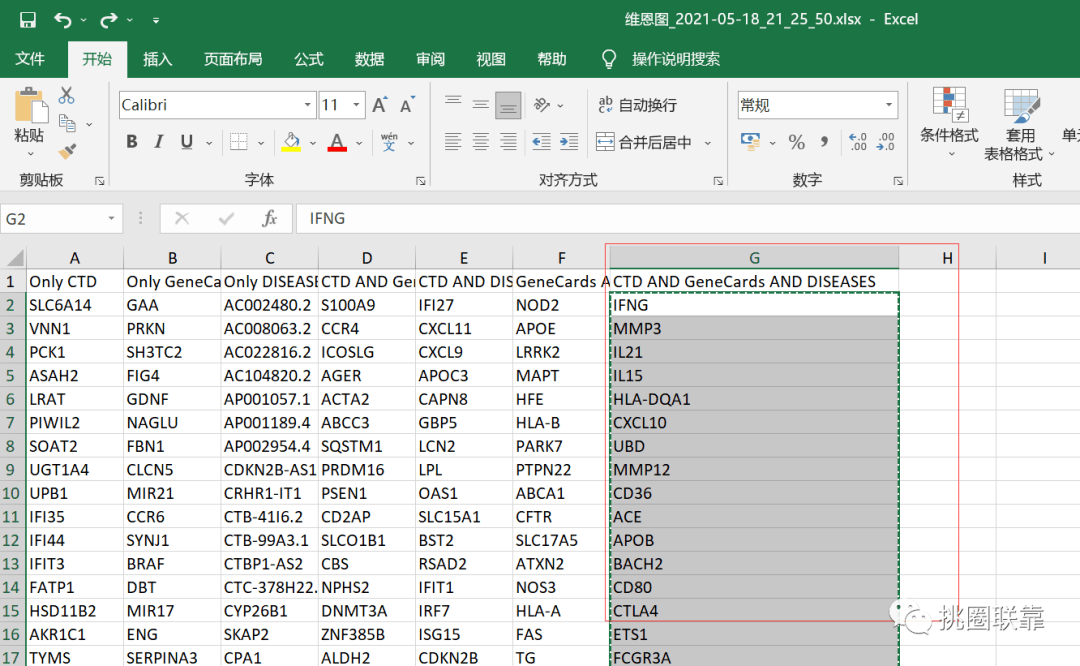
打开结果报告,可以看到三个数据库的共同基因名称。
复现任务2 图3 差异基因的蛋白互作网络
说明:我们复现的结果比原文多,这里,主要学习操作即可。不要纠结最后的结果为啥与作者不同。
准备工作
首先,进入string数据库,将基因输入,获取交互关系。
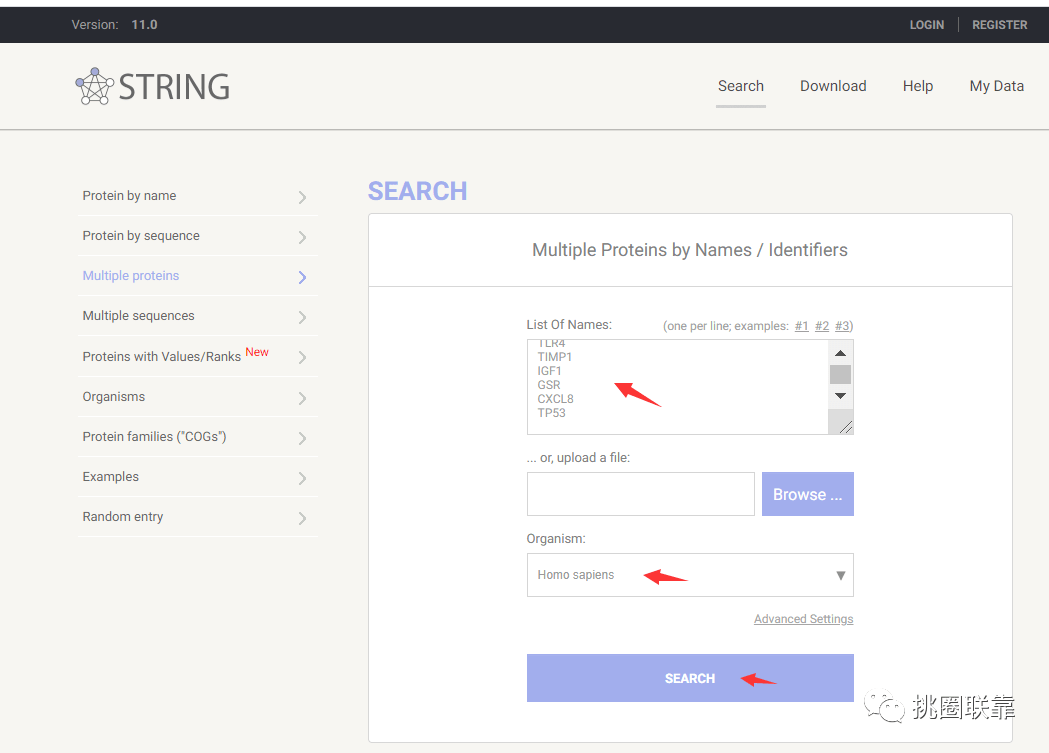
打开string数据库(https://string-db.org/cgi/input?sessionId=b2oQKsypYrfs&input_page_active_form=multiple_identifiers),将差异基因输入,选择物种,点击search。
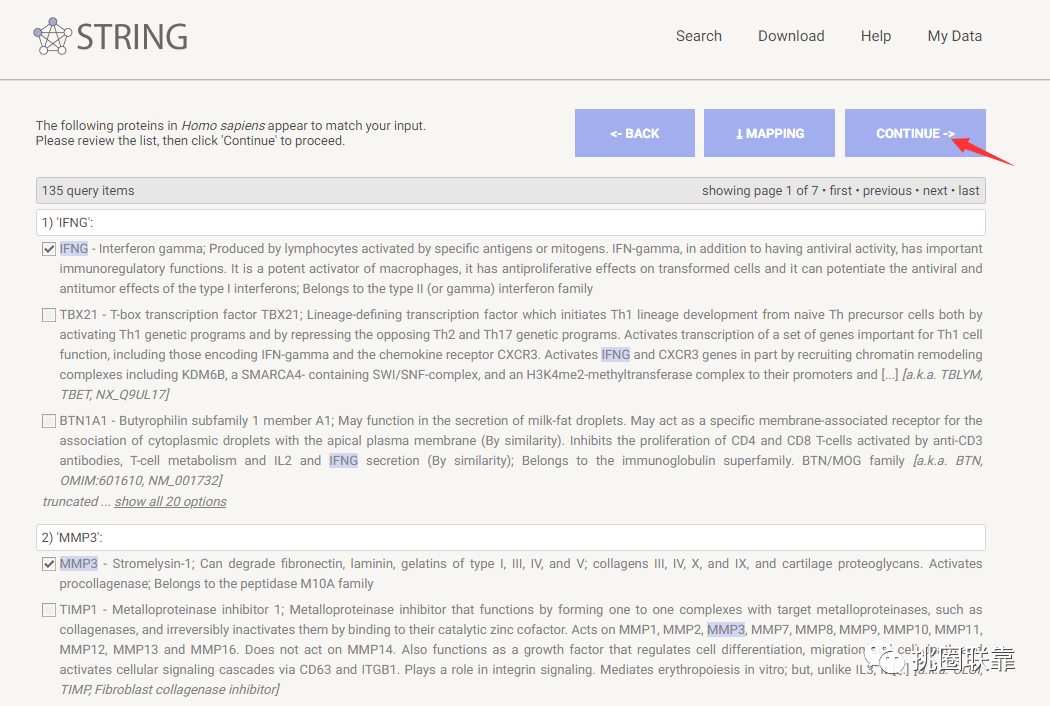
点击继续
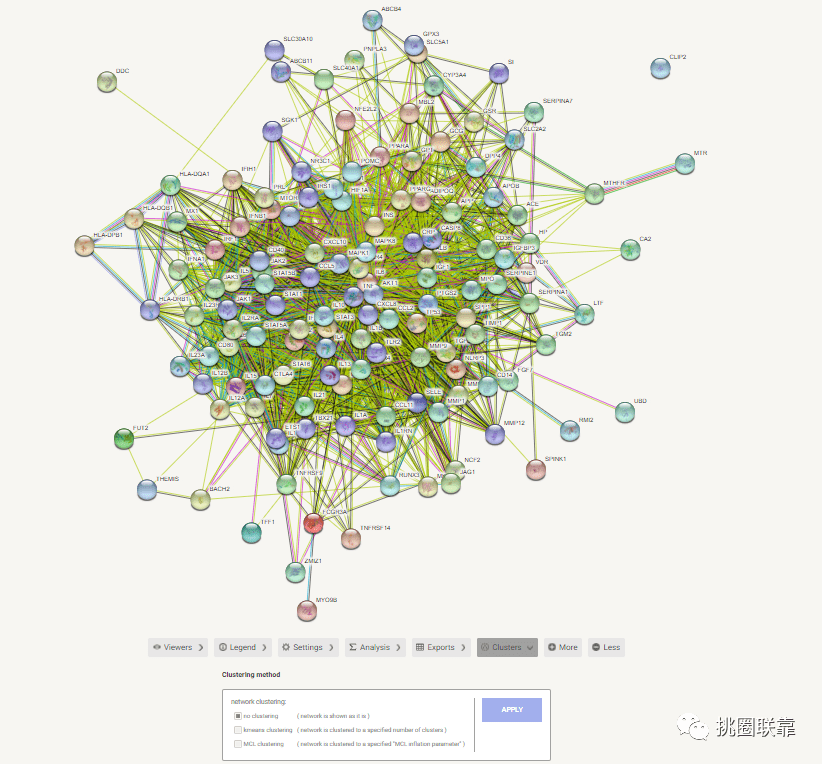
点击继续
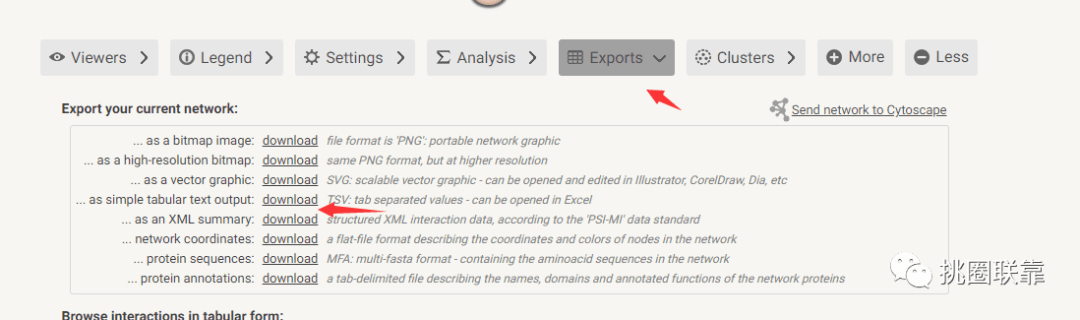
选择exports,下载tsv格式数据
作图
使用cytoscape进行PPI作图
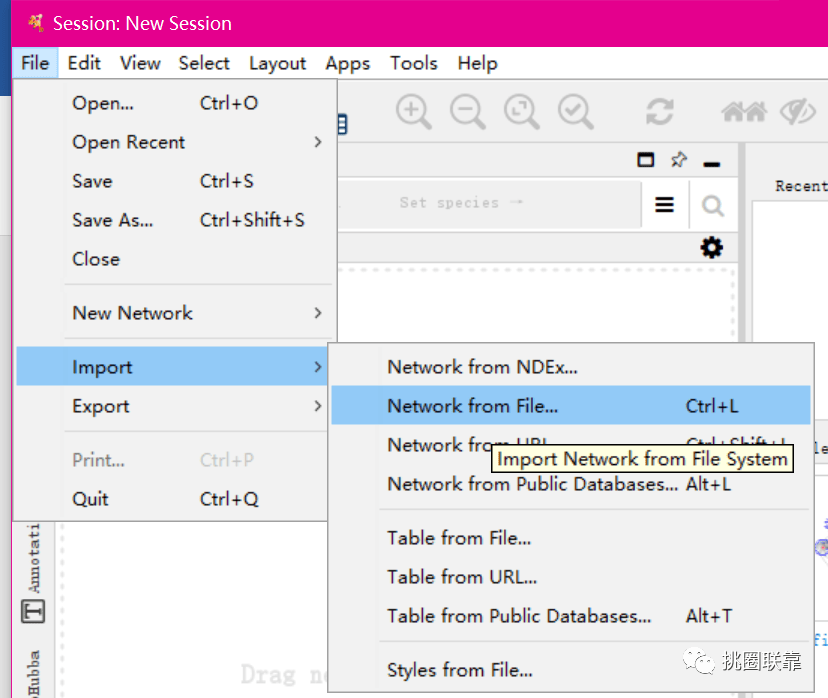
导入string分析好得网络文件
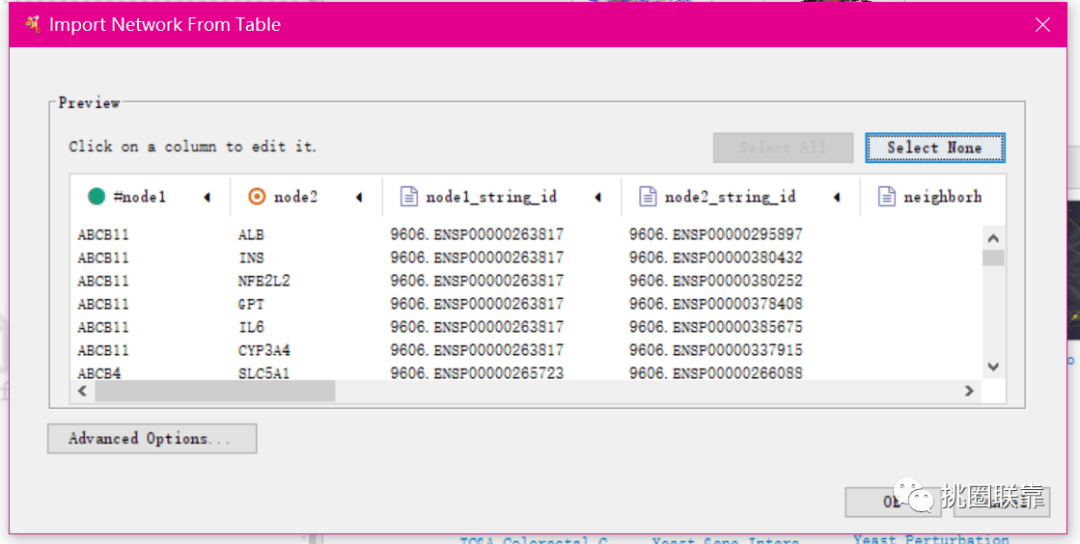
点击确定
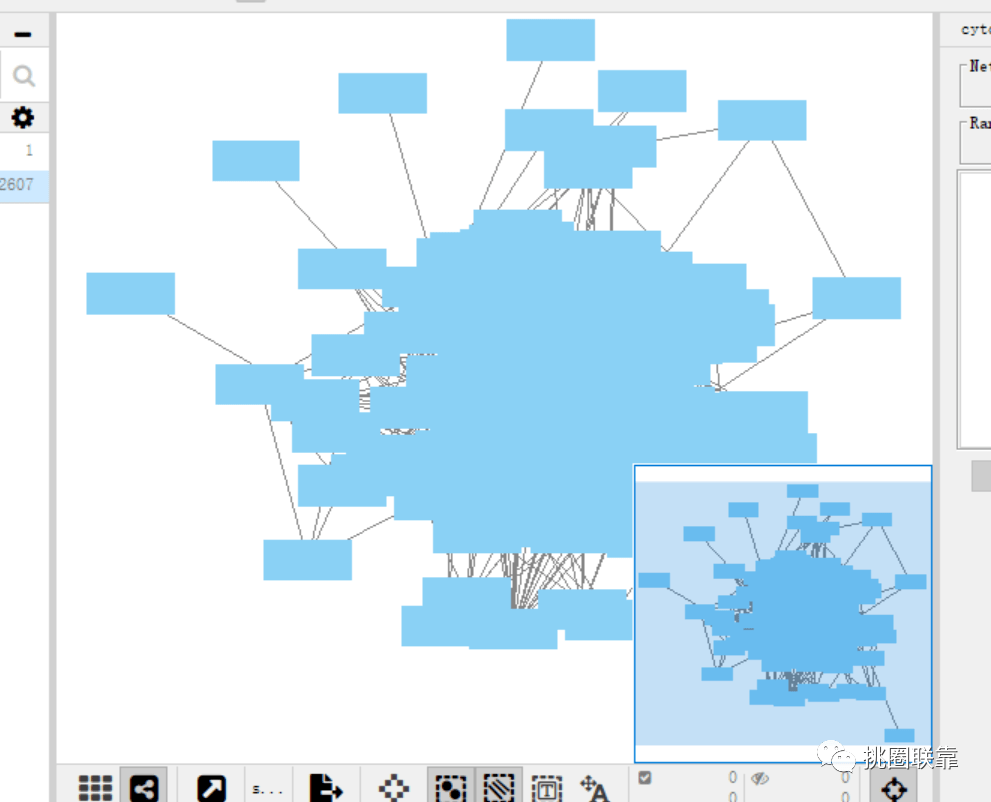
原始PPI图
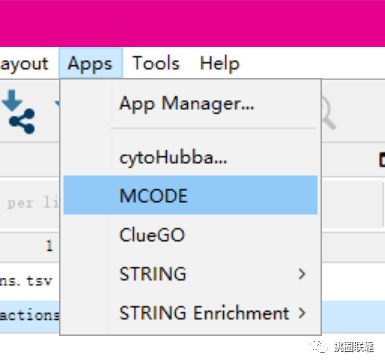
选择MCODE功能
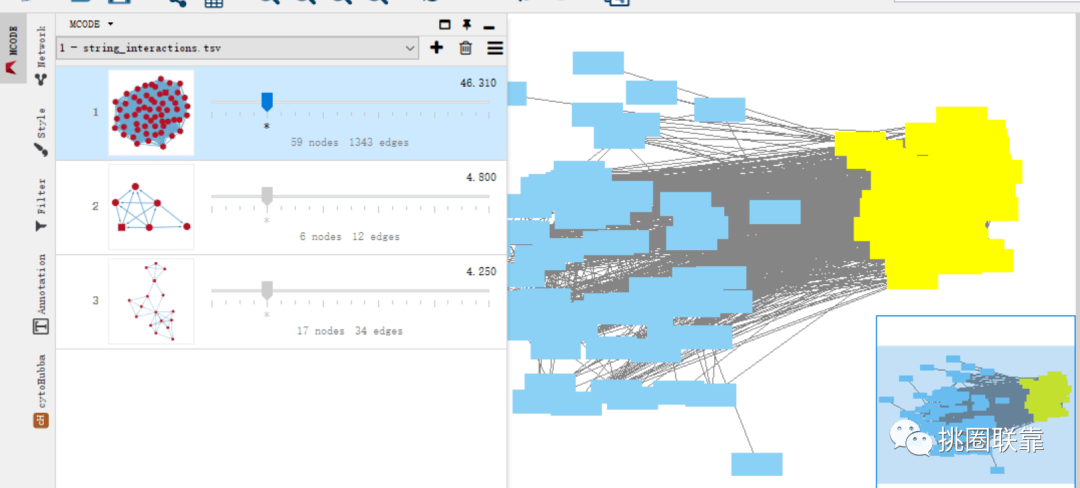
点击第一个MCODE计算模型
打开cytoHubba功能
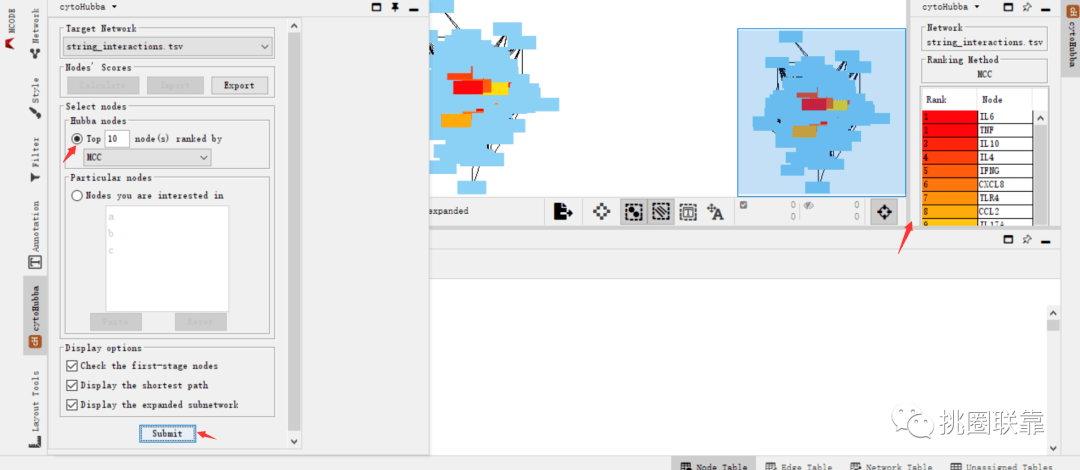
选择MCC法,提交
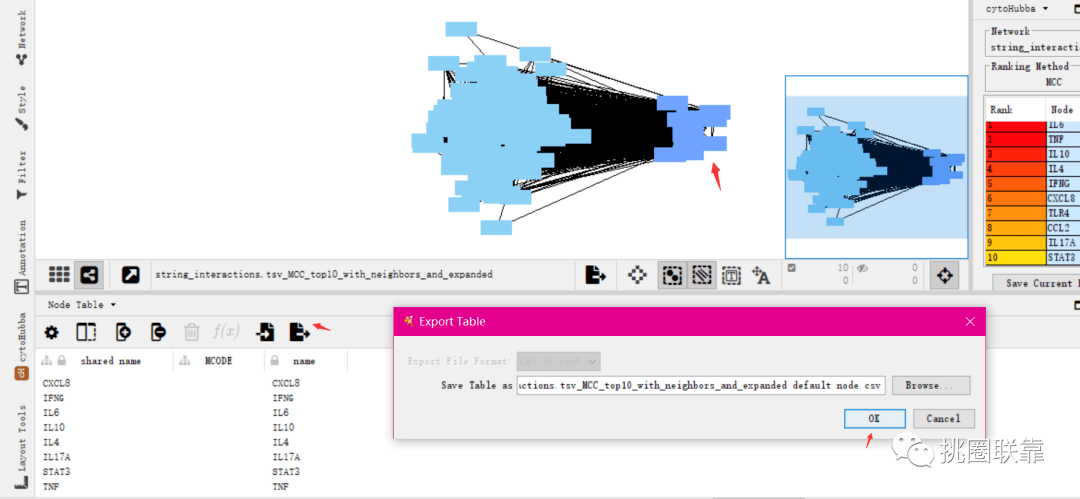
获取Top10 Hub基因,下载保存相关信息
(同时可制作表6)
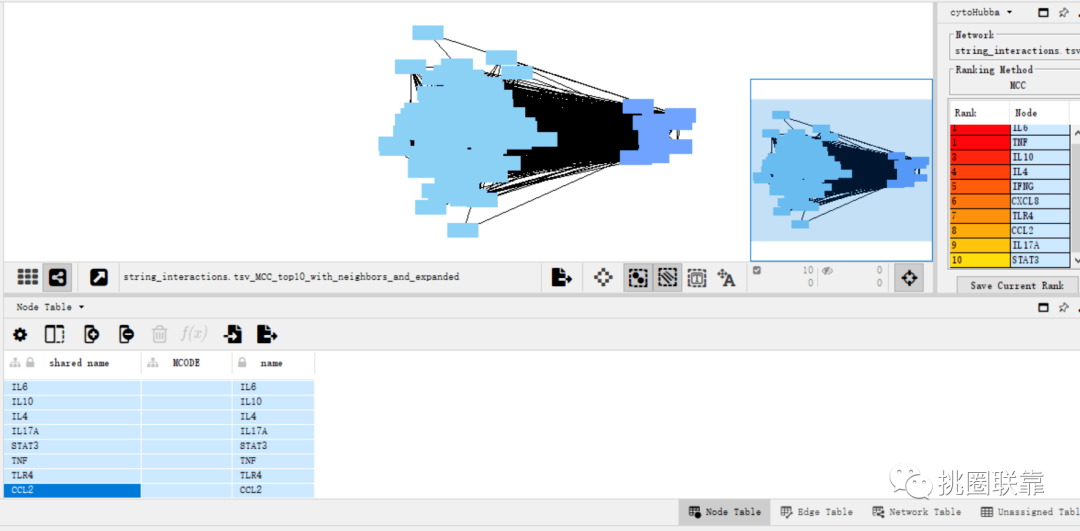
全选Hub 基因
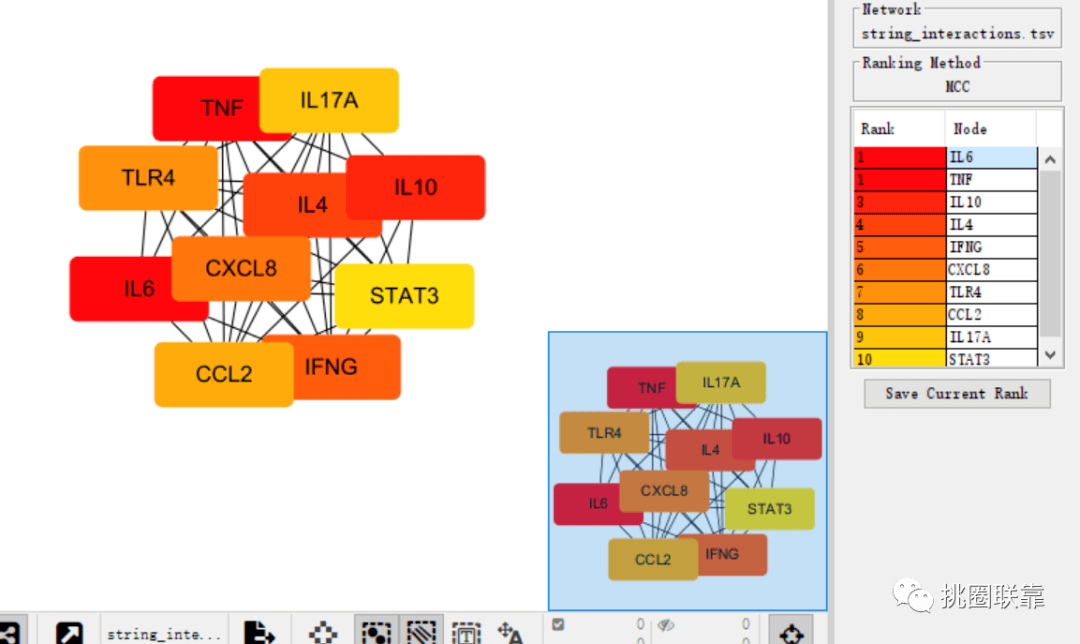
即可显示出三个数据库差异基因中,Top10得关键基因信息。
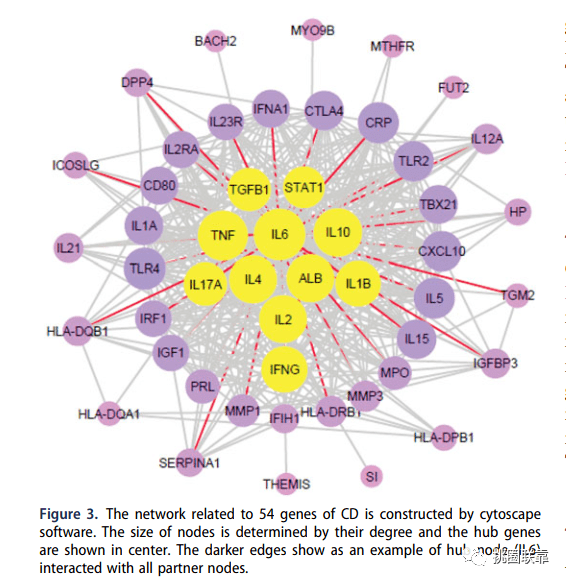
这里需要说明一下,原文中的图,是将所有54个差异基因全部显示,并且按照hub基因在中间,其他基因分别在周围的模式进行分布,这里我们不做过多演示,大家知道操作方法即可。( 话说美丽带了两次Cytoscape训练营了,如果还有不会的小伙伴,可能要等下一期开营咯)
复现任务4 图4 差异基因的PPI分析②
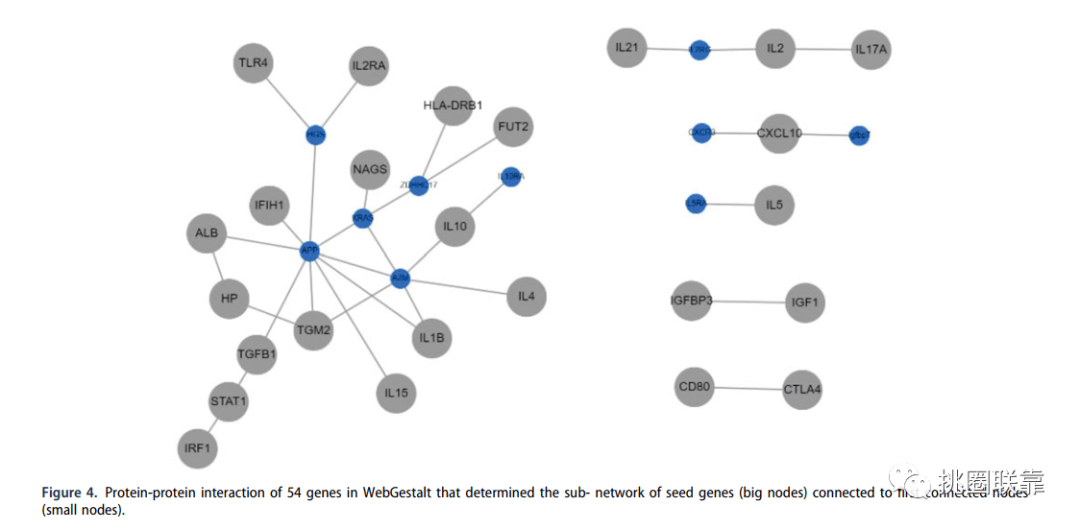
操作步骤
打开WebGestalt网站(http://www.webgestalt.org/)

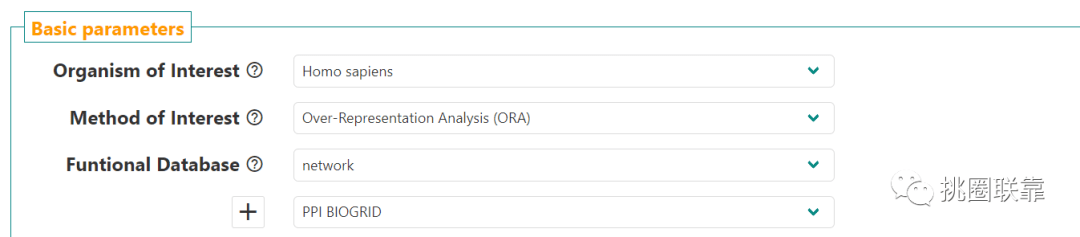
设置方法,以及基因集和详细信息
将基因名称全部粘贴
设置参考信息,点击提交

获取结果页面
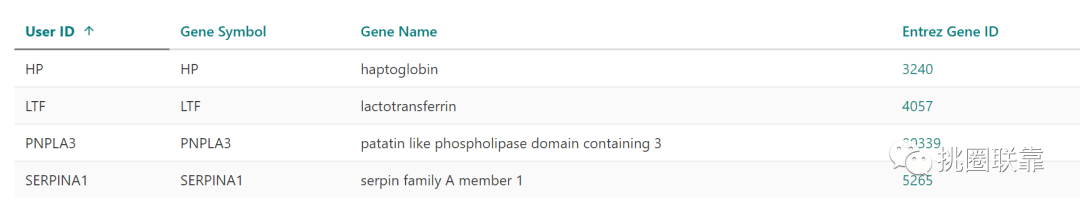
最下方显示Hub基因信息。
复现任务5 图4 差异基因的PPI分析②
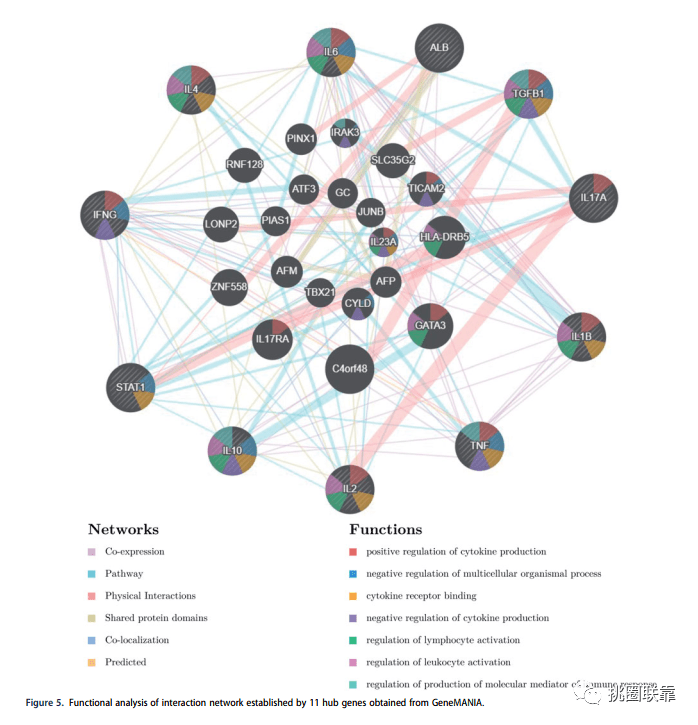
操作步骤
打开GeneMANIA网站(http://genemania.org/)
默认物种为人类,将全部基因粘贴在检索框中,点击检索
程序运行中( 目前GeneMANIA网站不是很好用,检索了好久也没出结果,希望小伙伴们实操的时候可以获取检索结果,美丽在这里主要跟大家介绍一下这个数据库的使用方法即可)
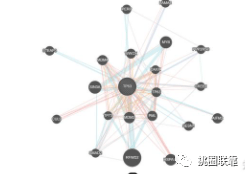
最终可以获得类似上图的结果
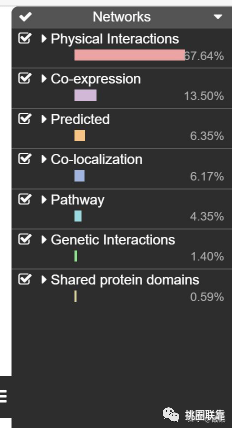
然后我们可以根据个性化按钮进行调整。
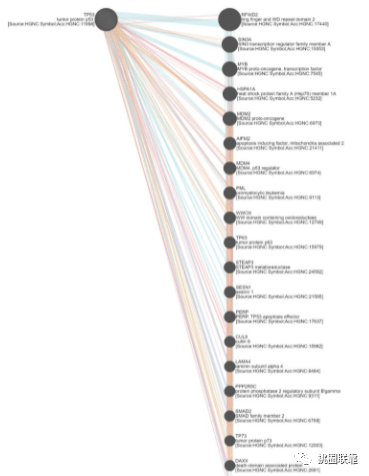
修改布局格式,直至最终结果。
复现任务6 图6 多套数据集的共同表达基因韦恩图
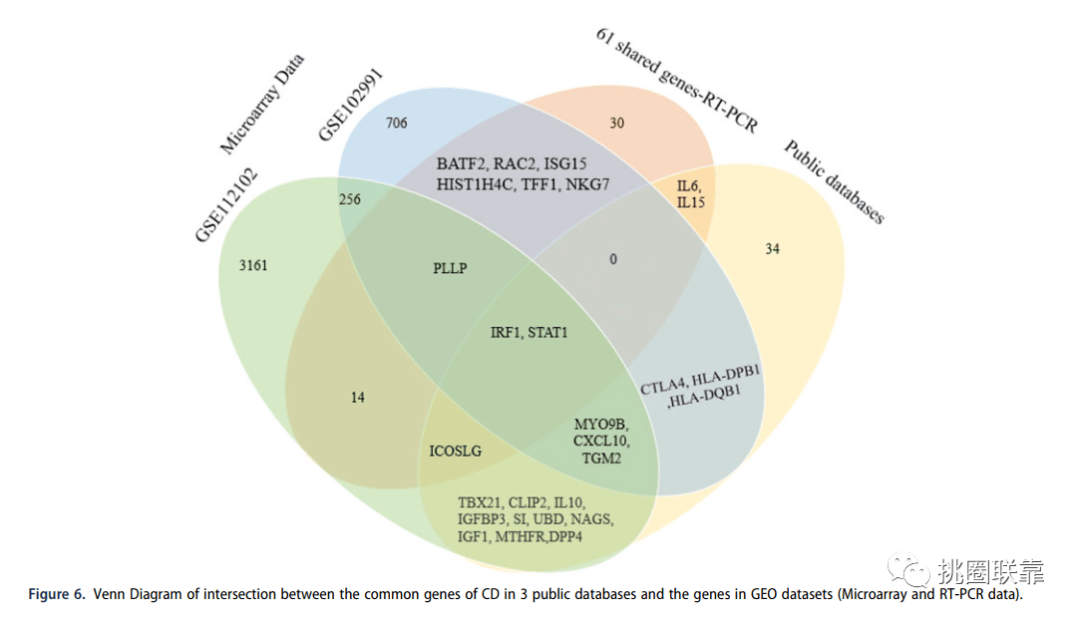
准备工作
数据清洗、整理
设置实验组和对照组,并分析获取差异基因。
依次将文中提及得基因集进行整理。
利用前面介绍过得仙桃工具-韦恩图模块,进行差异基因韦恩图的绘制。( 操作步骤同上,故省略)
复现任务7 表格
表1 在OMID中,CD相关的前十种疾病(详见复现图1)
表6 hub基因信息(详相见图3)
表2三种数据集的GO-BP通路结果
表3三种数据集的GO-MF通路结果
表4三种数据集的GO-CC通路结果
表5三种数据集的KEGG富集分析结果
Tips:
这里得表2-5均为通路富集分析结果,可以一起进行绘制,也是有些凑工作量得嫌疑yo~
复现步骤
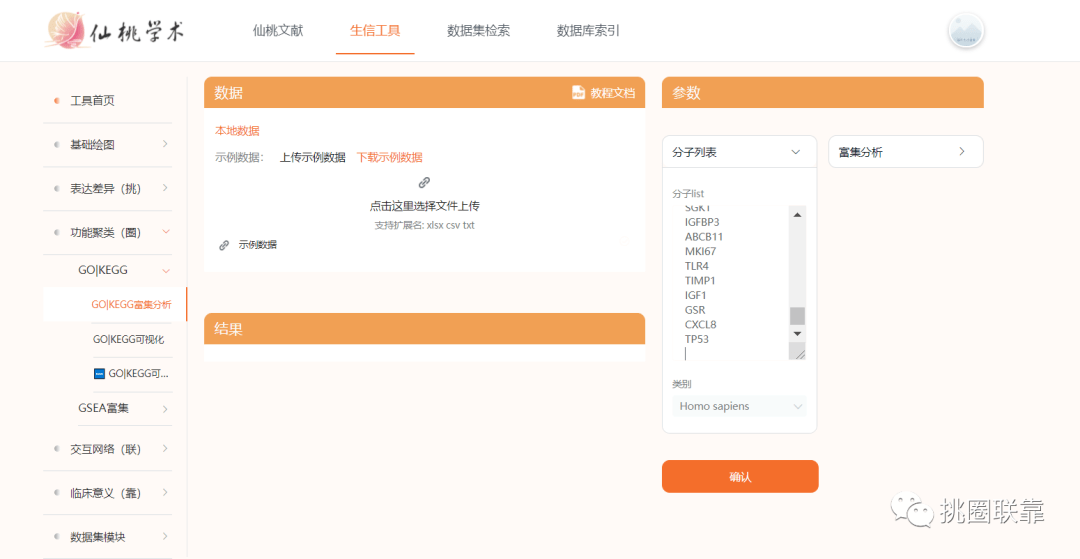
打开仙桃学术,功能聚类-gokegg分析,输入差异基因集,点击确认进行计算。
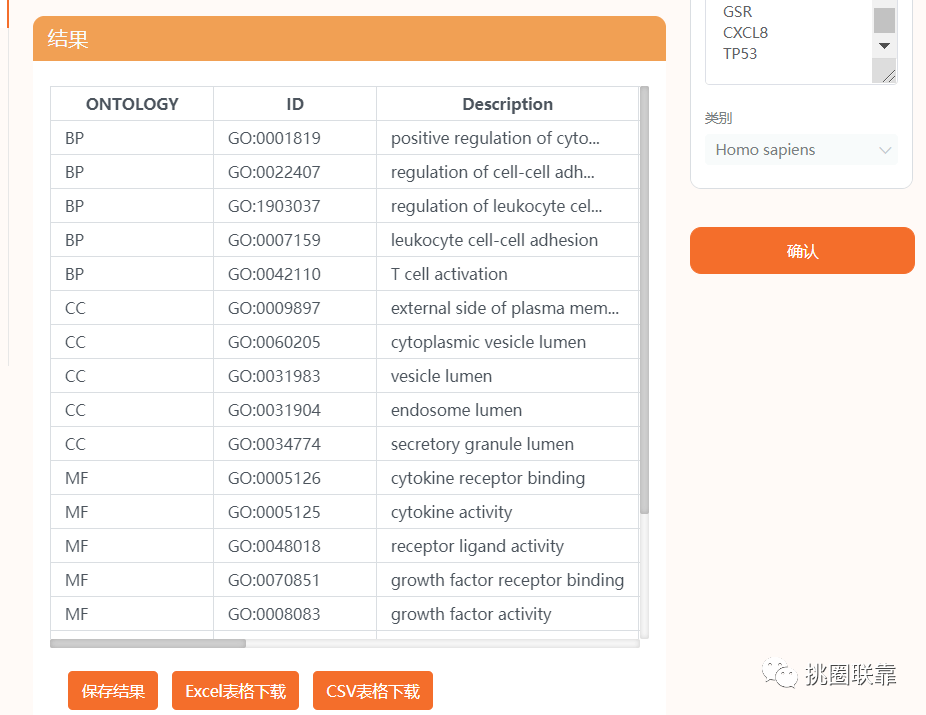
保存结果,分别将BP,CC,MF,KEGG整理为表2-5。
好了,以上是复现步骤。
全文总结
最后一个模块:根据本文的图表进行逻辑梳理
挑图2 三组数据库的疾病韦恩图
联图3 差异基因的蛋白互作网络
联图4 差异基因的PPI分析②
联 图5 hub基因的富集分析
挑图6 多套数据集的共同表达基因韦恩图
挑表1 在OMID中,CD相关的前十种疾病
圈表2三种数据集的GO-BP通路结果
圈表3三种数据集的GO-MF通路结果
圈表4三种数据集的GO-CC通路结果
圈表5三种数据集的KEGG富集分析结果
挑表6 hub基因信息
挑图7 WebGesalt获取的种子基因
挑图8 GEO数据挖掘的上调、下调基因
挑图9 GEO数据集的共同差异基因展示
美丽的唠叨时刻——思路串讲
首先,想到疾病或表型数据集,大家最先在脑海里浮现的,就是Genecard网站,但是作者,没有局限在一个网站,而是 联合CTD数据库(http://ctdbase.org/)、Genecards数据库(https://www.genecards.org/)、DISEASE数据库(https://diseases.jensenlab.org/Search)三个网站,进行共同差异基因分析,从而绘制出全文的大半个江山的版图,接下来的GEO数据库,可以说是锦上添花,只占据很少的笔墨,也没有走常规套路的分析过程。整体给人耳目一新的感觉。这一点,也是大家在分析的时候,值得借鉴的地方。
这里我们发现,不论是获得的单个数据集数量,还是最终混合的基因集个数,都远远超过文献本身的数据。可能的原因包括数据更新,或者作者增加其他检索关键词等。
最后,美丽再次提醒大家,我们学习复现,是学思路的,不是为了100%复现出图,哪怕全部复现出来,文章也是已发表的,对我们而言,思路为王,复现是辅助工具,能够100%一致,绝对是小概率事件。哪怕作者自己,也不能保证再次分析,结果还是一样的。所以,大家不要再问,为啥我跟你的步骤一样,但是最后,分析的结果差别这么大了。
希望通过本文复现,可以给大家一点新的思路,争取早日拿下自己的第一篇生信文章。发挺!
— END—
撰文 丨王美丽
排版丨四金兄
主编丨小雪球
转自解螺旋生信频道-挑圈联靠