肿瘤变异数据分析和可视化工具maftools:安装和文件格式要求
2023-3-5 萌小白
Maftools系列文章:
《maftools使用方法总结以及常见问题》
《肿瘤变异数据分析和可视化工具maftools:安装和文件格式要求》
《肿瘤变异数据分析和可视化工具maftools:突变数据下载和可视化》
《肿瘤变异数据分析和可视化工具maftools:突变的数据分析》
《肿瘤变异数据分析和可视化工具maftools:CNV的可视化》
Maftools简介
Maftools是一款可以对MAF格式(Mutation Annotation Format)的变异数据进行统计、分析和可视化的R包。除了可以对TCGA来源的MAF文件以外,其他任何变异数据只要是MAF格式都可以使用这款工具进行分析。
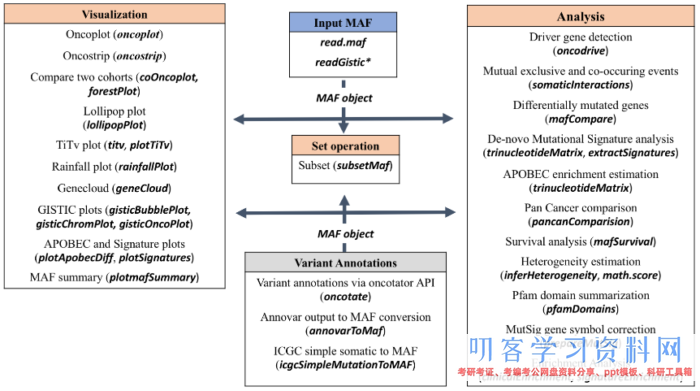
Maftools包可主要概括为可视化和分析两大模块,流程和使用方法很简单:通过read.maf读入MAF文件(或者经过格式转换)得到MAF对象,然后将对象传递给对应的分析或者可视化函数就行了。主要模块、函数和主要的分析和可视化功能见下图:
Maftools和相关依赖的安装
从Bioconductor安装,推荐使用BiocManager安装Bioconductor包,biocLite()已过时:
if (!require(\"BiocManager\"))
install.packages(\"BiocManager\")
BiocManager::install(\"maftools\")
从GitHub安装最新开发版,包含了一些Bioconductor分支可能没有的功能(推荐,因为感觉maftools目前问题还挺多,作者迭代速度也很快,另外Bioconductor上版本太低了):
# 从Bioconductor安装相关依赖.
if (!requireNamespace(\"BiocManager\", quietly=TRUE))
install.packages(\"BiocManager\")
BiocManager::install(\"ComplexHeatmap\")
BiocManager::install(\"VariantAnnotation\")
BiocManager::install(\"Biostrings\")
# 直接从GitHub仓库安装maftools
library(\"devtools\")
install_github(repo = \"PoisonAlien/maftools\")
文件格式及读入
1. 其他文件格式转换
如果使用VCF格式或者table分隔的MAF-like格式储存的变异数据,可以使用vcf2maf/maf2maf脚本很容易转换为MAF格式。另外所有样本的MAF文件在使用maftools前要合并成一个文件。
如果是ANNOVAR做的变异注释,可以使用maftools中的annovarToMaf进行格式转换。
可以使用icgcSimpleMutationToMAF处理ICGC的SSM格式(Simple Somatic Mutation)。
2. MAF格式的字段
TCGA下载的MAF文件没有问题,但是自己的课题得到要确认一下是否包含必须字段:
强制字段:Hugo_Symbol、Chromosome、Start_Position、End_Position、Reference_Allele、Tumor_Seq_Allele2、Variant_Classification、Variant_Type以及Tumor_Sample_Barcode。
可选但建议包含的字段:VAF(Variant Allele Frequency)以及氨基酸变化信息。
3. MAF文件读入和概括统计
需要的输入文件:
一个MAF文件,可以是gzip压缩的(必须)
与MAF文件中每个样本/Tumor_Sample_Barcode关联的临床数据(可选但是推荐)
拷贝数变异数据,可以是GISTIC的输出结果,也可以是table分隔的自定义文件,需包含样品名、基因名以及拷贝数状态如Amp或Del(可选)
附录
Maftools相关链接:
GitHub:https://github.com/PoisonAlien/maftools
Bioconductor:https://www.bioconductor.org/packages/release/bioc/vignettes/maftools/inst/doc/maftools.html
发表论文:https://www.ncbi.nlm.nih.gov/pubmed/?term=30341162
关于MAF格式的和SSM格式的详细介绍可阅读:
https://docs.gdc.cancer.gov/Data/File_Formats/MAF_Format/
https://docs.icgc.org/submission/guide/icgc-simple-somatic-mutation-format/
发表评论: