ATAC-Seq剖析教程系列
ATAC-Seq剖析教程:ATAC-seq的布景介绍以及与ChIP-Seq的异同
ATAC-Seq剖析教程:原始数据的质控、比对和过滤
ATAC-Seq剖析教程:用MACS2软件call peaks
ATAC-Seq剖析教程:对ATAC-Seq/ChIP-seq的质量评价(一)phantompeakqualtools
ATAC-Seq剖析教程:对ATAC-Seq/ChIP-seq的质量评价(二)ChIPQC
ATAC-Seq剖析教程:重复样本的处理-IDR
ATAC-Seq剖析教程:用ChIPseeker对peaks进行注释和可视化
ATAC-Seq剖析教程:用网页版工具做功能剖析和motif剖析
ATAC-Seq剖析教程:差异peaks剖析——DiffBind
ATAC-Seq剖析教程:ATAC-Seq、ChIP-Seq、RNA-Seq整合剖析
1. 学习方针
评论ChIP-seq数据质量评价的其他方法
用ChIPQC发生质量计算陈述
鉴定低质量数据的来历
概览图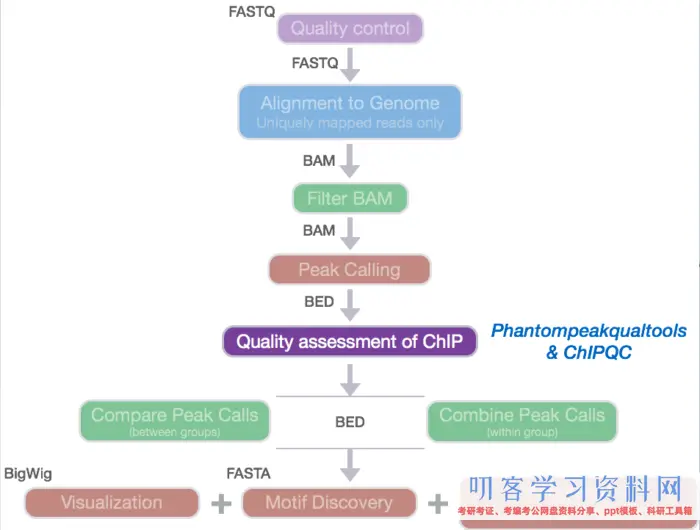
ENCODE评价数据质量采用多种目标,如前面已经评论过的链相关的目标NSC和RSC。这一节将会评论评价信号散布的其他目标。
NOTE:这里给出的评价目标仅仅反映数据质量的好坏,契合阈值的并不意味着试验是成功的,不契合阈值的也不一定意味着失利。
2.常见质量评价目标的介绍
SSD
SSD值是对富集作用的评价。SSD值依赖于全基因组的pile-up信号强度,对实在的ChIP富集和搅扰的强信号区域都很灵敏。SSD值越大标明富集越好。
“It
provides a measure of pileup across the genome and is computed by
looking at the standard deviation of signal pile-up along the genome
normalised to the total number of reads. ”
FRiP:Fraction of reads in peaks
FRiP表示的是peaks中的reads与总reads的份额。它是另一个反映样本富集作用或IP好坏的评价目标。能够理解为是“信噪比”即文库中结合位点片段占布景reads的份额。一个典型质量好的TF富集FRiP值约5%或许更高,polII的FRiP值约为30%或许更高,也有一些质量好的数据FRiP值<1%(如RNAPIII)
Relative Enrichment of Genomic Intervals (REGI)
REGI是对peaks在不同基因组特征位点散布的计算。
RiBL: Reads overlapping in Blacklisted Regions
过滤人工形成的高信号区域非常重要,如ENCIDE和modENCODE提供的DAC
Blacklisted Regions
track。这些区域经常在特定的重复序列处出现,如着丝粒、端粒、卫星重复序列等,经过简单的比对过滤是不能去除的。来自blacklisted
regions的信号会形成call peak 和片段长度评价的混淆。
RiBL值能够表示布景信号或input的信号水平,与input sample的SSD值以及input和ChIP sample的读长掩盖值相关。这些区域通常是基因组的0.5%,或许更高的份额(10%)。
3. ChIPQC: quality metrics report
ChIPQC是一个Bioconductor包,输入文件包含BAM和peak文件,能够主动计算一些质量评价值,并发生质量陈述。
预备数据
BAM files
首要对比对过滤后的bam数据(chr12_aln.bam)建索引,然后将bam和index文件从~/ngs_course/chipseq/results/bowtie2移动到自己的目录文件夹data/bams
peak files
将narrowPeak 文件从macs2目录下~/ngs_course/chipseq/results/macs2移动到自己目录下data/peakcalls
sampleSheet file
sampleSheet file是仅有需求自己依据试验设计和数据存储地址等信息创立的一个csv格式文件(bam,peak文件分别在比对和call peak的过程发生)。sampleSheet具体需求包含的信息如下:

SampleID: 样本ID
Tissue, Factor, Condition: 不同的试验设计对照信息,三列信息必须包含在sampleSheet里,假如没有某一列的信息设为NA。
Replicate : 重复样本的编号
bamReads : 试验组BAM 文件的途径(data/bams)
ControlID : 对照组样本ID
bamControl :对照组样本的bam文件途径
Peaks :样本peaks文件的途径
PeakCaller :peak类型的字符串,能够是raw,bed,narrow,macs等。
下载安装ChIPQC
source(\"http://bioconductor.org/biocLite.R\")
biocLite(\"ChIPQC\")
Running ChIPQC
ChIPQC只需求三步就能够完成质量评价和陈述生成。
首要载入包和sampleSheet信息
## Load libraries
library(ChIPQC)
## Load sample data
samples <- read.csv(\'meta/samplesheet_chr12.csv\')
View(samples)
创立ChIPQC目标
使用sampleSheet的信息读取每个样本的bam和narrowpeak文件,并计算质量评价值,成果存在一个目标里。
## Create ChIPQC object
chipObj <- ChIPQC(samples, annotation=\"hg19\")
生成ChIPQC陈述
## Create ChIPQC report
ChIPQCreport(chipObj, reportName=\"ChIP QC report: Nanog and Pou5f1\", reportFolder=\"ChIPQCreport\")
ChIPQC陈述解读
ChIPQC生成的成果包含一个网页陈述和陈述中含有的一切图片。
网页陈述有三部分:QC Summary ;QC Results;QC files and versions

(1)QC Summary - Overview of results
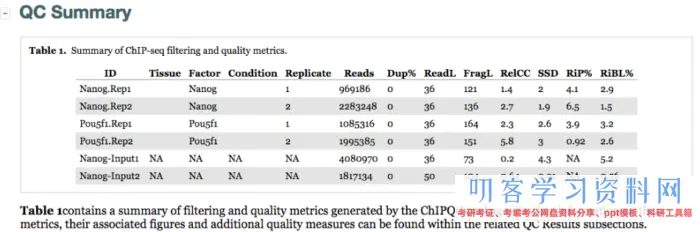
QC
summary包含sampleSheet里填写的样本的基本信息Tissue,Factor,Condition,Replicate。另外还有上面说到的质量评价的常用目标SSD、RiP%和RiBL值。越高的SSD值标明富集作用越好,Pou5f1样本(2.6,3)有较高的SSD值,RiBL值不是很高,FRiP的份额在5%邻近或许更高,除了Pou5f1-rep2。
SSD - SSD score (htSeqTools)
RIP% - Percentage of reads wthin peaks
RIBL% - Percentage of reads wthin Blacklist regions
同时表格中还给出了其他计算信息:
Reads - Number of sample reads within analysed chromosomes.
Dup% - Percentage of MapQ filter passing reads marked as duplicates
FragLen - Estimated fragment length by cross-coverage method
FragLenCC - Cross-Coverage score at the fragment length
RelativeCC - Cross-coverage score at the fragment length over Cross-coverage at the read length
(2)QC Results - Full QC results and figures
Mapping, Filtering and Duplication rate
榜首部分是比对、过滤和重复率质检成果,包含Table2 、Figure1和Figure2。
Table 2首要给出了比对质量和重复率,因为BAM文件是过滤后的,所以这里Dup%都是0.
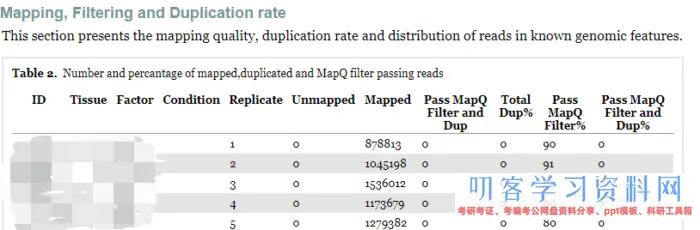
Total Dup%-Percentage of all mapped reads which are marked as duplicates.
Pass MapQ Filter%-Percentage of all mapped reads whichpass MapQ quality filter
Pass MapQ Filter and Dup%-Percentage of all reads which pass MapQ filter and are marked asduplicates.
Figure 1展示了reads在blacklists中的份额,
Figure 2是用基因组注释出现了reads在基因组特征方位如启动子的散布。这幅图里显现在启动子区域富集最显着。
ChIP signal Distribution and Structure
第二部分是ChIP信号散布和结构组成,包含Figure3和4。
Figure 3是一个coverage
plot, x轴代表在某bp方位read pileup的高度,y轴代表有多少方位有相同的pileup
高度(取log)。**有好的富集的ChIP样本会有一个tail,即更多的方位(y值大)有较高的测序深度。在我们的数据会集Nanog样本与Pou5f1
相比有较高的tails,尤其是重复样本2。可是Pou5f1有较高的SSD值。当SSD高可是coverage看起来低时,或许是存在大片段深度高的区域出现在blacklist
基因组区域。
Peak Profile and ChIP Enrichment
第3部分是peak的谱图和ChIP的富集,每个peak都会集在summit方位(summit 理解为peak的最高峰值点处)
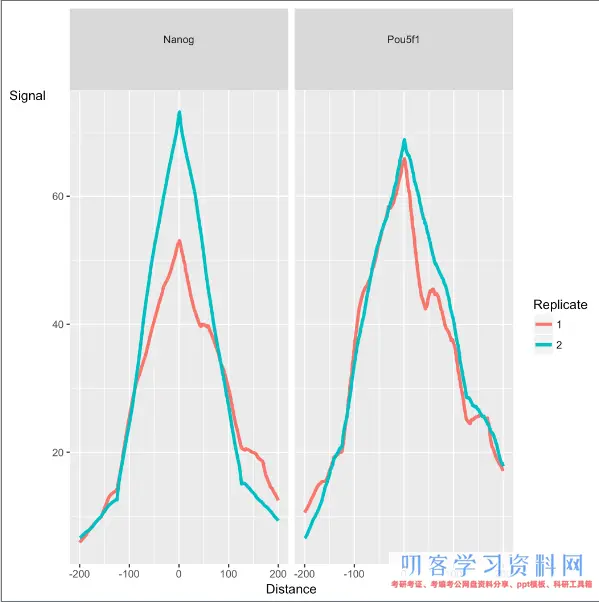
peak的性状取决于研究目标的类型,如转录因子、组蛋白标记、或其他DNA结合蛋白如聚合酶等,相同类型的目标通常有共同特征的谱图。
Figure6和7都是对比对到peak中的reads计算。富集作用好的ChIP样本的reads与peaks会有高份额的重合。尽管Nanog有较高的RiP,可是两个重复样本间的差异大于Pou5f1。
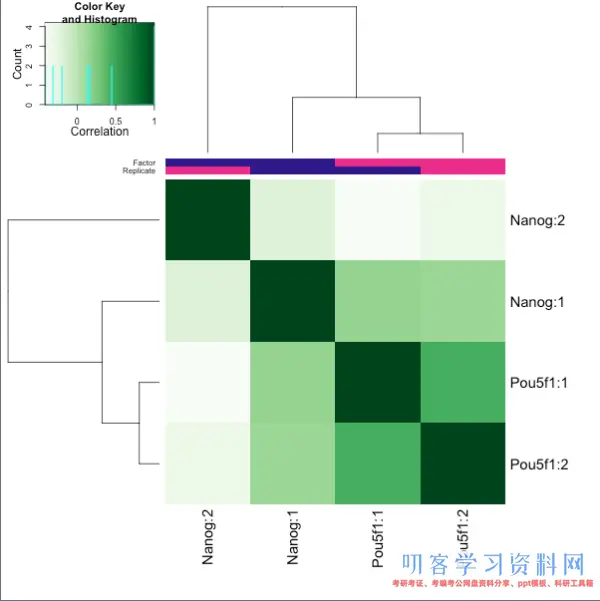
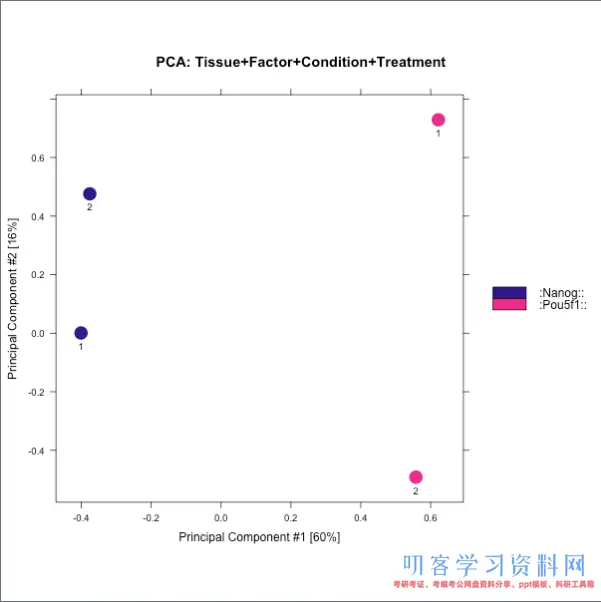
Figure8和9**表示样本的聚类作用,分别是相关性聚类热图和PCA。
4. 试验误差:ChIP-seq数据质量低的来历
免疫沉淀的特异性和有效性
影响因素如抗体的特异性,结和沉淀的强度
片段化
超声裂解发生不同大小的片段或许引进误差
文库构建时的误差
如PCR扩增