生信实操
ENA数据库:European Nucleotide Archive,隶属European
Bioinformatics Institute, 功能类似于SRA,
对每个研究产生的数据做了详细注解,主要优势:界面简介友好,直接以表格呈现,且可直接得到原始数据文件的下载地址,并且不用花时间做fastq数据文件的转换,对有数据需求的研究人员来说这是个数据再次挖掘的金库。
列举下生物数据的四大数据库,作为生信人员一定要必知:
>>NCBI(https://www.ncbi.nlm.nih.gov/):美国国家生物信息中心(National
Center for Biotechnology
Information,NCBI),它由基础数据库(一级)和派生数据库(二级组成),基础数据由相关研究的实验人员提交和修订,如GenBank、SNP、GEO、PubChem、Substance;二级数据由专业人员或第三方管理、编辑和修订,如NCBI
RefSeq、 UniGene、TPA、RefSNP、Protein、Sructure等。
>>ENA(https://www.ebi.ac.uk/):
欧洲分子生物实验室(European Nucleotide Archive),由EMBL-Bank
核酸序列数据库基础上发展起来,EMBL数据直接来源于测序工作者提交的数据,与其它数据机构协作交换的数据,欧洲专利局提供的数据,是欧洲最重要的核酸序列资源。
>>DDBJ(https://www.ddbj.nig.ac.jp/ddbj/index-e.html): 日本遗产研究所(DNA Data Bank of Japan):数据来源主要是日本研究者提交的序列和其它数据机构协作交换的数据。
>>BIGD(https://bigd.big.ac.cn/): 北京基因组研究所生命与健康大数据中心(BeiJing Institute of Genomics Data Center),目前的数据资源系统包括高通量测序的原始组学数据归档库GSA,具备可服务全球的基因组数据共享网络。

DDBJ、NCBI、ENA3个数据库在1988年已经达成数据协议,每天交换信息,并对某些测序记录达成了统一标准。每个机构负责收集来自不同地理分布的数据。
这四个主要数据库的区别首先当然是国家和地域的区别,中国也是生物组学数据产出的大国,推荐大家在以后的数据提交中优先选择BIGD,它在国内,上传和下载的速度都是优于其它数据库的,是中国科学家自己的数据库,下期教大家使用!
由于在文献里看到感信息的数据集,但是存放在国外数据库如ENA,所以一定也要会使用。
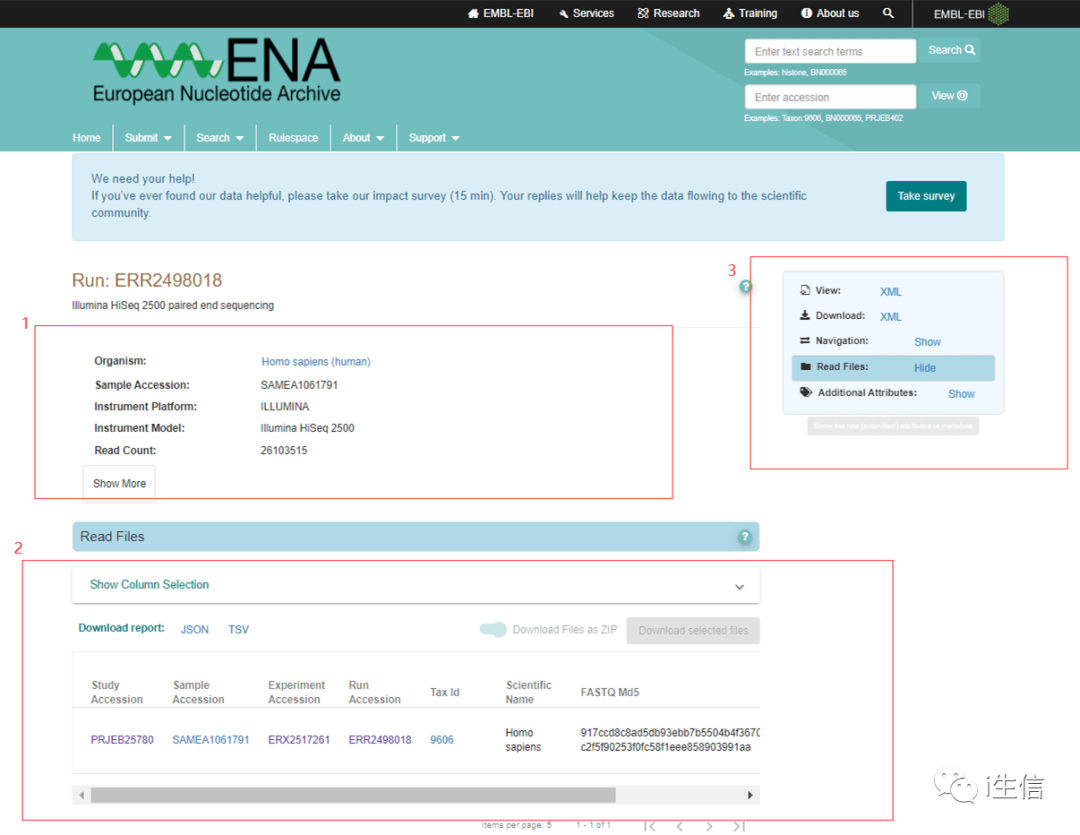
1:为研究的元数据
2:研究的数据访问链接
3:可选择你对当前研究感兴趣的信息项进行展示
那如何获得下载地址呢?以下操作我们看图进行,一定要自己去ENA数据库点一点才能记住哦,一定要跟着我一起去啦!

1.首先,看下图点击, 详细查看当前研究的数据样本信息
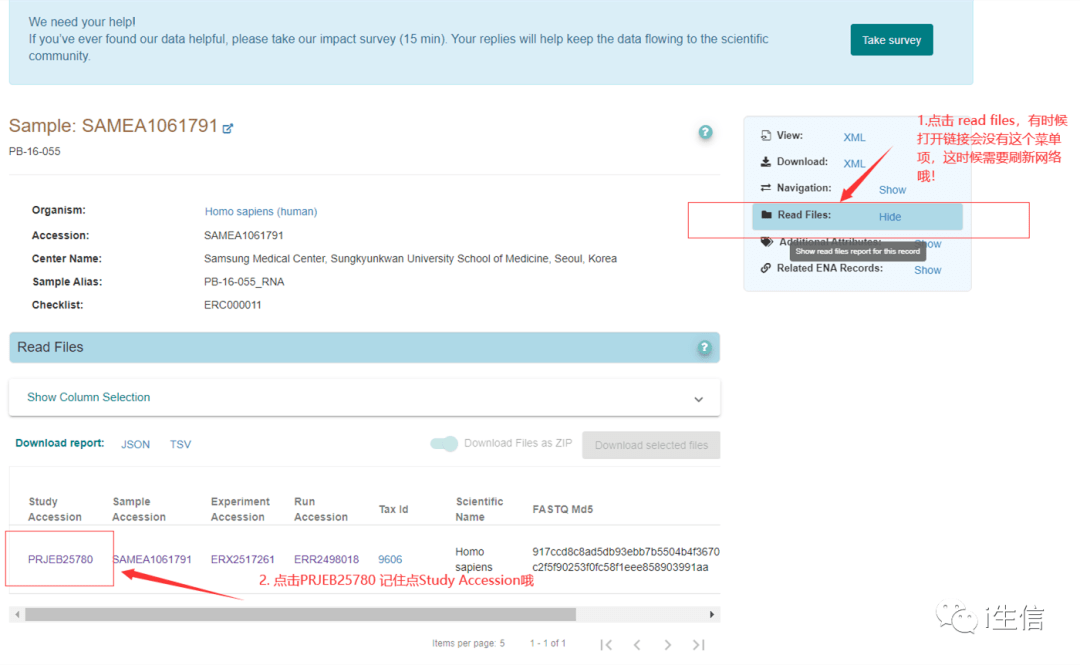
2.进入样本详细页面后点击Show Column Selection,就可以选择下载数据的下载地址了
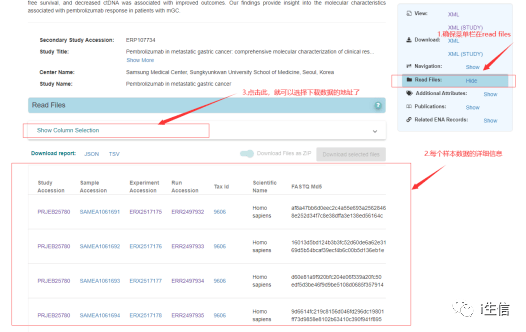
3.选择样本详细的具体信息后, 点击TSV下载哦
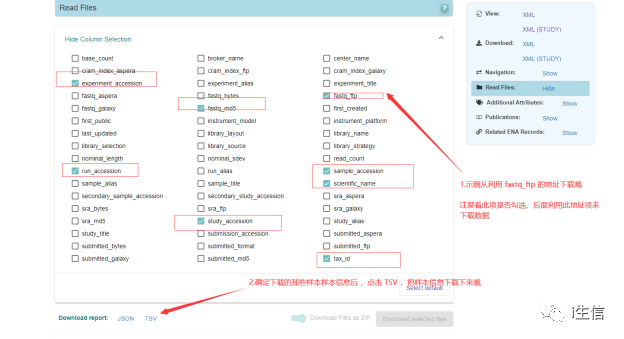
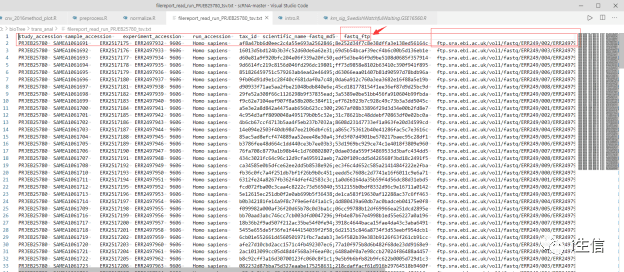
现在我们来看看这个TSV长什么样子:
可以看到,我们选的列就是下载的TSV文件的表头了,每一行代表一个样本,Fastq_ftq 就是样本的下载地址,那么如何下载呢?这里推荐使用Aspera( https://www.ibm.com/aspera/connect/),因为它相比其它下载软件速度更快,且不用做关于fastq的数据转换
4.下载Aspera( https://www.ibm.com/aspera/connect/),根据下载地址到官网选择自己要下载的版本,这里我们分析数据都是在Linux 上,所以一般都用下载Linux版本,把数据直接搞到服务器上
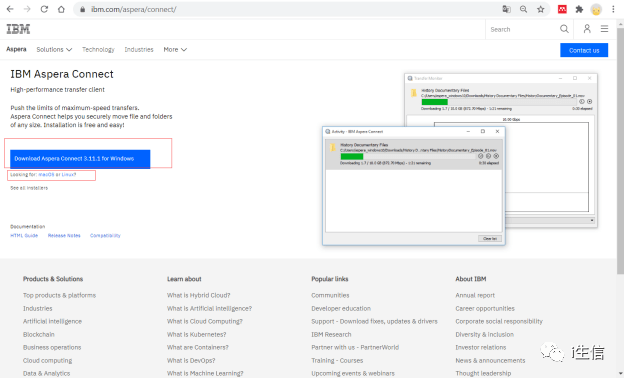
下载后解压后,获得的直接是shell脚本,直接安装
tar -xvf ibm-aspera-connect-3.11.1.58-linux-g2.12-64.tar.gz
默认安装的位置在当前用户的家目录下,方便使用,在当前用户的环境变量中加入它的路径
vim/home/zhenggm/.bashrc # 在用户环境文件下加入 export PATH="/home/zhengm/.aspera/connect/bin:$PATH"
source /home/zhenggm/.bashrc
export PATH="/home/andy/.aspera/connect/bin:$PATH"
然后拷贝下密钥文件,方便使用
cp ~/.aspera/connect/etc/asperaweb_id_dsa.openssh ~/
5.使用方法——单个数据下载
ascp 表示数据的下载命令,-i 表示密钥文件的指定,-QT参数,表示开启断点续传,-l带宽限制,启用该设置能够再次加快速度,-P 表示通信端口
era-fasp 表示数据下载的域名,这里从ENA下载所以域名指定为era-fasp,@后面
可以看到,是刚才在ENA 下载的TSV文件数据的fastq_ftq列的信息,具体格式为:
它就是@后指定的下载地址,单个指点下载地址为
命令中的
/udata/zhenggm/bioTree/trans_upAnal/PRJEB25780_trans/
为指定的数据下载的存放路径
6.使用——多个数据集的下载
样本量超过一个建议就进行批量下载哦,这样一个一个的下也不是个头啊!
首先构造下载地址的文档,把在ENA下载的tsv文件中的fastq_ftq列信息整理出来,并把原来的“ftp.sra.ebi.ac.uk”替换成“fasp.sra.ebi.ac.uk:”,得到如下的样本下载地址文档
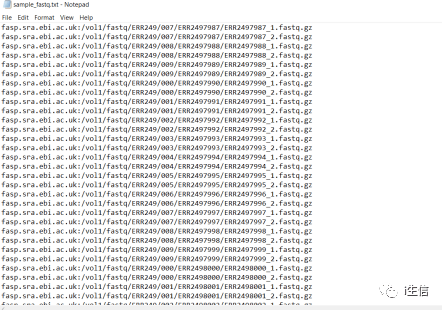
然后写shell脚本(aspera_download.sh)来循环下载:
cat sample_fastq.txt | while read id
do
ascp -i
/home/zhenggm/asperaweb_id_dsa.openssh -QT -l 300m -P33001 era-fasp@$id
/thinker/storage/udata/big/zhenggm/bioTree/trans_upAnal/PRJEB25780_trans/
done
然后在系统里后台运行此下载就可以坐等数据下载好了哦
nohup sh aspera_download.sh > step1.log 2>&1 &
7.如何一劳永逸?
还是会有小伙伴抱怨中途数据下载失败就得重新开始数据下载,得手动去看那些下好没,那些没下好,这里给大家提供一个升级版的python3下载脚本,可以自动检查那些数据下载成功,然后只下载没有下载成功的,直到数据都下载成功,超级方便,赶快用起来
importsubprocess
importargparse
importos
'''
需要替换-i指定的密钥文件位置,以及下载文件的位置,
'''
defrun_cmd(ids: list):
foridinids:
cmd = """ascp -i /home/zhenggm/asperaweb_id_dsa.openssh -QT -l 300m -P33001 era-fasp@{id}/thinker/storage/udata/big/zhenggm/bioTree/trans_upAnal/PRJEB25780_trans/"""\
.format(id=id)
p = subprocess.Popen(cmd, shell=True)
p.wait
p.terminate
argparser = argparse.ArgumentParser
argparser.add_argument('--conf_file', type=str, default='')
args = argparser.parse_args
conf_file = args.conf_file
print(conf_file)
id_list = []
withopen(conf_file, 'r') asf:
line = f.readline.replace('\n', '')
whileline:
id_list.append(line)
line = f.readline.replace('\n', '')
f.close
whilelen(id_list)>0:
done_list = [f forf inos.listdir(os.path.join(os.path.dirname(conf_file), 'PRJEB25780_trans')) iff.endswith('.gz')]
print(done_list)
id_list = [idforidinid_list ifid.split('/')[-1] notindone_list]
print('id count: %d'% len(id_list))
run_cmd(id_list)
这是依赖python3的脚本,需要安装python3,运行此python脚本需通过 –conf_file 来指定样本下载的文件哦,同样后台运行,可通过看日志得知数据的下载情况~
nohup python3 downlaod_fastq.py --conf_file sample_fastq.txt > step1.log 2>&1 &
今天的实操到这里,有问题可以留言~
转自:https://www.sohu.com/a/476510565_121124215