数据科学家通过许多工具软件利用庞大或者混杂数据去解决回答许多复杂的难题。
数据科学问题种类包括:
描述性分析(deive analyses)
探索性分析(exploratory analyses)
推断分析(inferential analyses)
预测分析(predictive analyses)
因果分析(casual analyses)
机理分析(mechanistic analyses)
在这期内容中我们将介绍数据科学家常用的工具软件的安装设置。
命令行界面CLI
命令行界面(command line interface)是用来处理文件和文件夹的一种方式。每一台计算机中都带有某种形式的命令行界面。
➢位置:
・windows:Git Bash
・Mac/Linux:Terminal
➢作用:
・浏览指定文件夹
・创建文件、文件夹、程序
・编辑文件、文件夹、程序
・运行程序
➢目录directories:
目录结构类似“树”
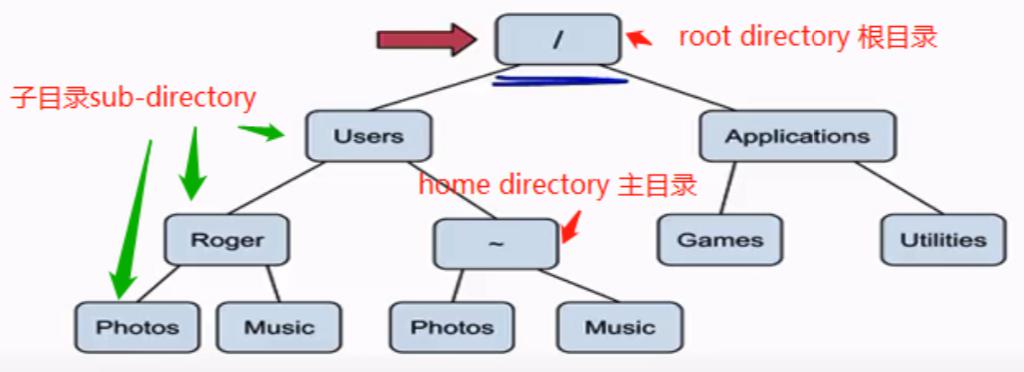
根目录:root,处于目录最顶端,用“/”表示
主目录:home,包含个人文件,通常使用用户名为名称,用“~”表示
打开CLI后,默认开始于Home目录,CLI正在工作的目录称为工作目录。从根目录到工作目录称为路径path。
➢命令格式:
命令 -参数选项 参数值
command -flagsarguments
示例:
clear
ls-a
cp-rDocuments More_docs
是否要用参数选项和参数值取决于命令本身,根据command,flags和arguments可以为0个或有多个。
➢常见命令:
・pwd:显示工作目录的路径 ・clear:清除当前窗口的命令
pwd
clear
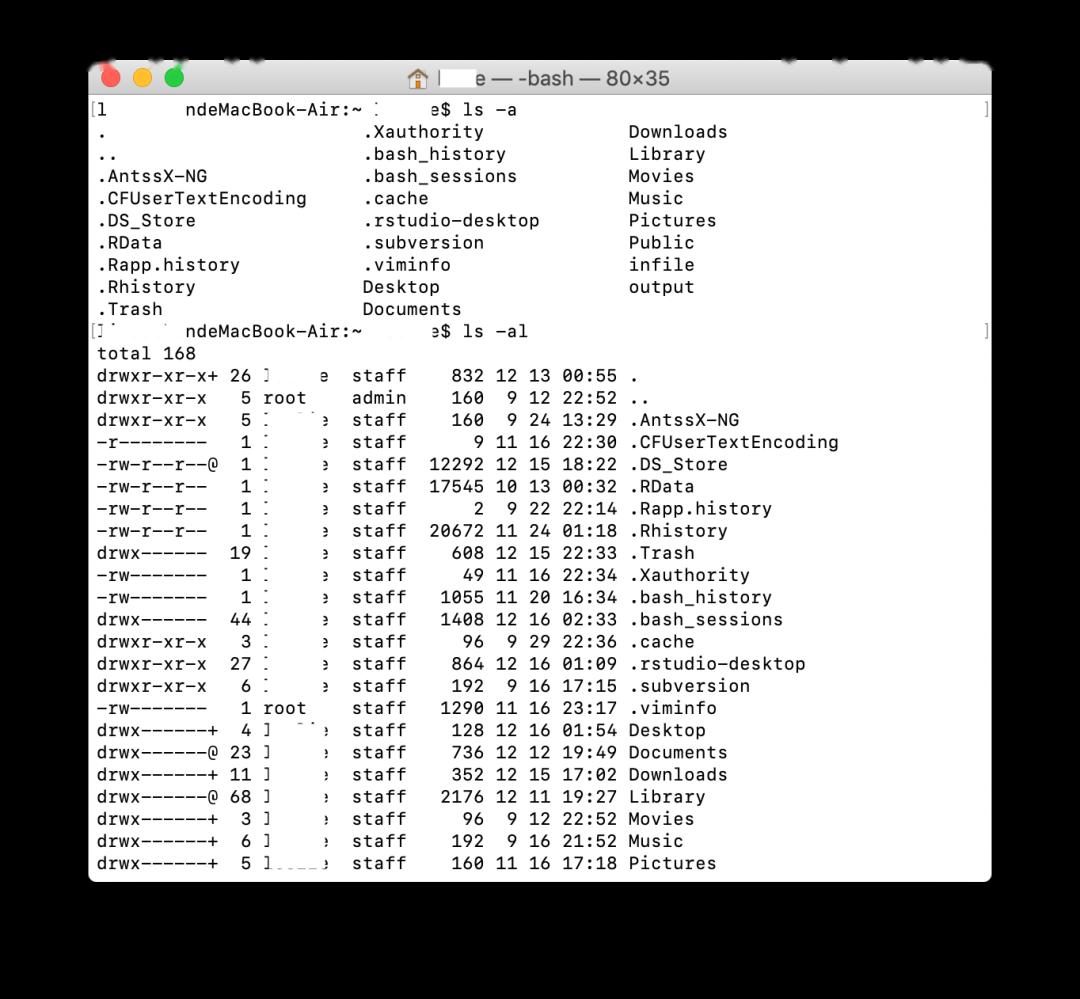
・ls:列出当前目录的文件和文件夹
ls-a#列出隐藏或非隐藏的文件和文件夹
ls-al#列出隐藏或非隐藏的文件和文件夹的细节
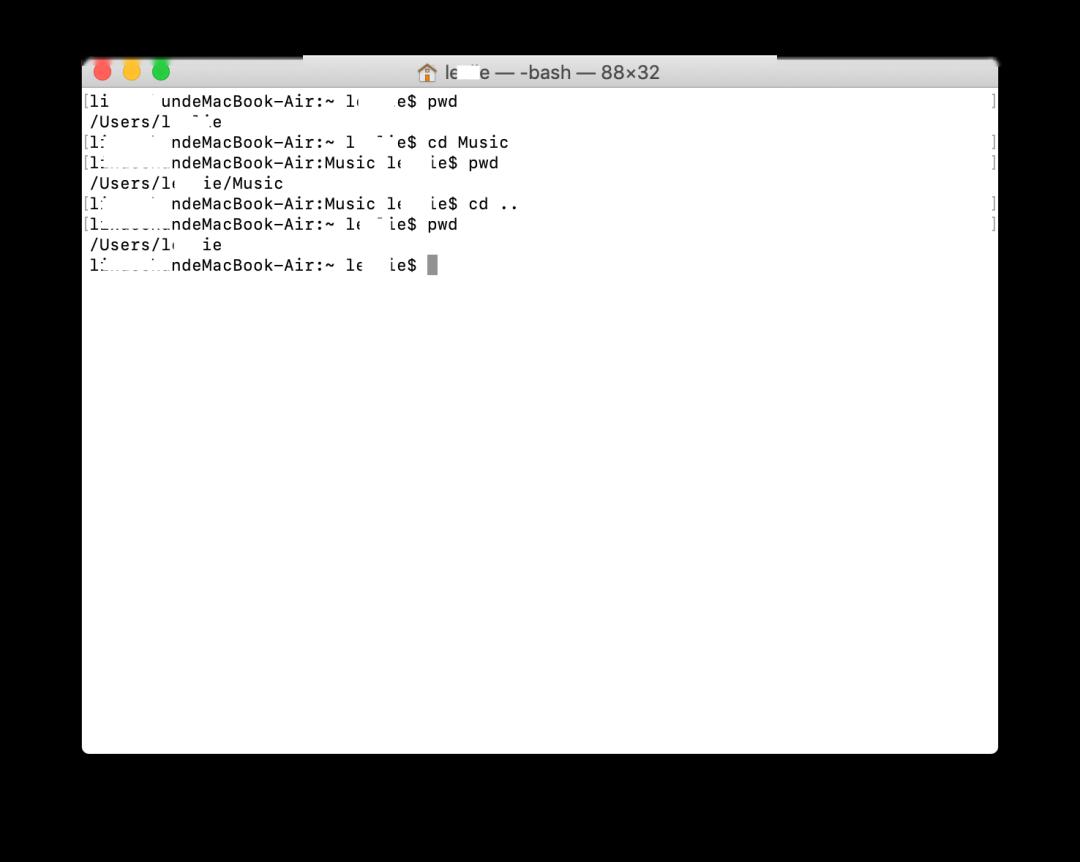
・cd:改变目录
cdMusic
cd.. #返回上一级目录
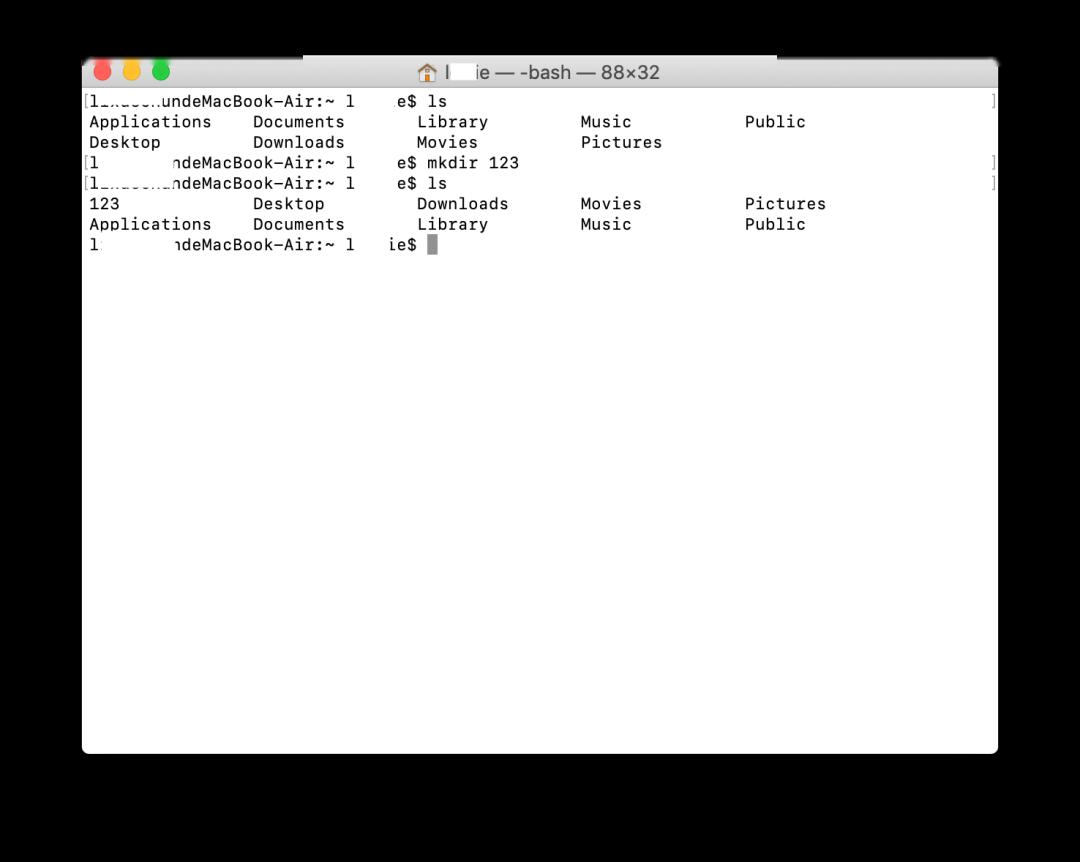
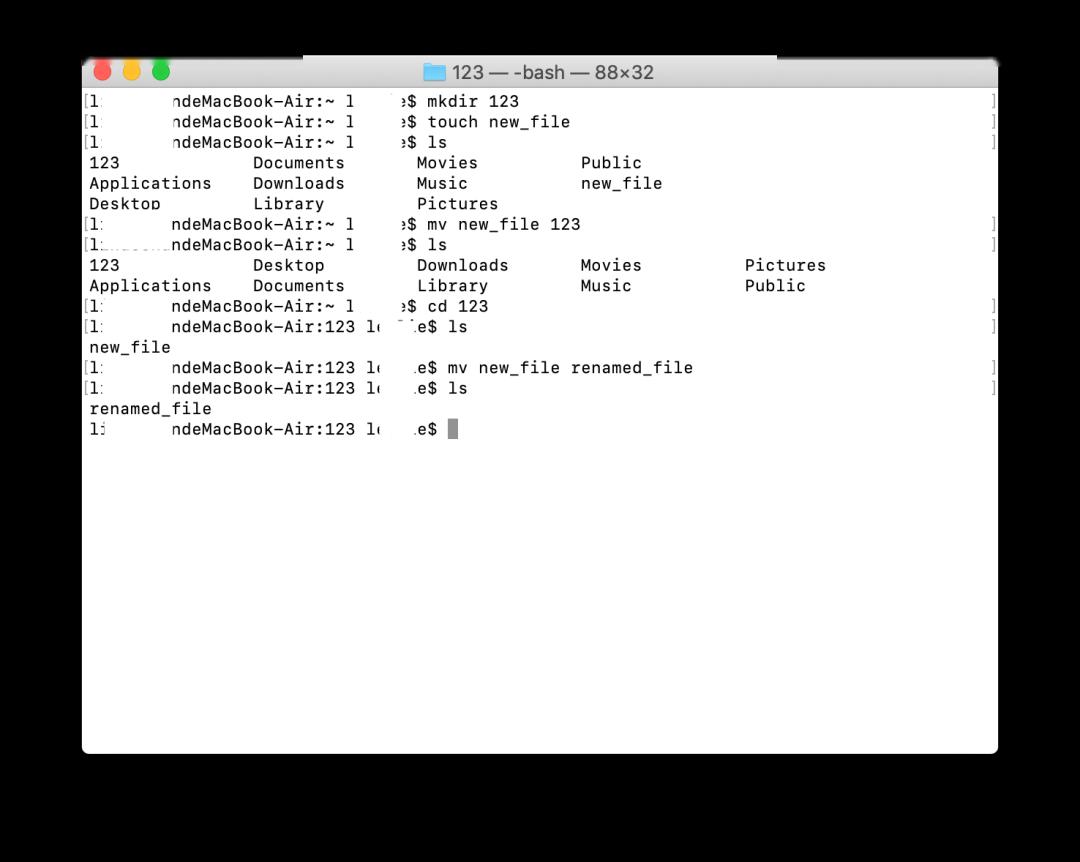
・mkdir:创建目录
mkdir123#创建名为123的目录
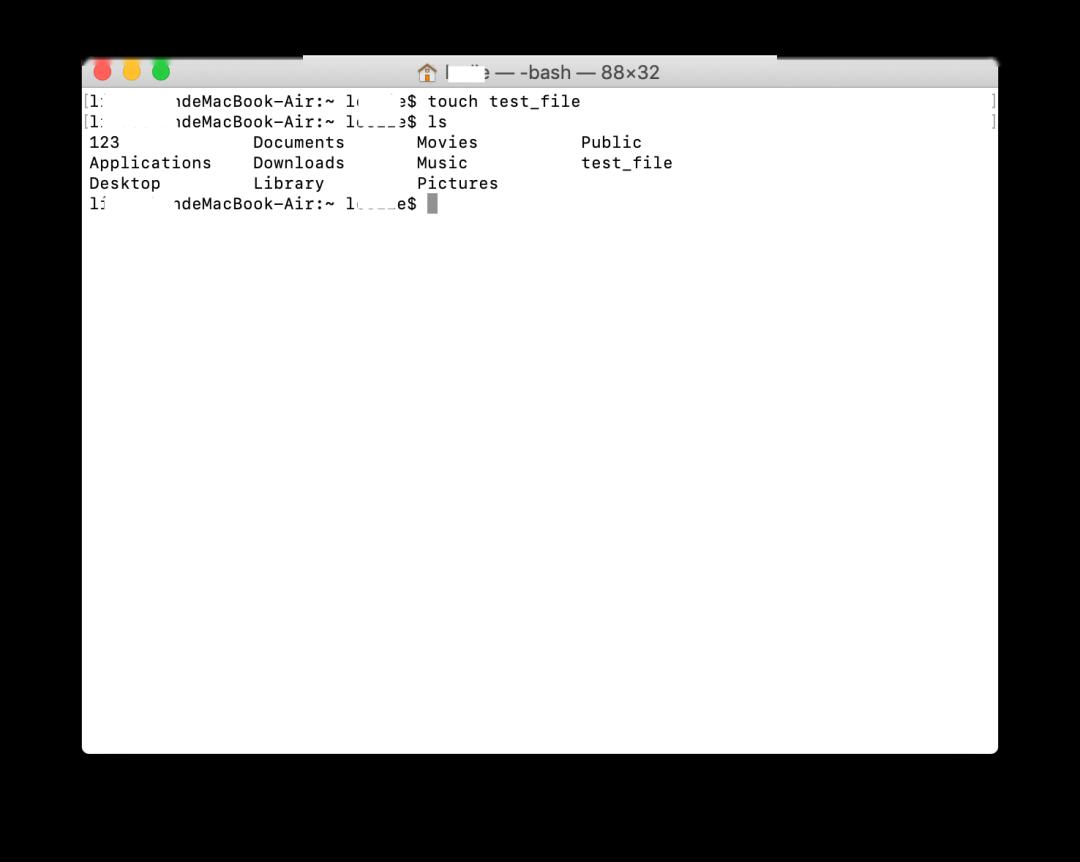
・touch:创建一个空文件
touchtest_file
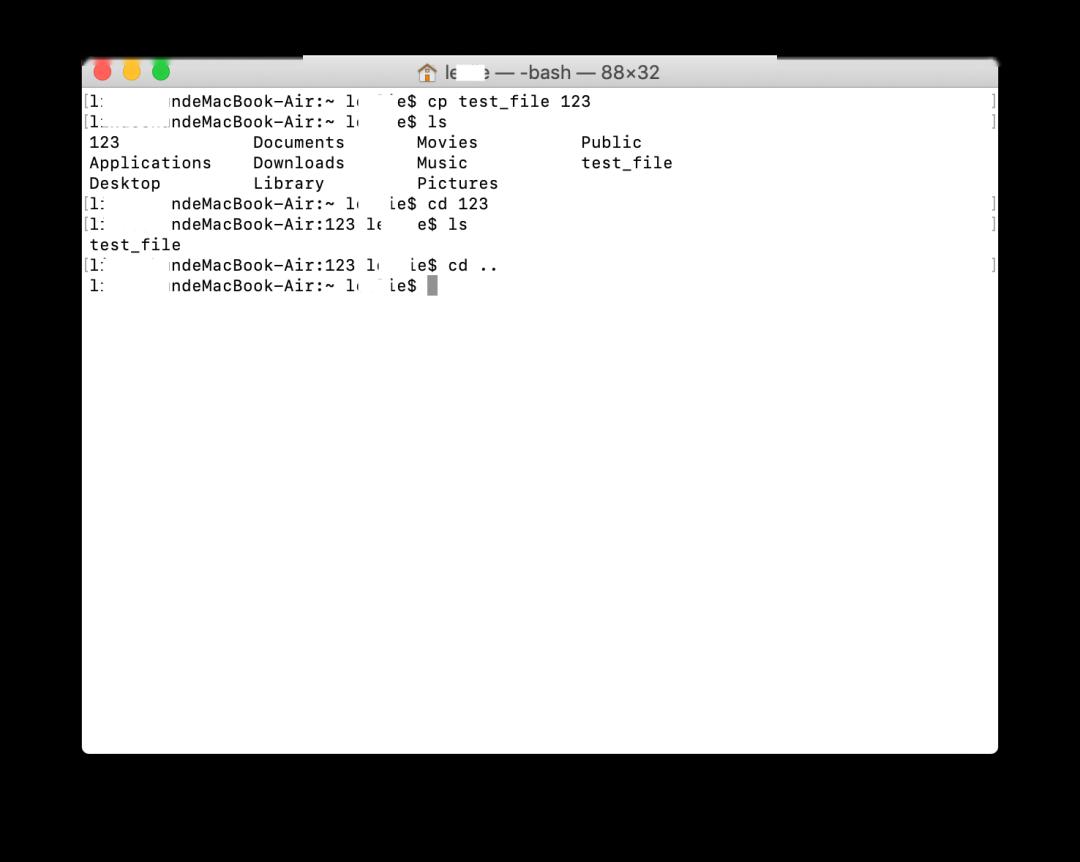
・cp:复制文件或文件夹
cptest_file 123#复制test_file文件到目录123中
・rm:移除文件或整个文件夹需要小心,没有操作可以撤销rm命令!
rmtest_file #移除test_file文件
rm-r123#移除整个目录123
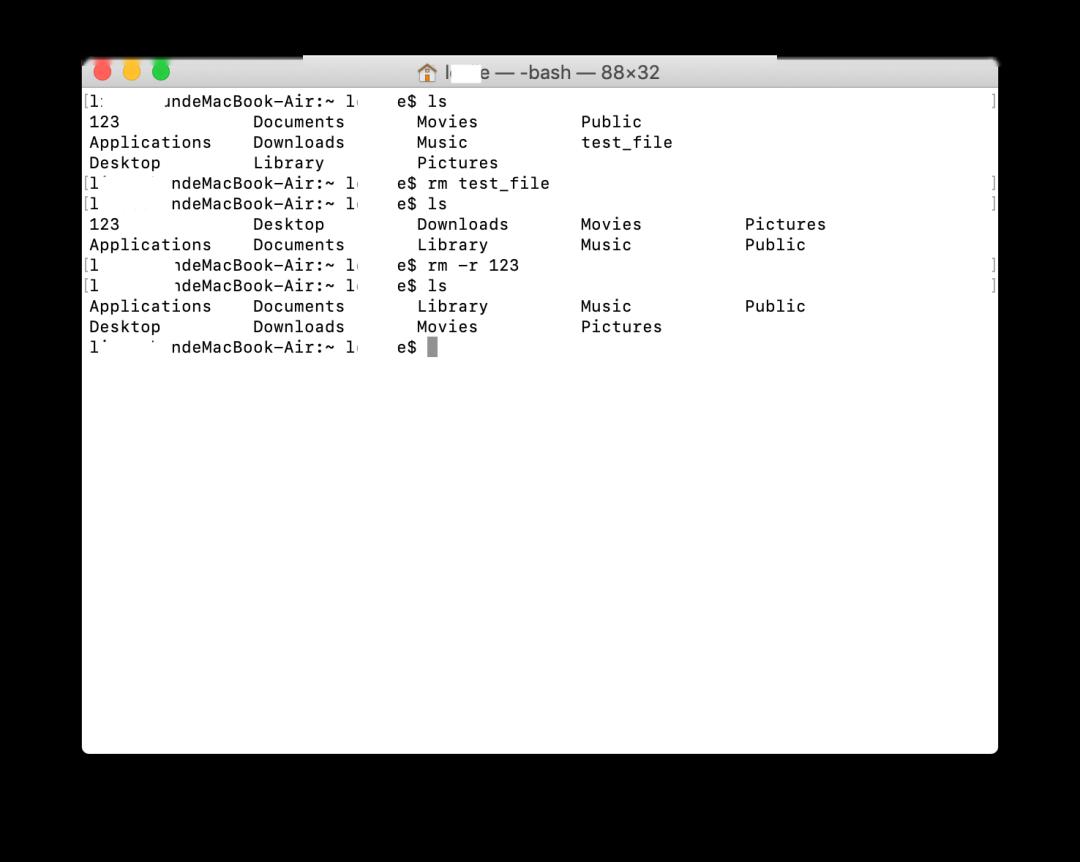
・mv:移动文件或重命名文件
mvnew_file 123#移动new_file文件到目录123中
mvnew_file renamed_file #将new_file重命名为renamed_file
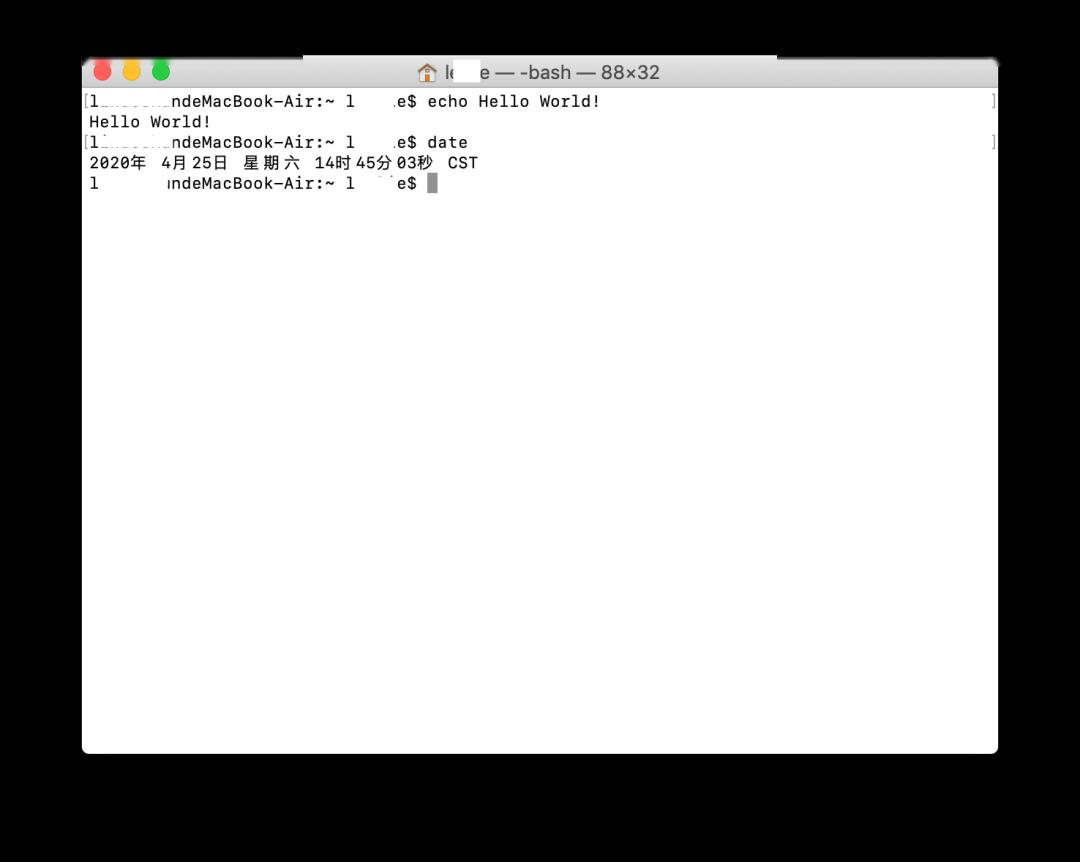
・echo:显示任何输入的参数・date:显示现在时间
echoHello World!
date
Git
Git是一个免费的开源的分布式版本控制系统,帮助控制和管理本地开发项目的各个版本,可用于敏捷高效地处理任何或小或大的项目,大多数的操作都可以通过命令行执行。
在Git网站[https://git-scm.com/]下载相应电脑系统的Git并安装,然后即可使用命令行运行。
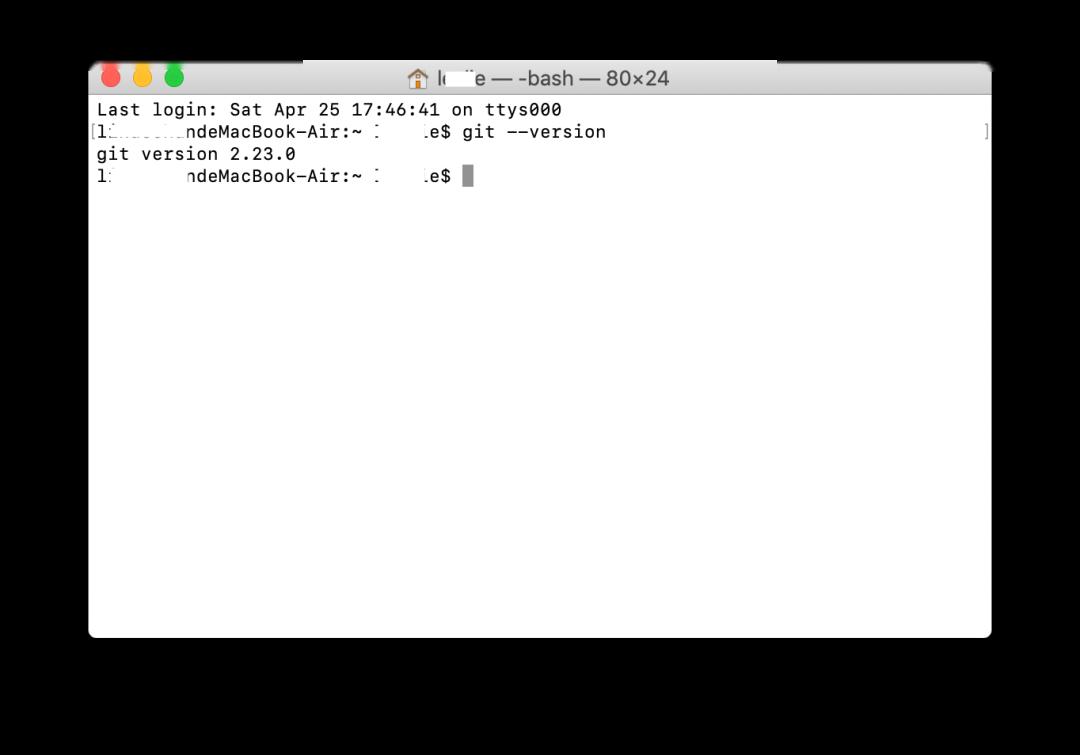
➢查看Git版本信息
git--version
➢设置关联到Git的用户名和邮箱(注意,邮箱与Github注册的邮箱要一致)
gitconfig --globaluser.name "Your Name Here"
gitconfig --globaluser.email "[email protected]"
➢用list命令查看用户信息并用exit命令退出
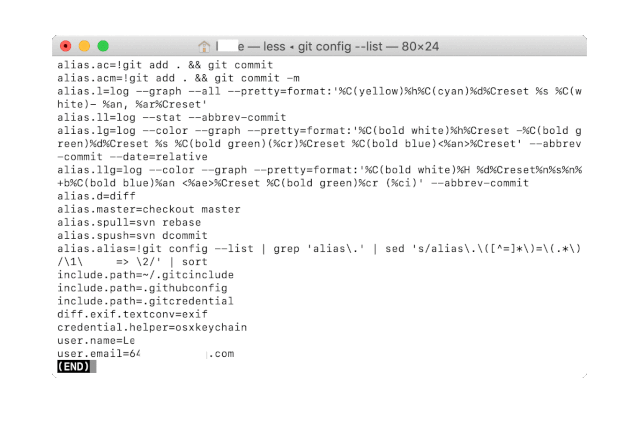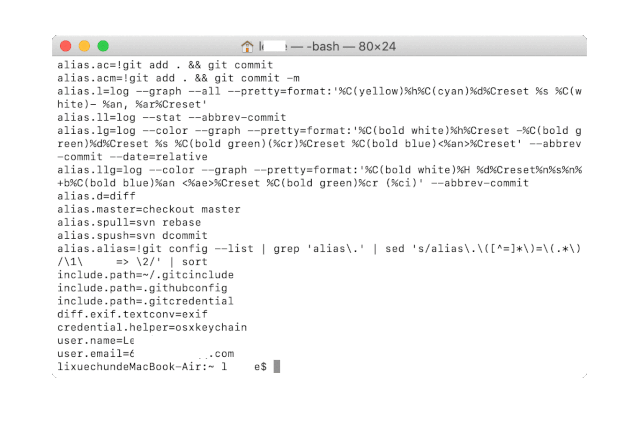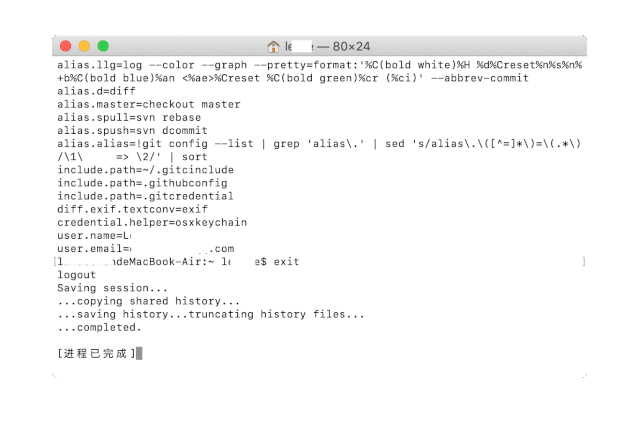
gitconfig --list
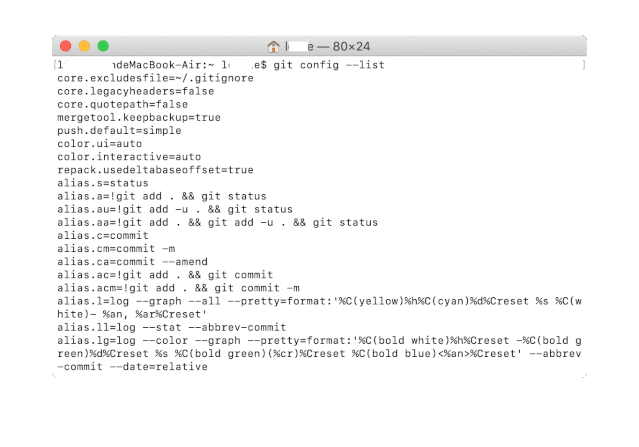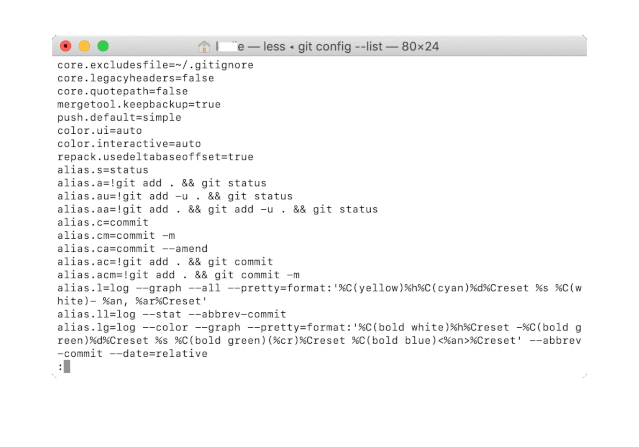
界面出现":"之后按下enter键继续显示信息,直到出现END,此时输入"q"可以返回到命令行输入界面,在"$"后面输入"exit"之后即可退出。
GitHub
Github是通过Git进行版本控制的软件源代码托管服务平台,可以在线开放项目,也能将项目提交到网上方便其他人查阅和开发。
在github网站[https://github.com/join?source=header-home]注册帐号,填写用户名,密码和注册邮箱(要和git的邮箱一致)。
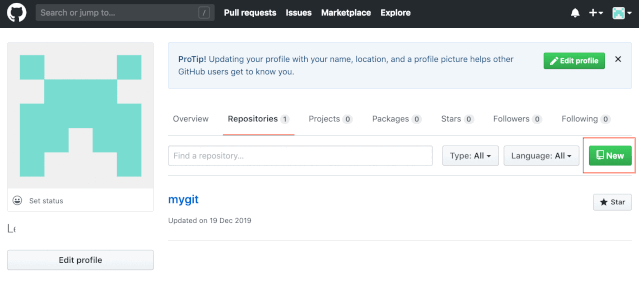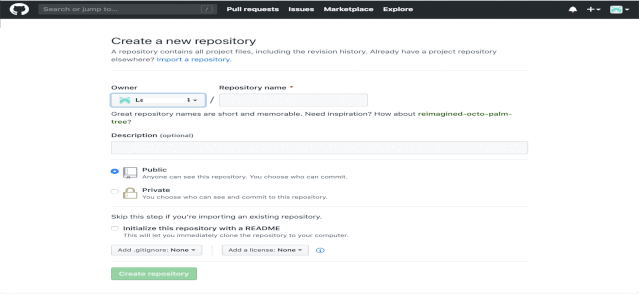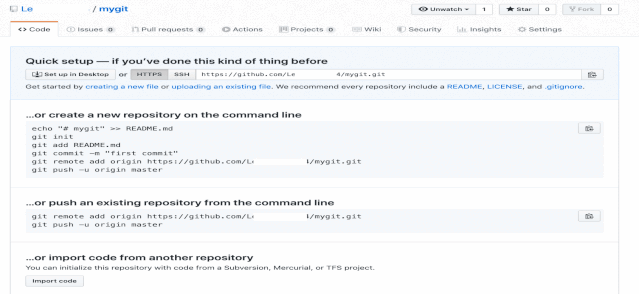
➢在线新建一个资源库(repository/"repo")
・登录github账号后,点击右侧的New按钮打开新建repo页面・命名repo・为repo添加简要的描述・选择public类型的repo(private资源库为付费服务)・选择initialize
this repository with a README・点击Create repository按钮
➢在本地创建资源库拷贝
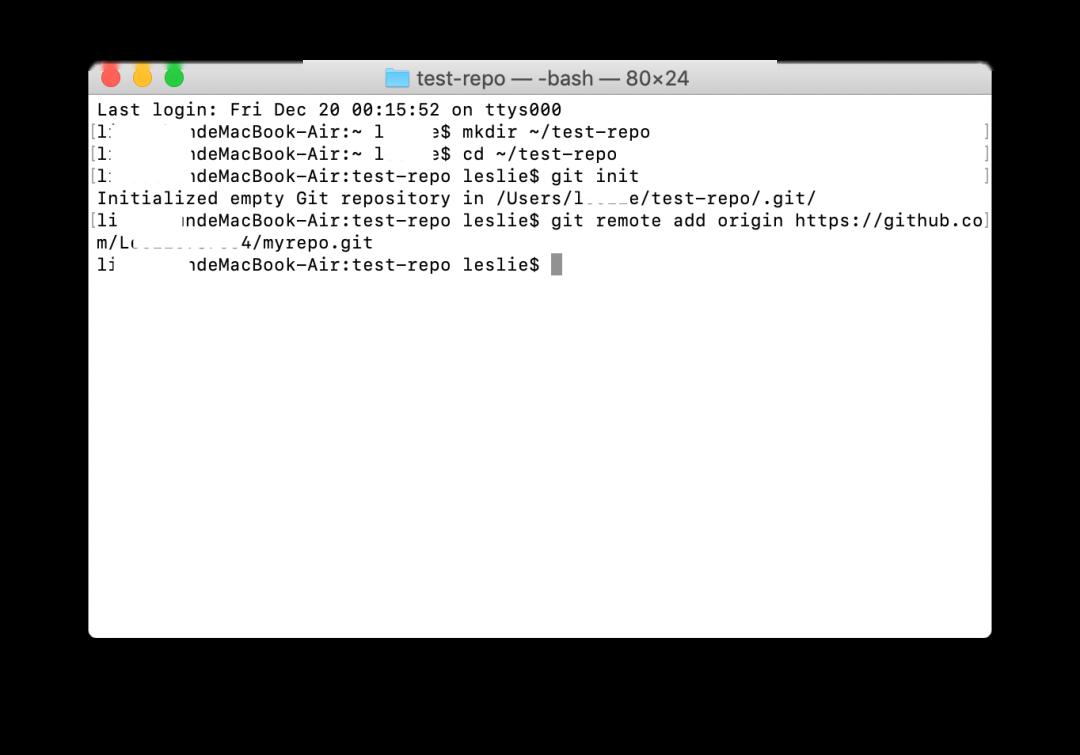
mkdir~/test-repo #在本地创建一个存放拷贝的文件夹
cd~/test-repo #进入新建的文件夹
gitinit #在本地创建一个git资源库
gitremote add origin https://github.com/UserName/repoName.git #把本地的资源库指向远程资源库
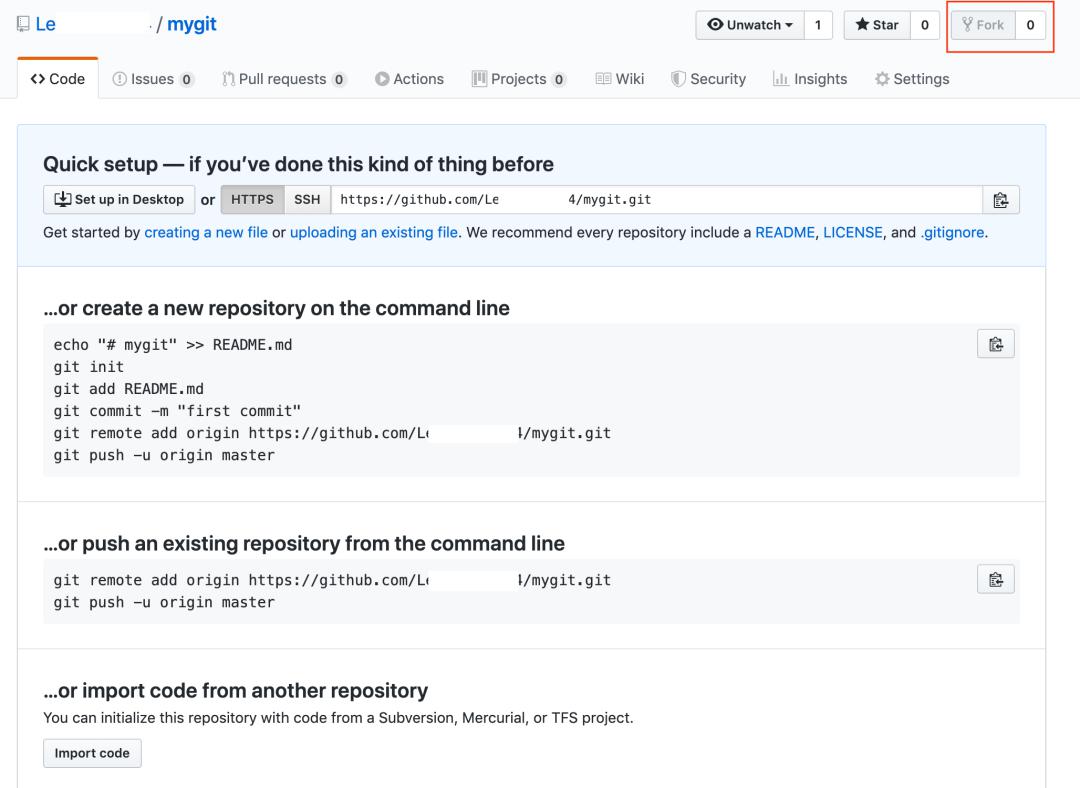
➢拷贝其他人的资源库
・可使用fork在线拷贝其他人的repo到自己的在线资源库
・拷贝到本地
gitclone https://github.com/UserName/repoName.git
➢基本Git命令
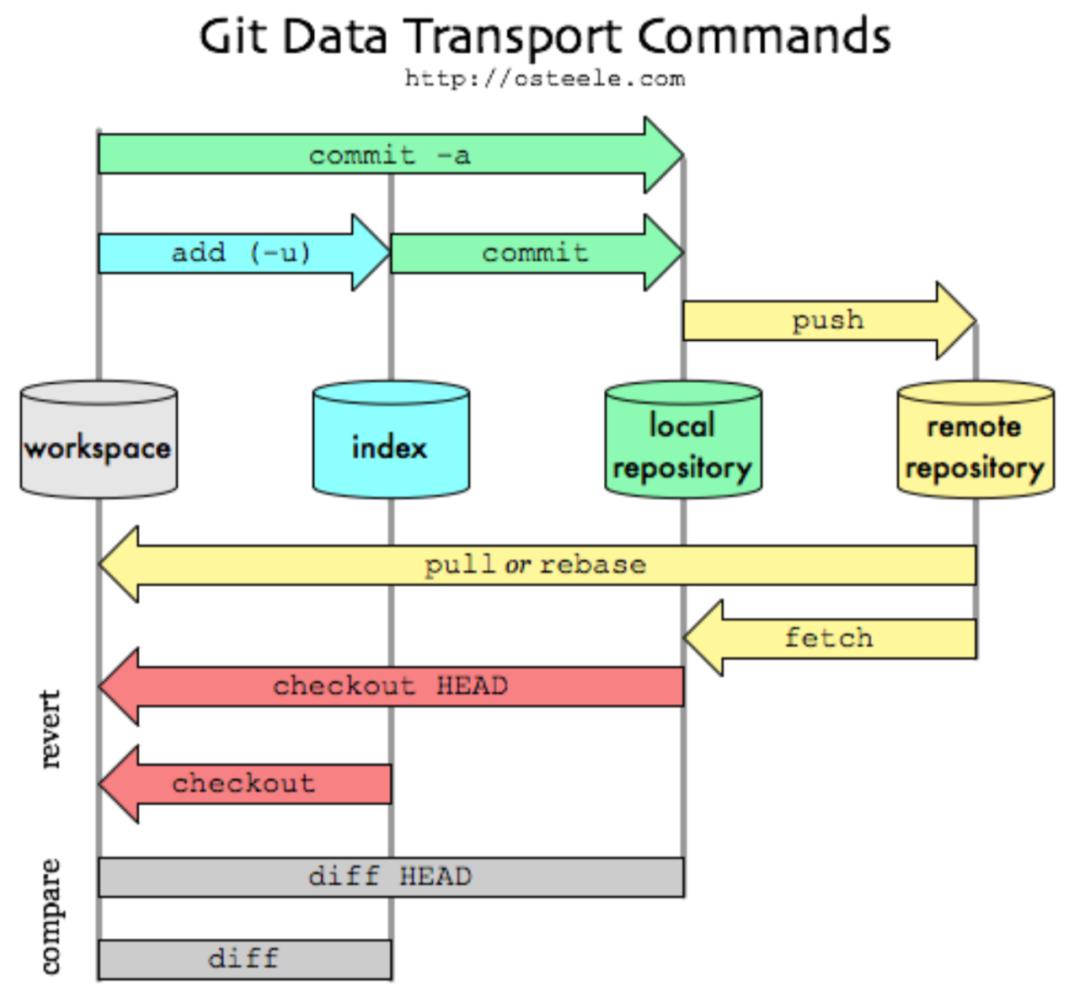
工作区的变化首先通过add命令保存到暂存区index,再通过commit命令传递到本地repo,最后通过push命令上传到远程repo。
add命令
git add. 将工作目录的修改或者新文件保存到暂存区index,但不保存被删除的
git add -u 保存被修改的或删除的文件
git add -A 上述所有变化都将被保存,包括新文件和修改或删除的变化
commit命令
git commit -m"message" 添加信息,描述所做的工作
push命令
git push 将本地提交的修改更新到GitHub 远程repo
分支branch相关命令
git checkout -b branchname 创建一个新的本地分支
git branch 查看本地所有分支
git checkout master 回到主线
git pull origin master 获取远程服务器origin的master分支
Markdown 安装R包
R包可以从CRAN或Bioconductor上下载。
1. CRAN
➢查看CRAN中前三个R包的名称:
方式一:
a<-available.packages#获取CRAN中可用的程序包信息
head( row.names( a), 3)
[ 1] "A3""aaSEA""ABACUS"
方式二:在CRAN网页上查看

➢安装R包:方式一:
install.packages( "slidify")
install.packages( c( "slidify", "ggplot2", "devtools"))
方式二:在Rstudio中安装CRAN上的R包或已下载好的R包在Rstudio的工具栏点击Tools,将打开一个对话框:
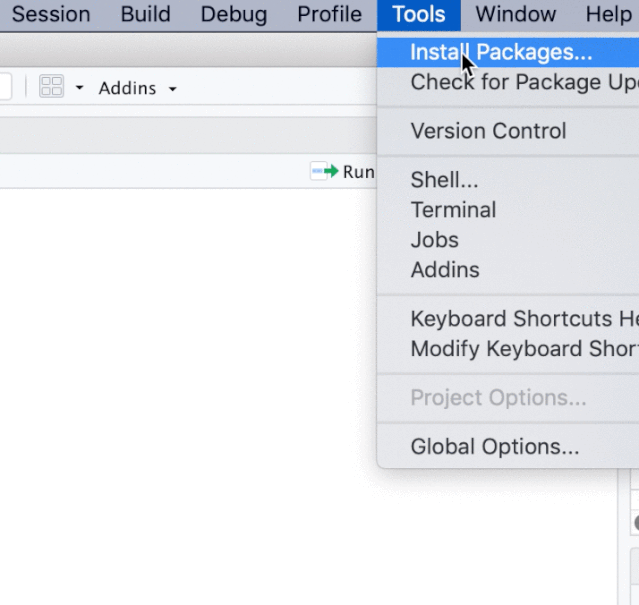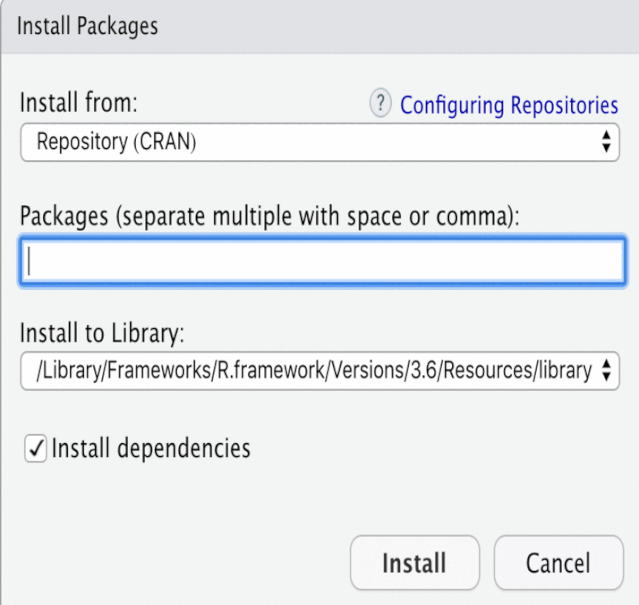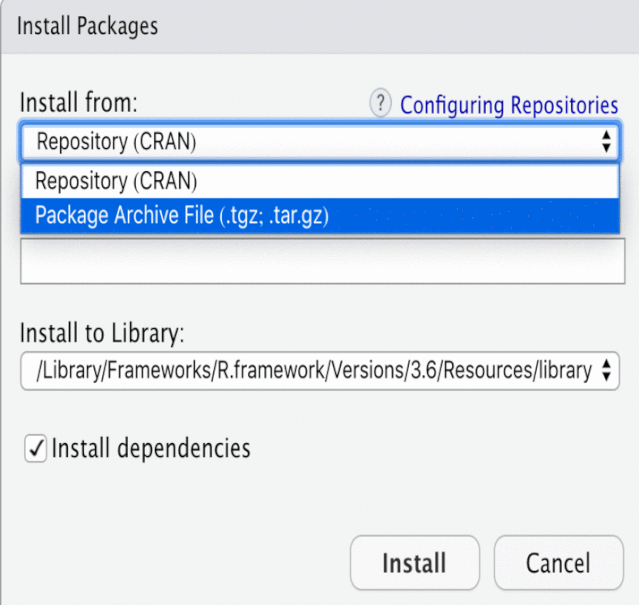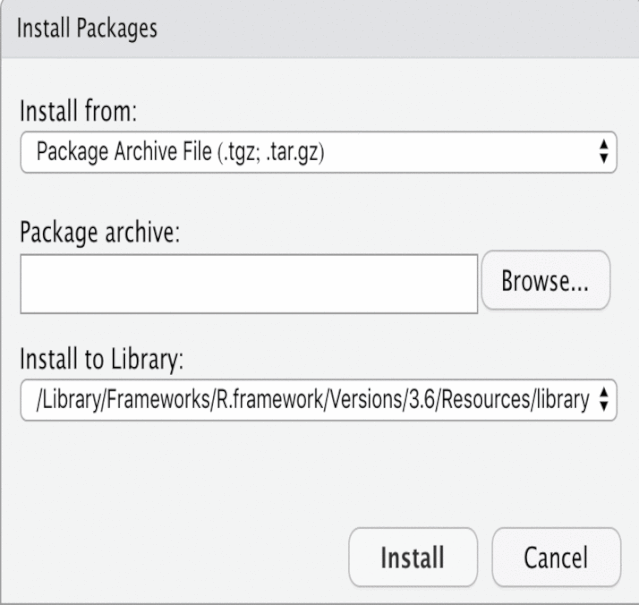
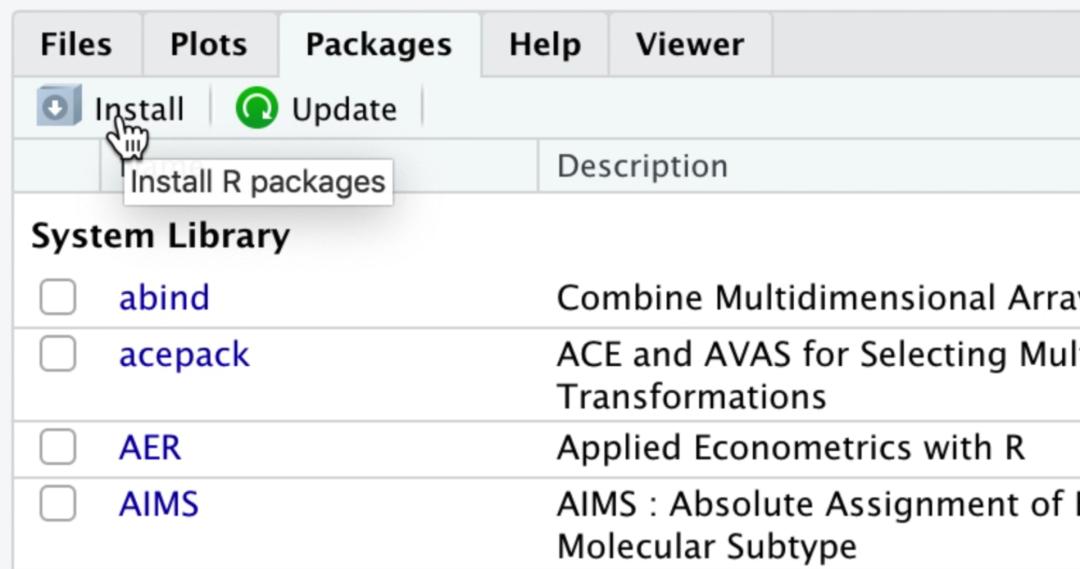
在Rstudio右侧Packages栏点击Install也可以打开同上的对话框:
2. Bioconductor
source( "http://bioconductor.org/bioLite.R")
biocLite#安装Bioconductor的基础版本,包括许多R包
biocLite( c( "GenomicFeatures", "AnnotationDbi"))
上面安装Bioconductor的方法已经过时,安装Bioconductor的R包可以通过BiocManager包。
options( BioC_mirror="http://mirrors.tuna.tsinghua.edu.cn/bioconductor/") #设置R包镜像网站
install.packages( "BiocManager")
BiocManager::install( "GenomicFeatures")
BiocManager::install( c( "GenomicFeatures", "AnnotationDbi"), ask=F, update=F)
3. 加载R包
安装完成后,必须加载R包后才能使用。
library( ggplot2)
search#可以查看刚刚加载的R包的所有函数
[ 1] ".GlobalEnv""package:ggplot2""tools:rstudio""package:stats"
[ 5] "package:graphics""package:grDevices""package:utils""package:datasets"
[ 9] "package:methods""Autoloads""package:base"
4. 获得帮助
・获取函数的帮助文件
? rnorm
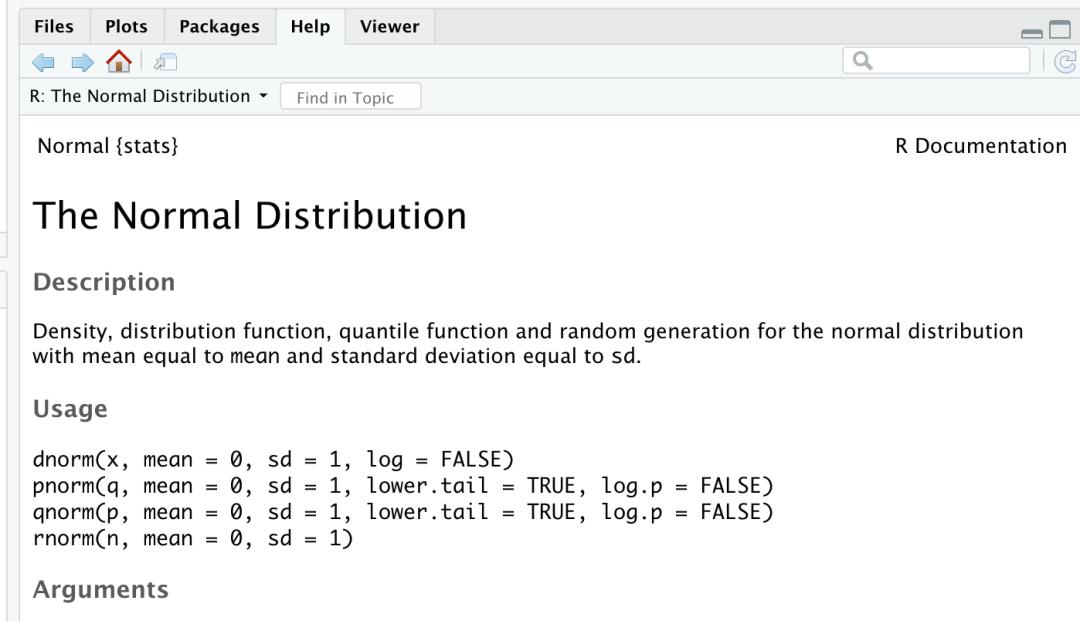
・搜索帮助文件
help.search( "rnorm")
・获取函数参数
args( "rnorm")
function( n, mean=0, sd=1)
NULL
・查看代码
rnorm
function( n, mean=0, sd=1)
.Call( C_rnorm, n, mean, sd)
<bytecode:0x7f8ddbb8e578>
<environment:namespace:stats>安装Rtools
Rtools主要面向windows系统的用户,Rtools是windows系统下编译R包的工具。
install.packages( "devtools")
library( devtools)
find_rtools
[ 1] TRUE#若安装成功将返回TRUE