相信不少小伙伴都遇到过这样的疑惑:收到一份转录组测序结果,辛辛苦苦查找文献,挖掘关键基因信息,眼看一篇高大上的文章即将诞生,却卡在了进行qPCR验证这最后一步:我关注的RNA序列在哪里?没有序列信息我要怎么设计引物做荧光定量PCR呢?
莫慌,我们整理了派森诺各种常规转录测序产品的RNA序列信息,下面就一起来看看吧!
1、 有参转录组
有参考基因组的转录组项目在进行分析时,我们是将测序reads与参考基因组进行比对,根据比对结果进行基因定量分析,进而进行差异分析的。从分析方法就可以发现,有参转录组的基因信息来自参考基因组,因此基因序列也是来自对应的数据库。
那么,我们如何从数据库中查找目标基因序列呢?我们整理了最常规的两大数据库:Ensembl和NCBI的基因序列查找方法,如果您的基因组也是来自这两个数据库,可点击查看
干货 |如何查找目标基因序列?掌握这几招就够了!(Ensembl篇)(点击查看)
干货 | 如何查找目标基因序列?掌握这几招就够了!(NCBI篇)(点击查看)
2、 无参转录组
无参考基因组的转录组项目在进行分析时,我们首先要基于测序结果拼接出一个“基因集”,并对其进行注释,再将测序reads比对到这个基因集上,进而进行表达量分析和表达差异分析。
具体而言,我们首先利用Trinity软件对高质量的测序reads进行从头拼接,得到转录本序列,再对转录本进行聚类,挑选最长的转录本作为Unigene。Unigene的序列存放路径:\report\result\1_RNASeq_Pretreat\Assembly
另外,对于Unigene序列,我们使用Transdecoder软件对其进行ORF(开放阅读框)预测,从而得到CDS和蛋白序列,存放路径:\report\result\6_Structure\ORF
3、 LncRNA
LncRNA为片段长度大于200nt的长链非编码RNA。分析时,我们用Stringtie软件利用Hisat2的比对结果来组装转录本,在将链方向不确定的转录本去除之后,在剩下的组装转录本集合中进行LncRNA的筛选,获得候选LncRNA。然后我们利用三种软件:PLEK、CNCI、Pfamscan,对这些候选LncRNA进行编码潜能预测,三种软件均判定为没有编码潜能的新转录本是高可信度LncRNA,可用于后续分析。
LncRNA序列存放路径:\report\result\LncRNA\1_Novel_LncRNA
4、 CircRNA
CircRNA是一类特殊的非编码RNA分子,与传统的线性RNA(linear
RNA,含5’和3’末端)不同,CircRNA分子呈封闭环状结构,其首尾相连的“接头”位置的序列由于来自相隔较远的外显子区域,直接比对基因组时存在大片段的缺口。利用这一特点,我们鉴定CircRNA时首先筛选未比对上参考基因组的测序reads,再截取两端20bp作为Anchors序列再次比对到基因组上,用于检测CircRNA。
CircRNA序列存放路径:\report\result\CircRNA\1_CircRNA_Prediction
5、 miRNA
miRNA是一类长度在18-36nt之间的短链非编码RNA,由于其序列较短,我们对它的获取与鉴定是一般是基于一些miRNA数据库,如miRBase进行的。
miRBase (http://www.mirbase.org/)是由曼彻斯特大学的研究人员开发的一个在线的miRNA数据库,该数据库中收录了来自200多个物种、接近4万个miRNA的信息,是最全面的miRNA数据库。
对于被miRBase收录的物种,我们分析时会下载该物种miRNA的前体和成熟体序列,然后将去重后的测序reads与其进行比对,从而对测到的miRNA进行注释。对于这种miRNA测序项目,最简单的获取序列方式就是从miRBase中下载所有物种的miRNA序列,从中搜索目标miRNA的ID即可:

对于未被miRBase收录的物种,我们将去重后的测序reads与数据库中所有动物(或植物)的成熟miRNA序列进行比对,从中筛选保守miRNA。
得到的保守miRNA序列存放路径:\Results\03_Annotation\miRNA

看到这里各位可能已经发现了,无论是哪一种RNA,似乎所有的序列文件都是以.fasta或.fa为后缀的,这是什么格式呢?我们应该用什么软件打开呢?
在生物信息学中,FASTA格式(又称为Pearson格式),是一种基于文本用于表示核苷酸序列或氨基酸序列的格式,可用文本编辑软件打开,如UltraEdit
、EditPlus等软件,系统自带的记事本(txt)工具也可以。然后利用搜索功能查找序列ID,就可以轻松找到对应的序列啦!
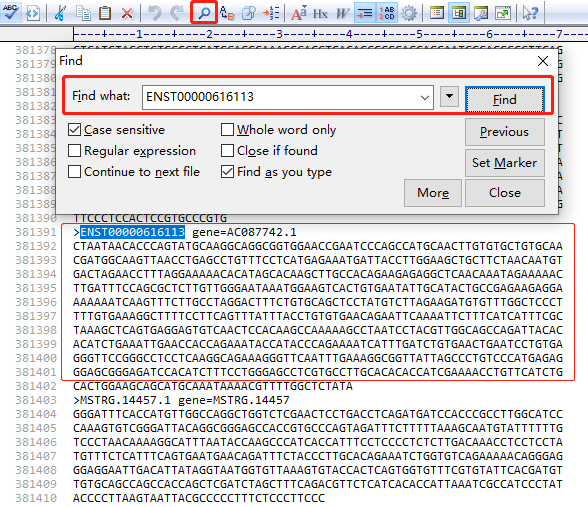
Ediplus搜索界面
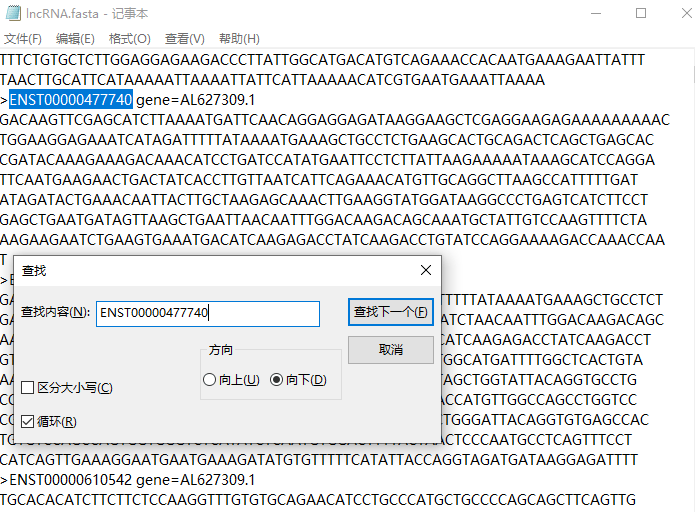
记事本搜索界面
转自:派森诺