Graphlan可视化进化树
撰文:文涛 南京农业的大学
责编:刘永鑫 中科院遗传发育所
GraPhlAn简介
总体来讲,Graphlan是一个可视化进化树和基于分类等级绘制层级分类树的工具。可以制作分类树是它不同于R包ggtree和iTOL的地方。然就进化树而言,iTOL功能最为全面,ggtree最容易上手,从功能上来讲graphlan很多地方不如iTOL,比如,iTOL无限制添加的数据集,外环可以制作各种图形包括箱线图等,可以使用多种符号填充外环,但是Graphlan就不能这么随便了,只能使用两个符号填充外环。再多的环属性也就是设置环数量和颜色,透明度了。ggtree最容易上手,但是就一张圈图来说,它不能添加除了热图以外的其他图形,但是在非圈图的模式下,可以对多种数据进行合并,方法将更为简单,操作也容易一些。
素颜vs美化
下面通过我们的实战,将这个原始的图形通过graphlan大法,改变到下面的样子
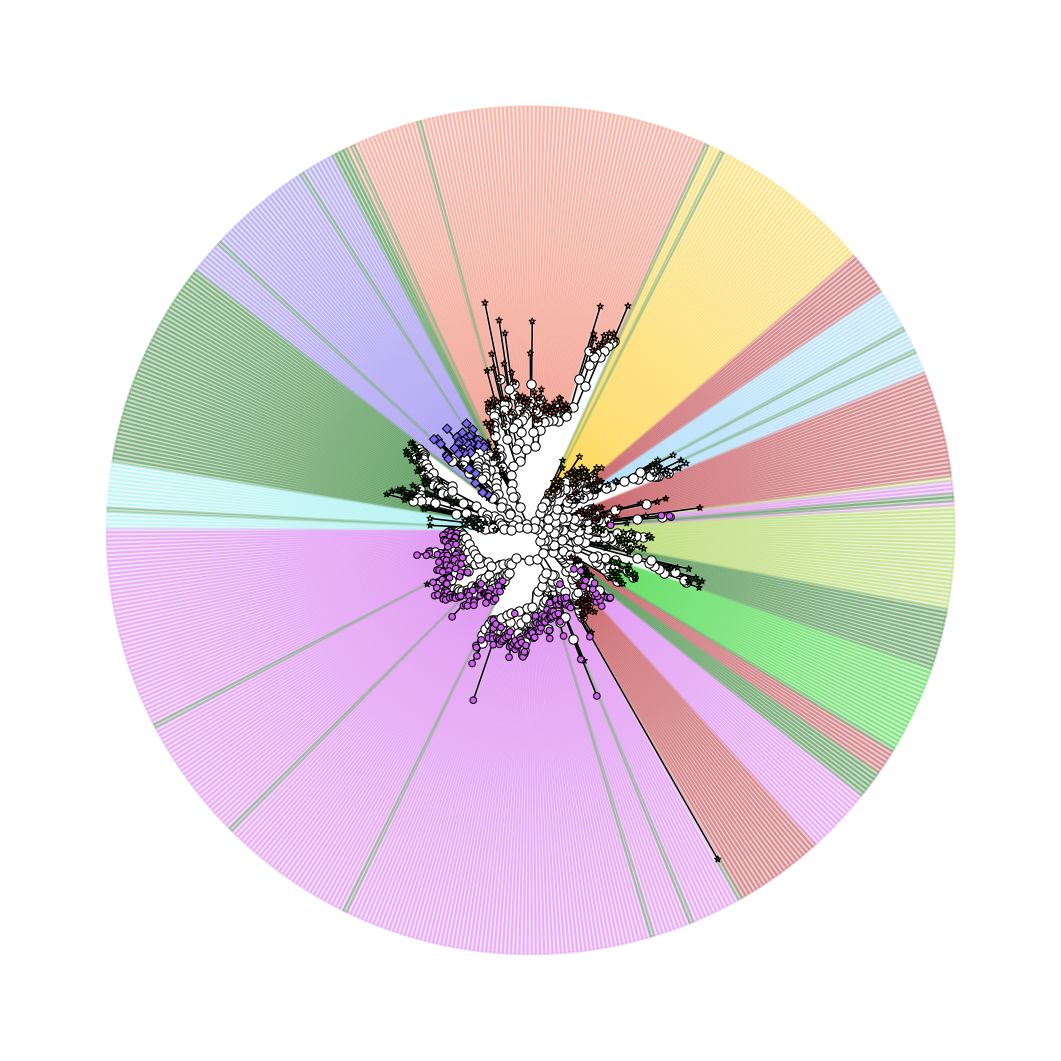
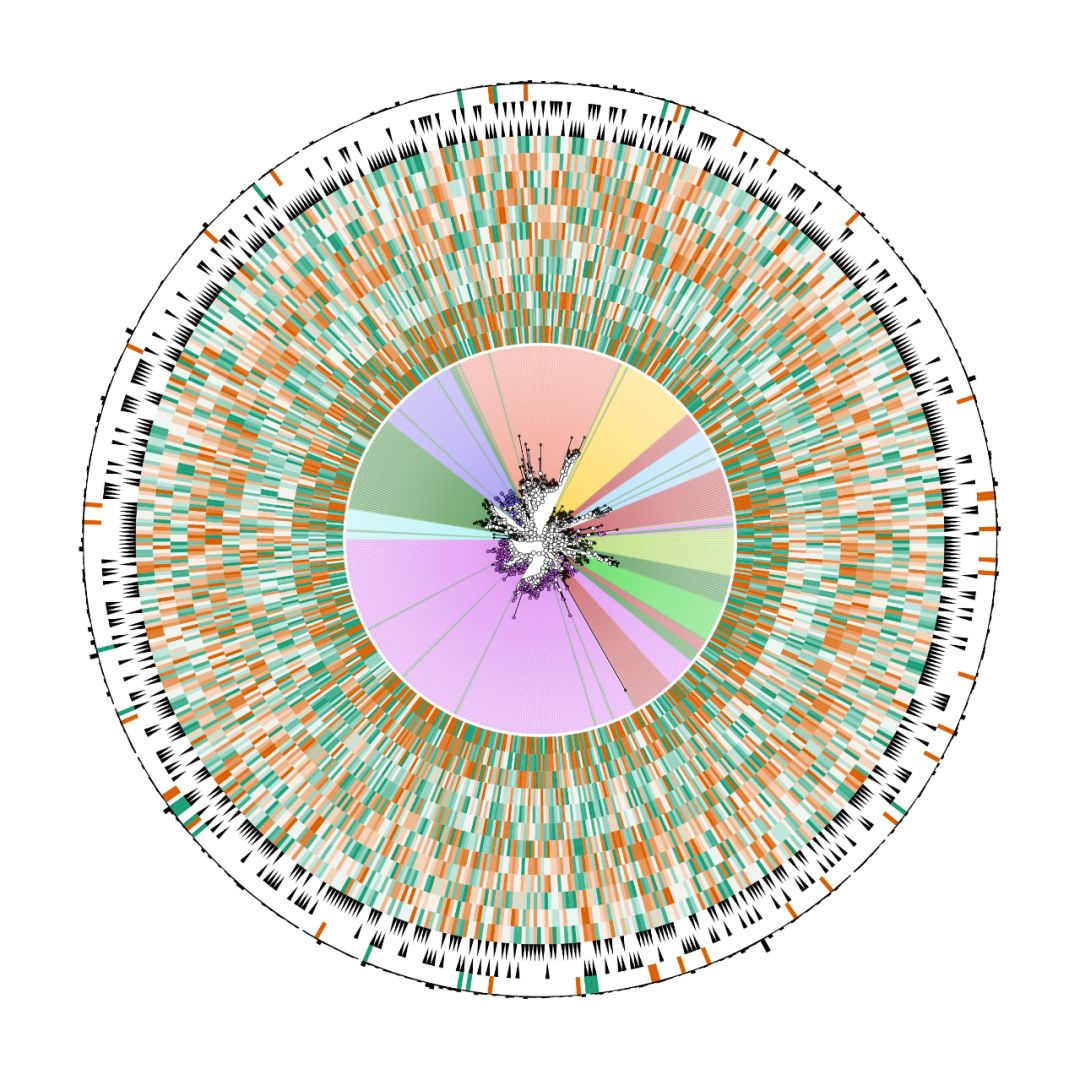
完全认不出来了吧,这就是美颜的重要性。
认识软件参数
参数虽多,你也不需要记住,只要认识即可,而且都是字面意思,懂英文看一遍全懂了,需要时可以来查。大多时间我们是在别人基础上修改,能看懂代码即可。
熟悉grahlan注释参数-全局参数,
一般全局参数使用方式为:参数名 + 参数值
分支控制,一般不做控制 branch_bracket_depth 0##控制一个分支块的聚集程度,值越大,树枝越集中
branch_bracket_width 0 0###控制一个分支点延伸出来的分支线的长度,设定大了会反向延长超多分支点。
annotation_background_alpha :只能设置全部的阴影的透明度 组合命令控制注释阴影的半径
一下两个指标设置为负值,且两者均设置为-0.7,会大大减小注阴影半径
annotation_background_separation -0.40
annotation_background_offset -0.34 一般全局参数 title:设置图片题目
title_font_size:60 题目字体大小
total_plotted_degrees 350:这个设置整个圆圈的展看角度,不会设置超过360度,一般设置一个比360度小一点的值是为了添加标签使用(例如:对环添加标签ring_label)。
start_rotation 270:对圆圈图进行旋转的起始位置;注意图形下方为270°,左方为0°。
class_legend_font_size 图例大小设置
## 全局注释
annotation_background_alpha 0.02:全部注释背景透明度;
annotation_background_width 0.03 注释背景宽度
annotation_font_size:设置注释字体大小
annotation_legend_font_size 设置图上容不下时,代表字母的对应注释的字体大小
branch_thickness:全局树枝的粗细设置
branch_color :设置全局树枝颜色
ignore_branch_len 10:控制全局树伸展和大小
clade_marker_size:全局的点的尺寸
clade_marker_edge_width:全局的点的边缘宽度
clade_separation 0.5 进化树分枝簇的紧凑程度
ring_internal_separator_thickness 0.01 设置环与环之间的厚度
ring_separator_color :设置环间隔的颜色,结合厚度设置选项增多
ring_width:设置在这个环上每个点所占有的宽度;
ring_height:设置环与环之间的距离,默认环与环之间的距离为1;
ring_shape:对分类树外面每层的空间设置形状填充,有不多的中形状可以选择。v:正v字型;^:v字型;
ring_alpha 0.01:这是对每个叶节点占据的位置不透明度,最大值为1。值越高颜色越深,当然需要结合设置颜色ring_color
ring_color:每条环的颜色
设置全局的环参数,必须建立在叶节点对应的shape上,不可只设置全局参数,尤其是全局ring color,仅仅设置该参数是无法进行全环上色的。默认节点shape是R,也就是整个叶节点对应的方块。
节点基本参数
节点参数使用方式:节点名称 + 参数名称 + 参数值
#节点基本参数
clade_marker_size 节点的大小,从1到200都可以设置
clade_marker_color:颜色:默认无色,r:红色;b:蓝色(基本为颜色首字母)
clade_marker_shape:形状:默认圆形,*:代表五角星,h:代表六边形
clade_marker_label:设置点内部写一个标签,后面跟上标签内容
clade_marker_font_color:设置点内标签的字体颜色
clade_marker_edge_color #555555 控制节点的颜色
clade_marker_edge_width 1.2#节点的边的宽度 节点对应的形状
这些标签有我们熟悉的点,圈,各个方向的三角,五边形,五角星,钻石等。具体符号和对应名称如下:
'.' : point marker
',' : pixel marker
'o' : circle marker
v' : triangle_down marker
'^' : triangle_up marker
'<' : triangle_left marker
'>' : triangle_right marker
'1' : tri_down marker
'2' : tri_up marker
'3' : tri_left marker
'4' : tri_right marker
's' : square marker
'p' : pentagon marker
'*' : star marker
'h' : hexagon1 marker
'H' : hexagon2 marker
'+' : plus marker
'x' : x marker
'D' : diamond marker
'd' : thin_diamond marker
'|' : vline marker
'_' : hline marker 节点注释 #节点注释
annotation_background_color
annotation_background_alpha 0.02:注释背景透明度;
annotation_background_width 0.03 注释背景宽度
annotation_font_size:设置注释字体大小
annotation_legend_font_size设置图上容不下,设置成代表字母的对应注释的注释字体大小 枝条参数 branch_thickness 枝条的厚度
branch_color 枝条颜色
branch_color_from_ancestor:使用上级分支的颜色 环参数 ring_internal_separator_thickness 0.01 设置环与环之间的厚度
ring_separator_color :设置环间隔的颜色,结合厚度设置选项增多
ring_width:设置在这个环上每个点所占有的宽度;
ring_heigh:设置环与环之间的距离,默认环与环之间的距离为1;
ring_shape:对分类树外面每层的空间设置形状填充,有不多的形状可以选择。v:正v字型;^:^字型;
ring_alpha 0.01:这是对每个叶节点占据的位置不透明度,最大值为1。值越高颜色越深,当然需要结合设置颜色ring_color
ring_color:每条环的颜色 进化树实战
使用材料
我们使用已经做好的16s序列构建的进化树(由Fastree生成,一般为rep_set.tree)
对应OTU的丰度表格和注释文件,分别为otu_table.txt和rep_seqs_tax.txt,有时可能两者存于同一文件内;
实验设计,即样本对应属性,如分组信息(可选)。
分析要求
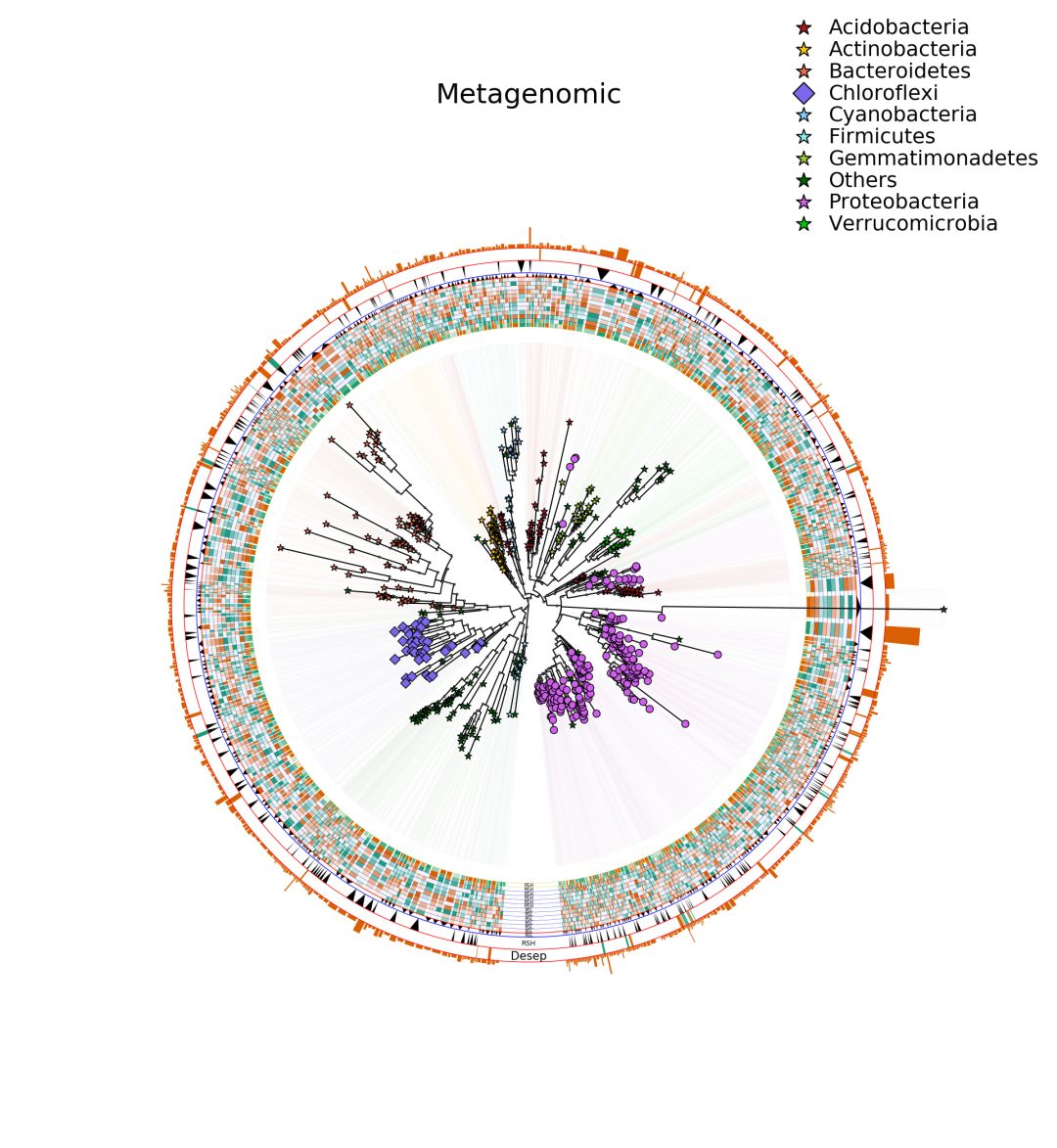
做一张进化树,并将不同门的OTU节点标注不同的颜色和形状
对不同的门的OTU添加不同颜色的阴影(低透明度颜值高)。
根据每个样品的OTU丰度使用不同颜色,添加外环。
全部样品分为两个组,根据OTU在两个组中的丰度信息添加不同的标签环
进行差异分析,添加外环对差异OTU根据其分组进行上色并标注。
将OTU总体丰度通过最外环高度展示出来。
构建OTU丰度和物种文件说明
上面我们见过软件的参数,每个节点都要注释,如果手动搞,可能真要3天至半个月画个美颜树。只要是批量操作且有规律,我们就可以用编程来实现。
使用R语言构造注释文件,这里我们明确,基于GraPhlAn的注释文件按照注释占有列数量我分为一下三个部分:
占两列的注释:题目,全局参数,例如,全局点和枝参数
占三列注释:叶,枝参数,全局环参数。图例参数
占四列参数:环参数
由于注释文件编写耗费很大时间,Excel适合编写的也仅仅只有占两列的数据,和总环的参数。这部分编写完成之后每次绘图基本只根据不同的实验,会小范围修改。参考模板:annot1.txt
首先根据之前要求,准备要处理文件
并确定工作目录包括所需的OTU表、物种注释、实验设计等文件,文件名与代码中不对应请自行修改。
测试数据和代码下载链接,后台回复“graphlan”获取
准备需要展示的数据
OTU表和实验设计的读取、交叉筛选和标准化
# 读取OTU表
otu = read.table("otu_table.txt", sep="t",row.names= 1,header=T, check.names=F)
head(otu)
# 读入实验设计
design = read.table("metadata.txt", header=T, row.names= 1, sep="t")
head(design, n=12L)
# 交叉筛选,保持实验设计和OTU中样本对应
idx = rownames(design) %in% colnames(otu)
sub_design = design[idx,]
# 过滤OTU表
OTU = otu[,rownames(design)]
head(OTU)
OTU = as.matrix(OTU)
# 原始reads count表标准化
norm = t(t(OTU)/colSums(OTU,na=T)) #* 100 可 normalization to total 100
# 转置并转换为数据框
library(dplyr)
norm1 = norm %>% t %>% as.data.frame
norm1[1:3,1:3]
计算分组均值
# 数据分组计算平均值
otutab.split = split(norm1,as.factor(sub_design$SampleType_RS))
otutab.apply = lapply(otutab.split,function(x)colSums(x))
norm2 = do.call(rbind,otutab.apply) %>% t
# OTU表后面添加分组均值
OTU_all= cbind(as.data.frame(norm),norm2)
head(OTU_all)
追加物种注释
# 读取OTU对应的物种注释
# 9列,分别为ID,界门纲目科属种和置信度
tax = read.delim("rep_seqs_tax.txt", sep="t",row.names= 1,header=F, check.names=F)
# 只保留物种7级分类
tax = tax[1:7]
head(tax)
# 添加分类级列名:界门纲目科属种
colnames(tax) =c("kingdom","phylum","class","order","family","genus","species")
# OTU表再添加物种注释
index = merge(OTU_all,tax, by="row.names",all=F)
row.names(index) = index$Row.names
index$Row.names = NULL
head(index)
上面,我们制作OTU对应丰度、平均丰度和物种注释的数据
映射丰度至颜色
# 设定颜色梯度,橙-白-绿三色过滤
colorg = colorRampPalette(c( "#D95F02", "white","#1B9E77"))(12)
# 通用绘图缩放方法 Generic plot scaling methods
library("scales")
# 显示颜色和对应16进制RGB代码
show_col(colorg)
# 建立12种颜色的数据框
c = data.frame(id = c(1:12),col = colorg)
# 提取前面OTU中12个样品的丰度部分
a = index[1:12]
for(i in 1:nrow(a)){
a[i,1:12] = match(colnames(a[i,1:12]), colnames(sort(a[i,1:12])))
}
# 填充颜色:替换丰度顺序为对应的颜色
for(i in 1:nrow(a)){
aa = a[i,]
aa = as.data.frame(aa)
colnames(aa) = aa[1,]
ccc =t(c[colnames(aa),])
a[i,] = ccc[2,]
}
head(a)
out = cbind(index,a)
head(out)
映射分组为开关
##组比对映射形状
HH = c(rep("A",nrow(out)))
LL = c(rep("A",nrow(out)))
# 两组比较,大的标为v,小的为空
for(i in 1:nrow(out)){
if(out[i,13] > out[i,14]){
HH[i] ="v"
LL[i] =" "
}else if(out[i,13] == out[i,14]){
HH[i] =" "
LL[i] =" "
}else if(out[i,13] < out[i,14]){
HH[i] =" "
LL[i] ="^"
}
}
# 继续追加至表末
out2 = cbind(out,HH,LL)
head(out2)
差异分析
###下一步添加差异分析结果
# 安装bioconductor包安装工具
# if (!requireNamespace("BiocManager", quietly = TRUE))
# install.packages("BiocManager")
# 安装差异比较包DESeq2
# BiocManager::install("DESeq2", version = "3.8")
# BiocManager::install("GenomeInfoDb", version = "3.8")
# BiocManager::install("S4Vctors", version = "3.8")
library(DESeq2)
library(limma)
# BiocManager::install("pasilla", version = "3.8")
library(pasilla)
# library(DESeq)
head(OTU )
count = OTU
count=as.matrix(count)
#######首先比较发病和健康中的差异#############
# 读入矩阵和实验设计,指定分类
dds = DESeqDataSetFromMatrix(countData = count,
colData = sub_design,
design = ~ SampleType_RS)
dds2 = DESeq(dds)
##第二步,标准化
resultsNames(dds2)
# 将结果用results函数来获取,赋值给res变量
res = results(dds2, contrast=c("SampleType_RS","BL", "BH"),alpha=0.05)
# summary一下,看一下结果的概要信息
summary(res)
# 按校正P值和差异倍数,添加颜色
res$level = as.factor(ifelse(res$padj < 0.05 &
res$log2FoldChange > 1, "#D95F02",ifelse(res$padj < 0.05 &
res$log2FoldChange < -1, "#1B9E77"," ")))
head(res)
# 提取筛选的显著差异,添加至otu表
res1 = as.data.frame(res[ncol(res)])
OTU_TAX_all = merge(out2,res1, by="row.names",all=F)
head(OTU_TAX_all)
dim(OTU_TAX_all)
row.names(OTU_TAX_all) = OTU_TAX_all$Row.names
OTU_TAX_all$Row.names = NULL 构建三列注释文件,主要是节点注释 ##构建进化树节点属性注释文件
# head(OTU_TAX_all)
pcol = c(rep("A",nrow(OTU_TAX_all)))
psha = c(rep("A",nrow(OTU_TAX_all)))
# 按门水平手动设置颜色和形状
for(i in 1:nrow(OTU_TAX_all)){
if(OTU_TAX_all[i,16] == "p__Acidobacteria"){
pcol[i] ="#B0171F"
psha[i] ="*"
}else if(OTU_TAX_all[i,16] == "p__Proteobacteria"){
pcol[i] ="#D15FEE"
psha[i] ="o"
}else if(OTU_TAX_all[i,16] == "p__Acidobacteria"){
pcol[i] ="#B0171F"
psha[i] ="*"
}else if(OTU_TAX_all[i,16] == "p__Verrucomicrobia"){
pcol[i] ="#00CD00"
psha[i] ="*"
}else if(OTU_TAX_all[i,16] == "p__Cyanobacteria"){
pcol[i] ="#87CEFA"
psha[i] ="*"
}else if(OTU_TAX_all[i,16] == "p__Actinobacteria"){
pcol[i] ="#FEC80A"
psha[i] ="*"
}else if(OTU_TAX_all[i,16] == "p__Bacteroidetes"){
pcol[i] ="#EE6A50"
psha[i] ="*"
}else if(OTU_TAX_all[i,16] == "p__Chloroflexi"){
pcol[i] ="#7B68EE"
psha[i] ="D"
}else if(OTU_TAX_all[i,16] == "p__Gemmatimonadetes"){
pcol[i] ="#9ACD32"
psha[i] ="*"
}else if(OTU_TAX_all[i,16] == "p__Firmicutes"){
pcol[i] ="#8DEEEE"
psha[i] ="*"
}else {
pcol[i] ="#006400"
psha[i] ="*"
}
}
# OTUID对应物种颜色形状数据框
ano4 = cbind(row.names(OTU_TAX_all),pcol,psha)
# head(ano4)
ano4 = as.data.frame(ano4)
生成三列的graphlan要求结点注释文件
# 节点颜色映射
ste1 = data.frame(a = ano4$V1,b = c(rep("clade_marker_color",nrow(OTU_TAX_all))),c = ano4$pcol)
head(ste1)
# 节点形状映射
ste2 = data.frame(a = ano4$V1,b = c(rep("clade_marker_shape",nrow(OTU_TAX_all))),c = ano4$psha)
head(ste2)
# 节点阴影映射颜色,同节点颜色
ste3 = data.frame(a = ano4$V1,b = c(rep("annotation_background_color",nrow(OTU_TAX_all))),c = ano4$pcol)
head(ste3)
# 注释透明度,淡色,如0.1透明
ste4 = data.frame(a = ano4$V1,b = c(rep("annotation_background_alpha",nrow(OTU_TAX_all))),c = c(rep(0.1,nrow(OTU_TAX_all))))
head(ste4)
ste4$c = as.factor(ste4$c)
# 节点大小,这里统一写10,LEfSe中按丰度大小变化
ste5 = data.frame(a = ano4$V1,b = c(rep("clade_marker_size",nrow(OTU_TAX_all))),c = c(rep(10,nrow(OTU_TAX_all))))
head(ste5)
ste5$c = as.factor(ste5$c)
# 保存文件
ste_all = rbind(ste1,ste2,ste3,ste4,ste5)
head(ste_all)
# 保存统计结果,有waring正常
write.table(ste_all, "annot2.txt", append = F, quote = F, sep="t",
eol = "n", na = "NA", dec = ".", row.names = F, col.names = F)
构建四列注释文件,主要是环注释 # 构建单样品丰度映射文件,共12列
# 提取之前按丰度构建的颜色矩阵
anno1 = OTU_TAX_all[22:33]
head(anno1)
# 设置样本对应环编号
colnames(anno1 ) = c(1:3,7:12,4:6)
anno1$id = row.names(anno1)
library("reshape2")
# 构建3列文件,ID,环编号,对应颜色
anno11 = melt(anno1,id.vars = "id",variable.name = "ring",value.name = "color")
# 添加第4列,类型
anno11$ringcolor = c(rep("ring_color",nrow(anno11)))
# 调整列的序列,前两列为ID和类型,其它在后面
anno11 = select(anno11,id,ringcolor,everything)
head(anno11) ##构建平均丰度标志映射文件
anno2 = data.frame(OTU_TAX_all$LL,OTU_TAX_all$HH,row.names = row.names(OTU_TAX_all))
head(anno2)
# 位置为13/14圈
colnames(anno2 ) = c(13,14)
anno2$id = row.names(anno2)
anno22 = melt(anno2,id.vars = "id",variable.name = "ring",value.name = "shape")
anno22$ringshape = c(rep("ring_shape",nrow(anno22)))
anno22 = select(anno22,id,ringshape,everything)
# 只保留有差异,即有形状标记的行
anno222 = anno22[anno22$shape %in% c("^","v"),]
head(anno222) ##构建差异分析颜色标记文件
anno3 = data.frame(id = row.names(OTU_TAX_all),
ringcolor = c(rep("ring_color",nrow(OTU_TAX_all))),
ring =c(rep(15,nrow(OTU_TAX_all))),color = OTU_TAX_all$level )
anno3$ring = as.factor(anno3$ring)
head(anno3)
anno33 = anno3[anno3$color %in% c("#D95F02","#1B9E77"),] # 合并1,3批的颜色注释
head(anno11);dim(anno11)
ann_all = rbind(anno11,anno33)
# 第二批为形状,需改列名再合并
colnames(anno222) = colnames(ann_all)
ann_all2 = rbind(ann_all,anno222)
head(ann_all2) # 添加物种丰度总体特征
# 计算所有样品的均值
norm$mean = apply(norm,1,mean)
head(norm)
# 计算平方根,标准化数据更均匀
norm$mean = sqrt(norm$mean)
norm$mean = sqrt(norm$mean)
# 第16列行高度展示丰度的4次方根
ste6 = data.frame(a = ano4$V1,b = c(rep("ring_height",nrow(OTU_TAX_all))),c = c(rep(16,nrow(OTU_TAX_all))),d = norm$mean)
head(ste6)
ste6$c = as.factor(ste6$c) # 统一列名,并合并
colnames(ste6) = colnames(ann_all2)
ann_all3 = rbind(ann_all2,ste6)
head(ann_all3)
# 保存文件
write.table(ann_all3, "annot3.txt", append = F, quote = F, sep="t",
eol = "n", na = "NA", dec = ".", row.names = F, col.names = F) 总体环修饰
指标可以保存基本小修即可 title Metagenomic
title_font_size 12
total_plotted_degrees 350
start_rotation 270
branch_bracket_depth 0
branch_bracket_width 0
branch_color g
branch_thickness 0.5
ignore_branch_len 0
clade_marker_size 0.6
clade_marker_edge_width 0.35
class_legend_font_size 9
annotation_background_separation -0.4
annotation_background_offset -0.33
annotation_background_width 0.03
annotation_background_alpha 0.01
annotation_font_size 8
annotation_legend_font_size 9
ring_internal_separator_thickness 1 0.3
ring_internal_separator_thickness 2 0.3
ring_internal_separator_thickness 3 0.1
ring_internal_separator_thickness 4 0.1
ring_internal_separator_thickness 5 0.1
ring_internal_separator_thickness 6 0.1
ring_internal_separator_thickness 7 0.1
ring_internal_separator_thickness 8 0.1
ring_internal_separator_thickness 9 0.1
ring_internal_separator_thickness 10 0.1
ring_internal_separator_thickness 11 0.1
ring_internal_separator_thickness 12 0.1
ring_internal_separator_thickness 13 0.1
ring_internal_separator_thickness 14 0.1
ring_internal_separator_thickness 15 0.1
ring_internal_separator_thickness 16 0.1
ring_separator_color 1 y
ring_separator_color 2 y
ring_separator_color 3 b
ring_separator_color 4 b
ring_separator_color 5 b
ring_separator_color 6 b
ring_separator_color 7 b
ring_separator_color 8 b
ring_separator_color 9 b
ring_separator_color 10 b
ring_separator_color 11 b
ring_separator_color 12 b
ring_separator_color 13 r
ring_separator_color 14 b
ring_separator_color 15 r
ring_separator_color 16 r
ring_width 1 0.3
ring_height 1 0.5
ring_height 2 0.1
ring_height 3 0.1
ring_height 4 0.1
ring_height 5 0.1
ring_height 6 0.1
ring_height 7 0.1
ring_height 8 0.1
ring_height 9 0.1
ring_height 10 0.1
ring_height 11 0.1
ring_height 12 0.1
ring_height 13 0.1
ring_height 14 0.1
ring_height 15 0.1
ring_height 16 0.1 graphlan出图,三条命令三张图
注:如果没有graphlan,需要在linux服务器上,按官网或conda安装
推荐conda一条命令安装conda install graphlan
基于结点颜色、形状、背景色+透明度(annon2.txt)美化进化树(rep_set.tree)
# 进化树结合注释
graphlan_annotate.py --annot annot2.txt rep_set.tree 1.xml
# 绘制150dpi的PNG
graphlan.py --dpi 150 1.xml 1.png
# 绘制矢量图PDF
graphlan.py 1.xml 1.pdf
在上图的基础上,外圈添加丰度(anno3.txt),包括12个样品,各组中高/低,极是否显著
graphlan_annotate.py --annot annot3.txt 1.xml 2.xml
graphlan.py --dpi 150 2.xml 2.png
graphlan.py 2.xml 2.pdf
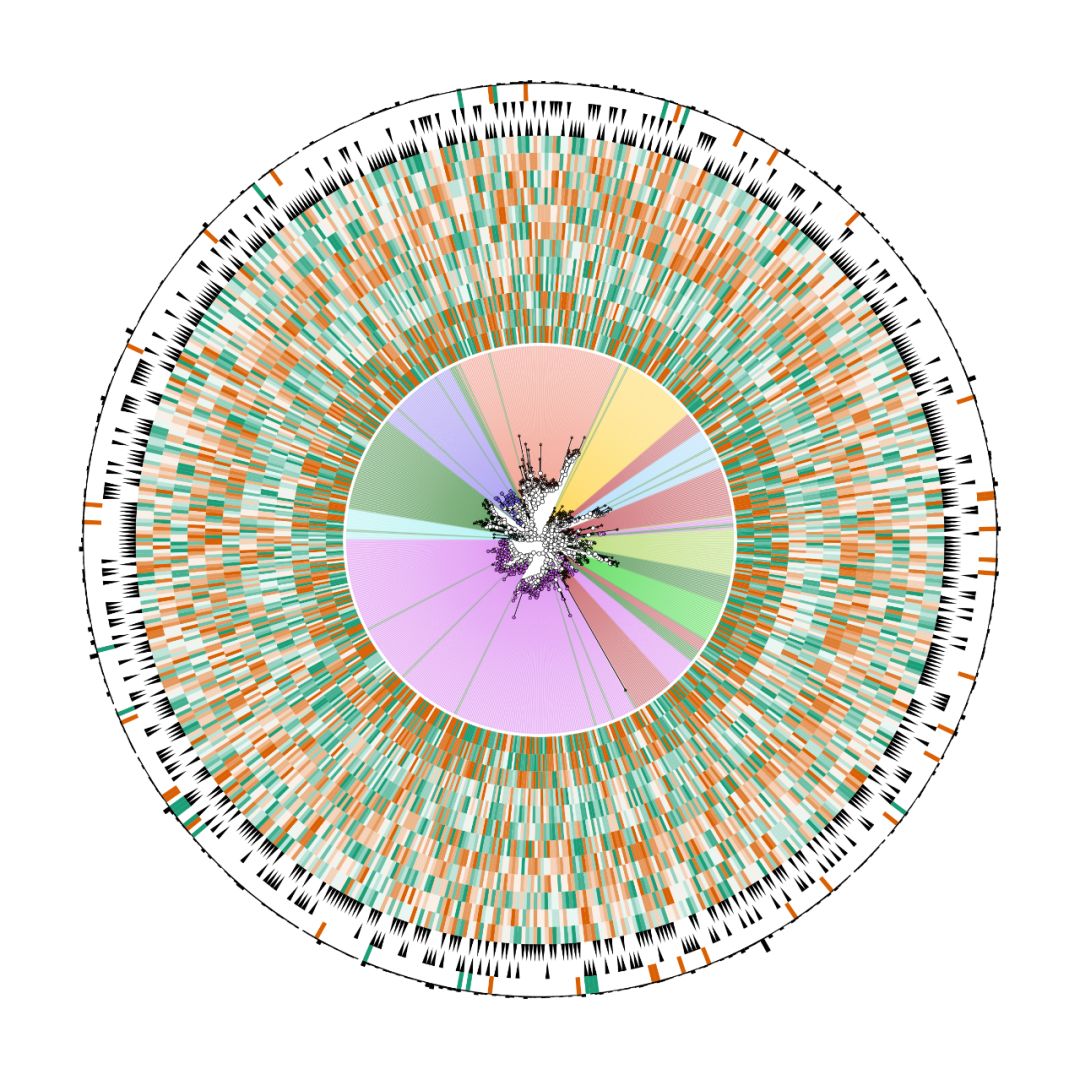
添加基本全局参数,调图形样式
graphlan_annotate.py --annot annot1.txt 2.xml 3.xml
graphlan.py --dpi 300 3.xml 3.png
graphlan.py 3.xml 3.pdf
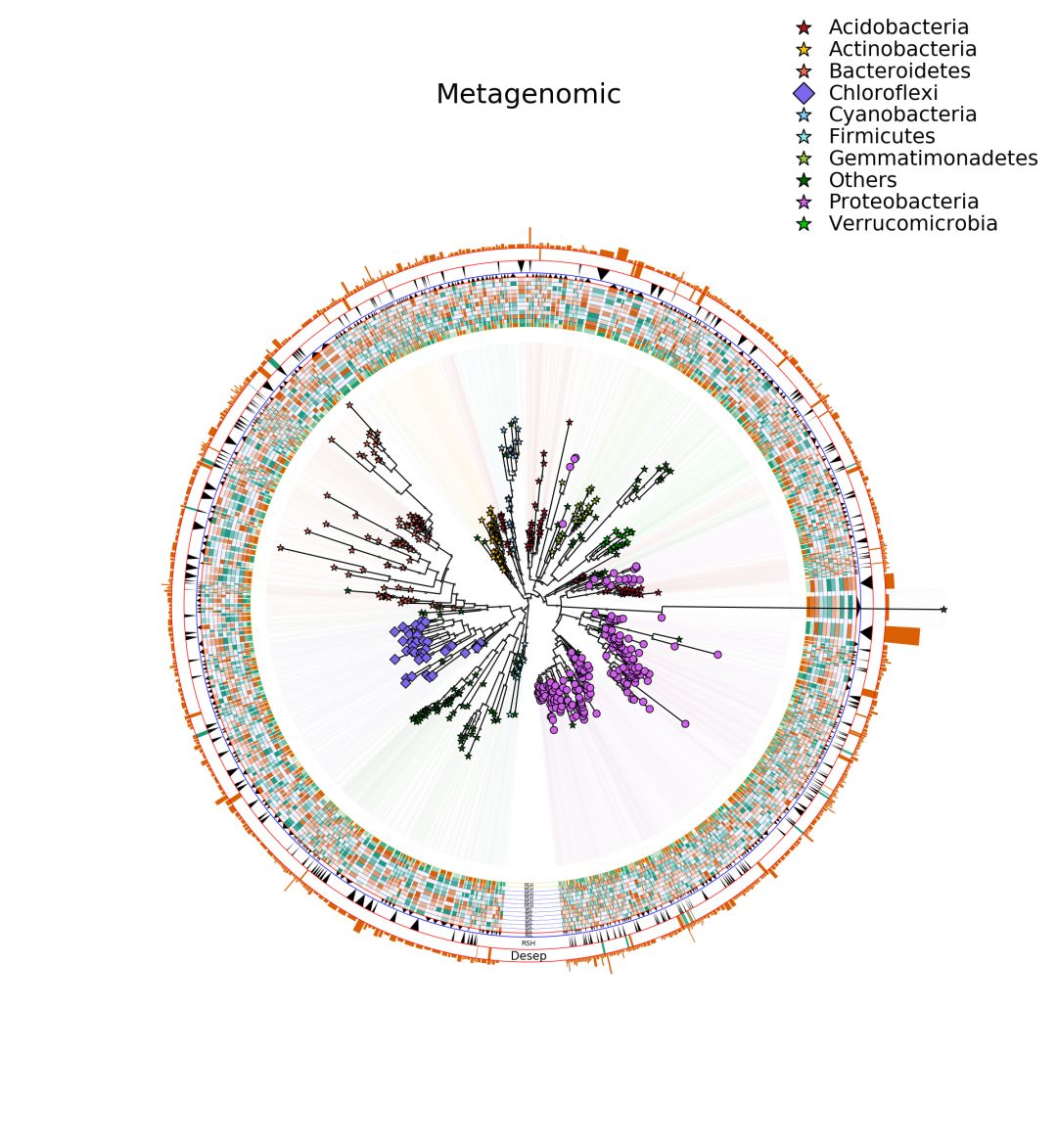
问题:分支节点未命名或无法命名
在扩增子分析中,我们经常构建的无根进化树,有没有根倒是无所谓,但是这些使用Fastree构建的进化树有共同的特征,就是除了叶节点,其余分支节点命名无法在graphlan指明。
进化树文件局部示例:
((((((((('OTU_512':0.01655,'OTU_592':0.05858)==1.000:0.15061==,((('OTU_409':0.05856,
但是这里我们可以看到分支节点名为1.000,但是在注释文件中通过这个节点无法添加相应的操作。
参考文献
Asnicar F, Weingart G, Tickle T L, et al. Compact graphical
representation of phylogenetic data and metadata with GraPhlAn[J].
PeerJ, 2015, 3: e1029. http://huttenhower.sph.harvard.edu/GraPhlAn
作者简介
文涛:2016年就读于南京农业大学。荣拜资源院沈其荣教授课题组,研究方向为根际微生物生态,具体为植物介导下根际小分子代谢组同土壤微生物群落在防控土传病害方面的相互作用。关注宏基因组和代谢组。大家有兴趣可以通过[email protected]交流。欢迎打扰!