OS论坛专栏
叮咚。“OS论坛专栏”上线了,我们将定期挑选OS论坛(网址:www.omicshare.com/forum)的优质内容分享给大家,如代码贴、实用工具分享、软件教程、文献解读等等,给论坛优质内容更多曝光机会,欢迎大家前往OS论坛投稿~,今天推荐论坛入驻网友“生信补给站”的一篇帖子(原帖链接:https://www.omicshare.com/forum/thread-6269-1-1.html),跟大家分享连锁不平衡图的绘制。
连锁不平衡图,用来可视化不同SNP之间的连锁程度,前同事间俗称“倒三角”图

。
本文使用自己的数据,因为安装R包后使用内置数据集运行出结果较容易,但是自己的数据就可能会有一些不大不小的“坑”,我替你们趟了。。。
一 、载入R包数据
数据为内置CEUData保存后,进行了“细微”的处理(去掉SNP碱基之间的“/”),因为这种基因型形式文件很常见;
library("LDheatmap")
#读入数据
SNP <- read.csv("CEUSNP.csv",header = TRUE)
pos <- read.csv("CEUDist.csv",header= TRUE)
#查看数据
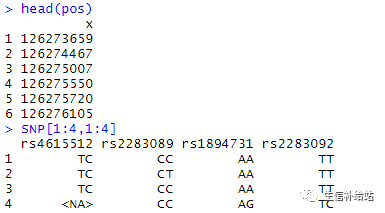
head(pos)
SNP[1:4,1:4]
二、 绘制连锁不平衡图
2.1 直接绘制SNPpos <- pos$x
LDheatmap(SNP, SNPpos,color = grey.colors(20))
Error in LDheatmap(SNP, SNPpos, color = grey.colors(20)) :
column 1 is not a genotype object
额, 也许是因为没有“/”的原因,加上试试?
2.2 碱基型之间加“/“
怎么加呢?首先想到 Tidyverse|数据列的分分合合,一分多,多合一 的 separate 和 unite ,可是没有分隔符。。
经高人指点 ,使用替换的方式,解决方法很多。此处使用R-do包的函数
library(do)
df <- na.omit(SNP)
#A,C,G ,T 替换为A/,C/,G/,T/
df1 = do::Replace(df,pattern = c("A:A/","C:C/","G:G/","T:T/"))
#去掉最后的/
SNPdata <- do::Trim(df1,"/")
SNPdata[1:4,1:4]
rs4615512 rs2283089 rs1894731 rs2283092
1 T/C C/C A/A T/T
2 T/C C/T A/A T/T
3 T/C C/C A/A T/T
5 T/C C/C A/A T/T
加上了,再次绘图
LDheatmap(SNPdata, SNPpos,color = grey.colors(20))
Error in LDheatmap(SNPdata, SNPpos, color = grey.colors(20)) :
column 1 is not a genotype object
额 ,还是不行,同样的报错。检索报错,尝试转换数据格式。
2.3 碱基型转为genotype object
使用genetics包的函数转化
library("genetics")
for(i in 1:ncol(SNPdata)){
SNPdata[,i]<-as.genotype(SNPdata[,i])
}
LDheatmap(SNPdata, SNPpos,color = grey.colors(20))

额 ,,,终于可以了。。。
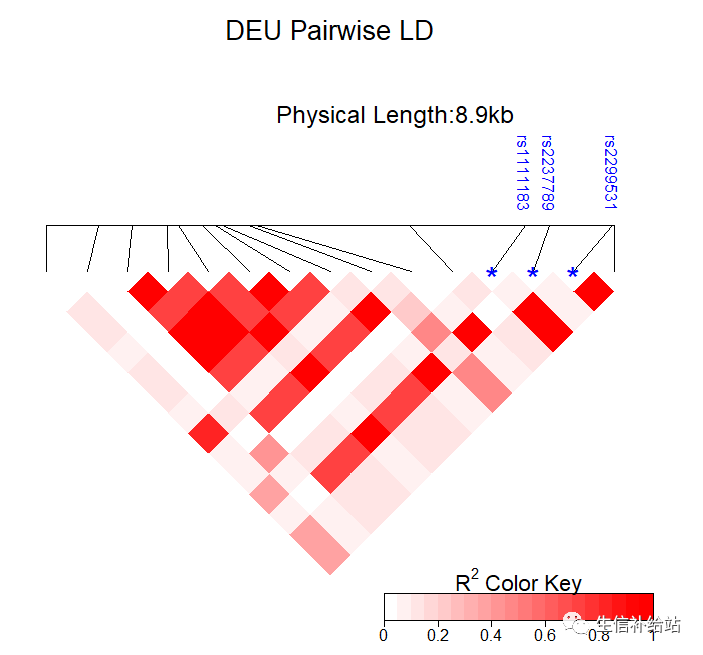
三 图形调整,优化3.1 调整颜色,更改标题,标示SNP名称color.rgb <- colorRampPalette(rev(c("white","red")),space="rgb")
## 绘制连锁不平衡图
names <- c("rs1111183", "rs2237789", "rs2299531")
LDheatmap(SNPdata, SNPpos,
color=color.rgb(20),
title = "DEU Pairwise LD",
SNP.name=names,flip=TRUE)
3.2 使用grid调整SNP标记名称字体大小、颜色
library(grid)
grid.edit(gPath("ldheatmap", "geneMap","SNPnames"),
gp = gpar(col="black",lwd = 1,cex=0.7))
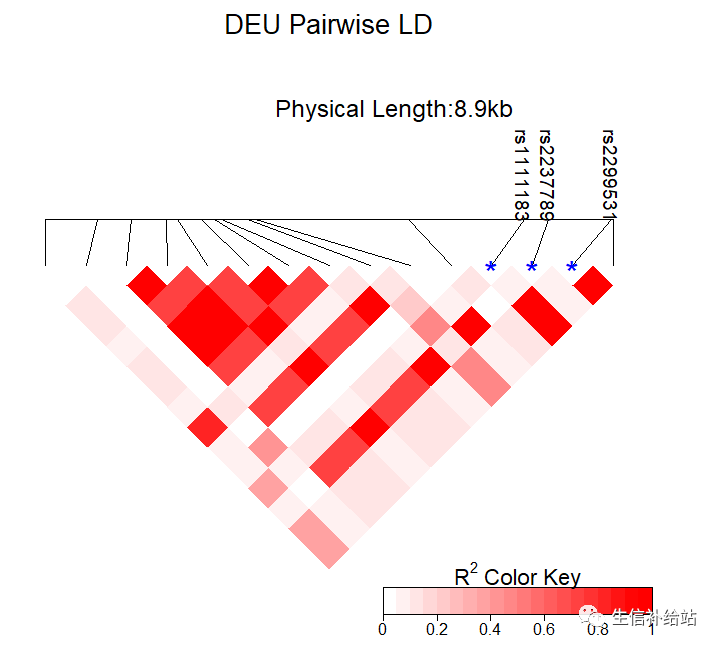
所谓的”倒三角图“完成,haploview软件也很好看,且有block,批量也许不太友好,见仁见智了!
转自:基迪奥生物