测序行业的蓬勃发展,带来微生物组学日新月异的变化。目前,单一组学的文章不断“贬值”,前沿研究的目光从单一组学逐步拓展至多组学对贯穿分析,即结合多个组学的分析角度,从多个层面阐述生物学机制。
微生物多组学贯穿分析策略十分丰富:如常见的16s与宏基因组贯穿分析,可以验证物种的特征、丰富功能的探究;而16s与代谢组的贯穿分析思路同样常见于高分文章中,通过16s探究不同处理/环境下菌群的物种组成变化,结合代谢组对应的代谢物的变化,进而找到不同处理/环境下引发细菌丰度差异最终导致代谢表型差异的机制。参考阅读《选好思路和方法,给自己一篇多组学高分文章 》
在16s与代谢组贯穿分析中,相关性热图是一个重要的分析手段,主要用于逐一呈现细菌物种与代谢物间的相关性高低,是筛选潜在关联的物种与代谢物的主要途径,对于下游的实验起到指导意义。此类相关性热图在高分文章中频繁出现,足见其重要性(图1、图2)。
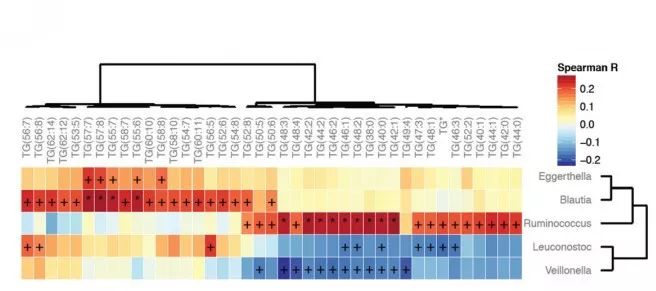
图1 物种代谢物热图(2015,Cell Host& Microbe,IF=15.753 )[1]

图2 物种代谢物热图(2018,NatureMedicine,IF=30.641)[2]
那么,该如何画出此类高分文章中的相关性热图呢?这里,以16s与代谢组的数据为例,向大家分享如何使用R语言进行两个组学数据的相关性计算、绘制相关性热图。
1.加载R包
#psych包用于计算相关性、p值等信息
library(psych)
#pheatmap包用于绘制相关性热图
library(pheatmap)
#reshape2包用于输出数据的整合处理
library(reshape2)
2.读入数据
#读取微生物丰度信息表
#表头需带有分类水平、物种名称等关键信息
#第一列为样本名称信息
phy <-read.table(file = "phy.xls", sep = "t", header = T,row.names= 1)
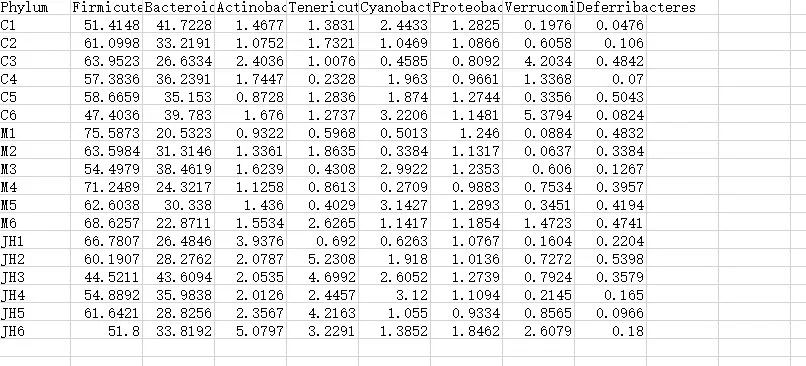
图3 微生物丰度信息表格
#读取代谢物信息表
met <-read.table(file = "met.xls", sep = "t", header = T,row.names= 1)
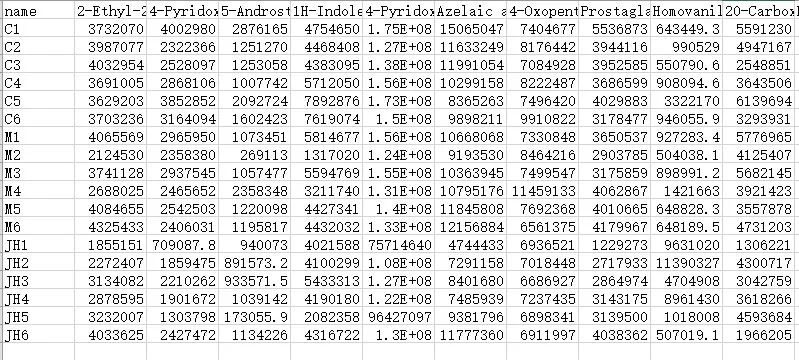
图4 代谢物丰度信息表格
3.计算相关性、p值
#计算相关性矩阵(可选:”pearson”、”spearman”、”kendall”相关系数)、p值矩阵
cor <-corr.test(phy, met, method = "pearson",adjust= "none")
#提取相关性、p值
cmt <-cor$r
pmt <- cor$p
head(cmt)
head(pmt)
4.数据保存
#输出相关系数表格,第一行为代谢物信息,第一列为物种信息
cmt.out<-cbind(rownames(cmt),cmt)
write.table(cmt.out,file= "cor.txt",sep= "t",row.names=F)
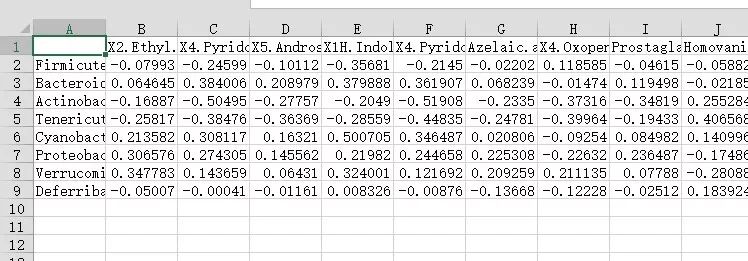
图5 相关性系数表格
#输出p值表格,第一行为代谢物信息,第一列为物种信息
pmt.out<-cbind(rownames(pmt),pmt)
write.table(pmt.out,file= "pvalue.txt",sep= "t",row.names=F)
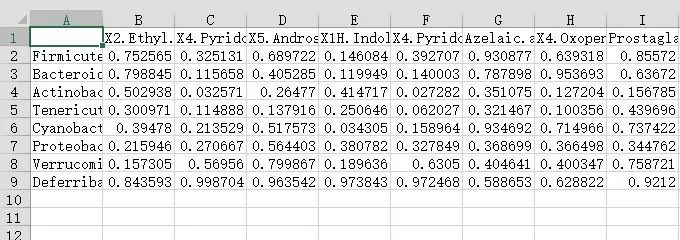
图6 p值表格
#以关系对的形式输出表格
#第一列为物种名,第二列为代谢物名,第三、第四列对应显示相关系数与p值
df <-melt(cmt,value.name= "cor")
df$pvalue <- as.vector(pmt)
head(df)
write.table(df,file= "cor-p.txt",sep= "t")

图7 关系对信息
5.绘制显著性标记
#对所有p值进行判断,p< 0.01的以“**”标注,p值 0.01<p< 0.05的以“*”标注
if(!is.null(pmt)){
ssmt <- pmt< 0.01
pmt[ssmt] <- '**'
smt <- pmt > 0.01& pmt < 0.05
pmt[smt] <- '*'
pmt[!ssmt&!smt]<- ''
} else{
pmt <- F
}
6.绘制相关性热图
#自定义颜色范围
mycol<-colorRampPalette(c("blue","white","tomato"))(800)
#绘制热图,可根据个人需求调整对应参数
#scale=”none” 不对数据进行均一化处理 可选 "row", "column"对行、列数据进行均一化
#cluster_row/col=T 对行或列数据进行聚类处理,可选F为不聚类
#border=NA 各自边框是否显示、颜色,可选“white”等增加边框颜色
#number_color=”white” 格子填入的显著性标记颜色
#cellwidth/height=12 格子宽度、高度信息
pheatmap(cmt,scale = "none",cluster_row = T, cluster_col = T, border=NA,
display_numbers = pmt,fontsize_number = 12, number_color = "white",
cellwidth = 20, cellheight =20,color=mycol)
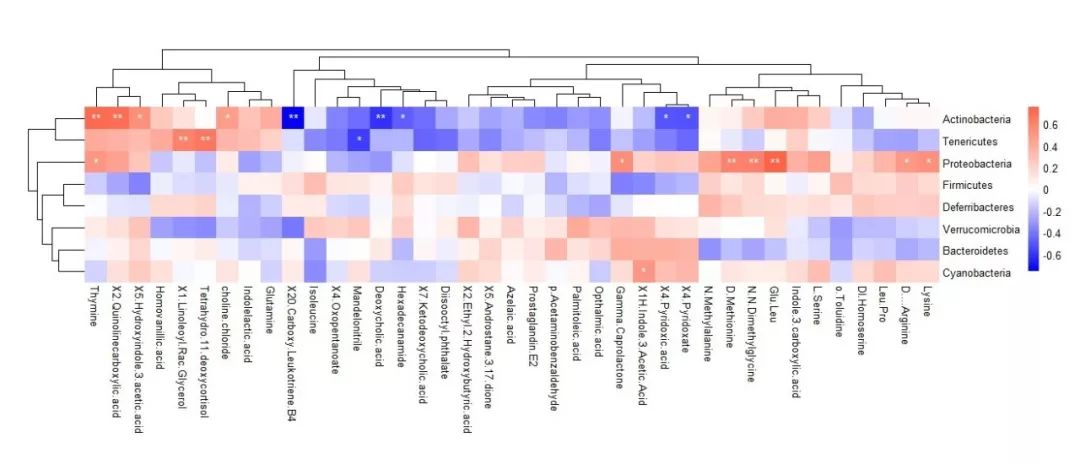
图8 R语言绘制的物种+代谢物相关性热图
#图片保存,代码中输入”filename=”,或在R语言软件中点击“文件-另存为” 进行保存
pheatmap(cmt,scale = "none",cluster_row = T, cluster_col = T, border=NA,
display_numbers = pmt, fontsize_number = 12, number_color = "white",
cellwidth = 20, cellheight = 20,color=mycol,filename= "heatmap.pdf")
参考文献
[1]Kostic AD, Gevers D, Siljander H,
et al. The dynamics ofthe human infant gut microbiome in development and
in progression toward type 1diabetes. Cell Host Microbe.
2015;17(2):260–273.doi:10.1016/j.chom.2015.01.001
[2]Hoyles, Lesleyet al. “Molecular
phenomics and metagenomics of hepatic steatosis innon-diabetic obese
women.” Nature medicine vol. 24,7 (2018):1070-1080.
doi:10.1038/s41591-018-0061-3